はじめに
ImageJ/Fijiにはしばらくお世話になっていたのですが、scipyやscikit-image、機械学習関連などといったPythonの豊富なライブラリが利用できず、何か実装したかったらJavaで書くしかありません。これは、実験者と開発者の分断を助長する、あまりにもよろしくない事態です。加えて、ImageJとExcelを行き来せざるを得なかったり、ウィンドウが増えすぎたりと、使っていてかなり不便だと感じる場面が少なくないと思います。
この現状を打破するべく調査していて行きついたのがnapariという素晴らしいPythonのライブラリでした。これがあれば、すべての解析をPythonで統一できます。ぜひ知ってもらいたいので、複数回に分けてまじめに紹介していこうと思います。
目次
napariとは
Pythonで画像を表示する際、Matplotlibでもいいのですが、拡大縮小したり、スライダーを動かして動画として見たり、立体的にぐるぐるしたりしたいですよね。napariは
napari is a fast, interactive, multi-dimensional image viewer for Python
と謳っている通り、快適に多次元画像を可視化することができます。詳しくは
をお読みください。ここでは重要なところに絞って使い方を説明していきます。
※ Julia言語からでも使えます。GitHubのこちらに上がっています。JuliaImagesを使い慣れているJulianの方は参考までに。
※ scikit-imageもviewerというサブモジュールを有していますが、バージョン0.20で消されるので他のを使ってねと言っており、その中でnapariが紹介されています(こちら)。
![]() この記事はnapariのバージョン0.4.19現在に関するものです。ホットに開発が進んでいるので、これから新機能の追加や変更があるかと思います。
この記事はnapariのバージョン0.4.19現在に関するものです。ホットに開発が進んでいるので、これから新機能の追加や変更があるかと思います。
napariでできること
ImageJとの比較みたいになりますが、主に次のような特徴が挙げられます。
1. 1つのウィンドウ内で、画像やラベル、長方形などのオブジェクトをレイヤーとして複数重ねることができる。
これでウィンドウが増えすぎることが防げるうえ、複数の画像を重ねながら比較する際に毎回"Merge Channel"とかやらずに済むので非常に便利です。
2. キーボードショートカット、マウスのクリック時・ドラッグ時の機能を非常に簡単に登録でき、レイヤーごとに設定することもできる。
例えば、画像の特定領域を切り出すのはGUI頼みである一方で、scipy.optimizeでフィッティングするといった高度なカスタマイズが必須となる解析はGUIメインのソフトウェアは非常に苦手です。napariでは、フィッティング関数を定義し、キーボードショートカットとして登録しておけば解決します。ソフトを使い分ける時代はもう終わりで、これからは必要に応じて素早くUIを作り変える時代です。
3. 手動で選択したレイヤーやオブジェクトの情報をコンソールから対話的に取得できる。
2.とも関連しますが、Python上での画像解析の弱点は、人間が目で見て判断する操作は自動化できないという点です。細胞の領域を取り出してくる、結晶の周期構造がx軸に平衡になるように回転させる、といった操作くらいは人間がやることだと思います。これを毎回matplotlibでプロットしてはおおよその値を入力するのでは効率が悪いですが、napariを通して行えばずっとスムーズになります。
4. なんならImageJを呼び出せる。
最近Nature methodsでも公開されたプラグインnapari-imagejで両ソフトウェア間でデータをやりとりできます。
インストール
pip install napari # pipを用いる場合
conda install napari -c conda-forge # condaを用いる場合
PyQtなどが最新のもの (>=5.12) になっていないと思うように動作しないかもしれないので、最新のものにしておきましょう。
pip install pyqt5 # pipを用いる場合
conda install pyqt -c conda-forge # condaを用いる場合
基本操作
Viewerを立ち上げて画像を表示
モジュールをインポートします。
import napari
import numpy as np
import skimage
napari.Viewerクラスが、napariのウィンドウ本体になります。ウィンドウ内にあるものはすべてここからアクセスします。次のようにインスタンスviewerを作成します。
viewer = napari.Viewer()
napari.run() # Jupyterであれば、これは不要
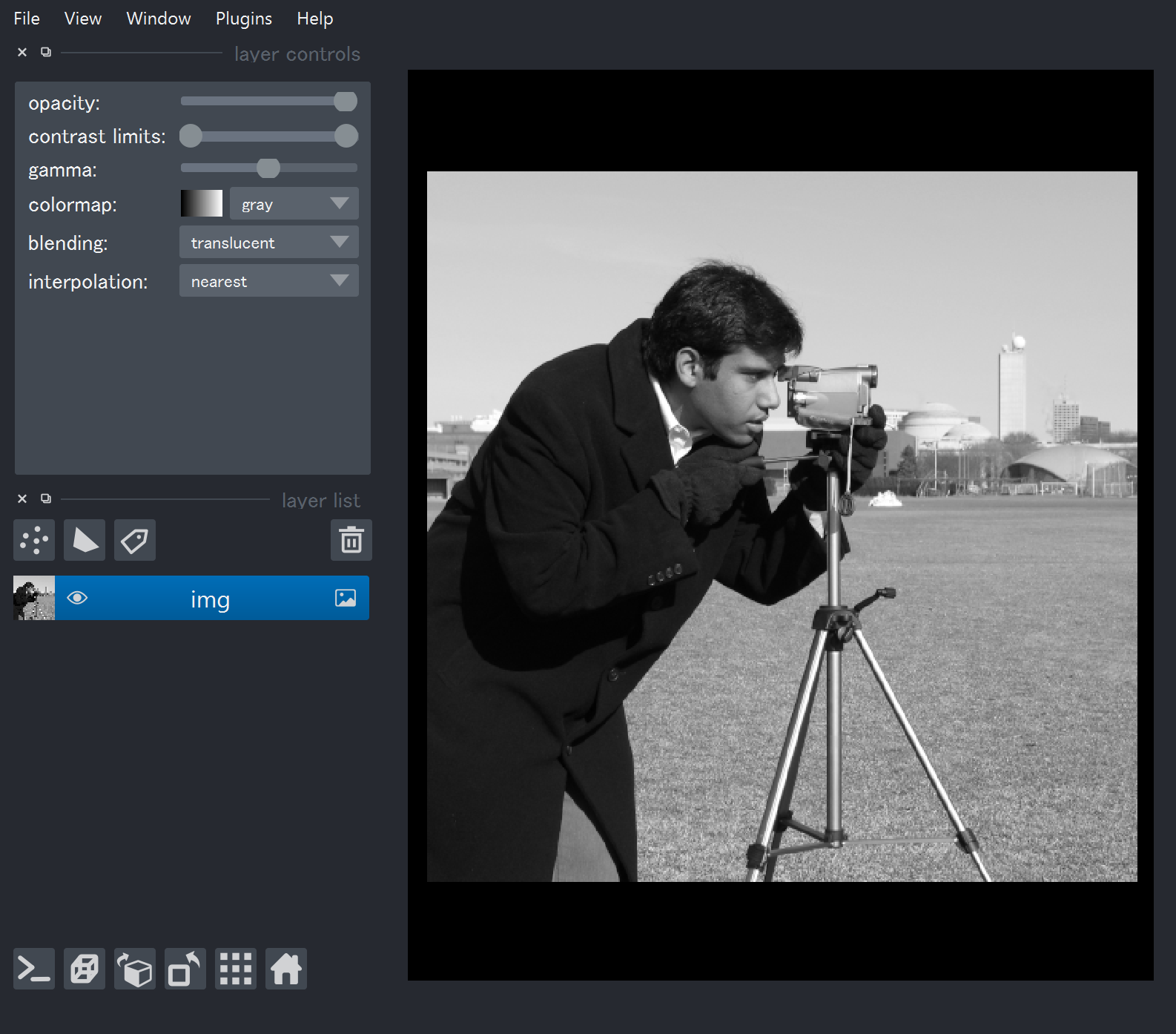
インスタンスが作成された時点で、図のようにnapariのウィンドウが開きます。
(初期設定によって多少レイアウトが変わる可能性あり)
続いて画像を追加します。napari.Viewerのメソッドadd_imageを使います。
img = skimage.data.camera() # 画像を取得
viewer.add_image(img)
[Out] <Image layer 'img' at 0x...>
これでウィンドウに画像が表示されました!ドラッグでカメラの平行移動、スクロールで拡大縮小できます。
ちなみに、ウィンドウを立ち上げるだけのことはほとんどないので、同時に画像を送る関数view_imageも用意されています。
viewer = napari.view_image(img) # 新しいウィンドウを立ち上げてそこに画像を送る
ウィンドウ上での簡単な操作
napariはレイヤーを追加していく形をとります。次のGIFのように、"layer list"ウィジェット内にあるボタンから順にPointsレイヤー、Shapesレイヤー、Labelsレイヤーを追加できます。
各レイヤーは選択中に、左上の"layer control"ウィジェットから固有の操作ができます。例えば、
-
Points... 散布図を手動で描くイメージ- 新しい点の追加/削除(3次元的にもできる!)
- 点の移動
- 点の形の変更
-
Shapes... PowerPointの図形のイメージ- 長方形、折れ線、楕円などのオブジェクトを追加/削除
- オブジェクトの選択、拡大/縮小、回転
- オブジェクト頂点の追加/削除
-
Labels... ペイントのイメージ- 色を塗ったり消したりする(異なる色は異なる自然数のラベルに対応する)
- 塗りつぶし
- カラーピック
- 3次元描画(!)
などができます。ショートカットの整備が不十分なのと、一部Ctrl+Zが利かないのがこれからの課題ですが、欲しい機能はだいたい揃っている感じがしますね。
他にも、上部のメニューバーから座標軸やスケールバーを表示したり、スクリーンショットと撮ったりできます。
画像の3D表示
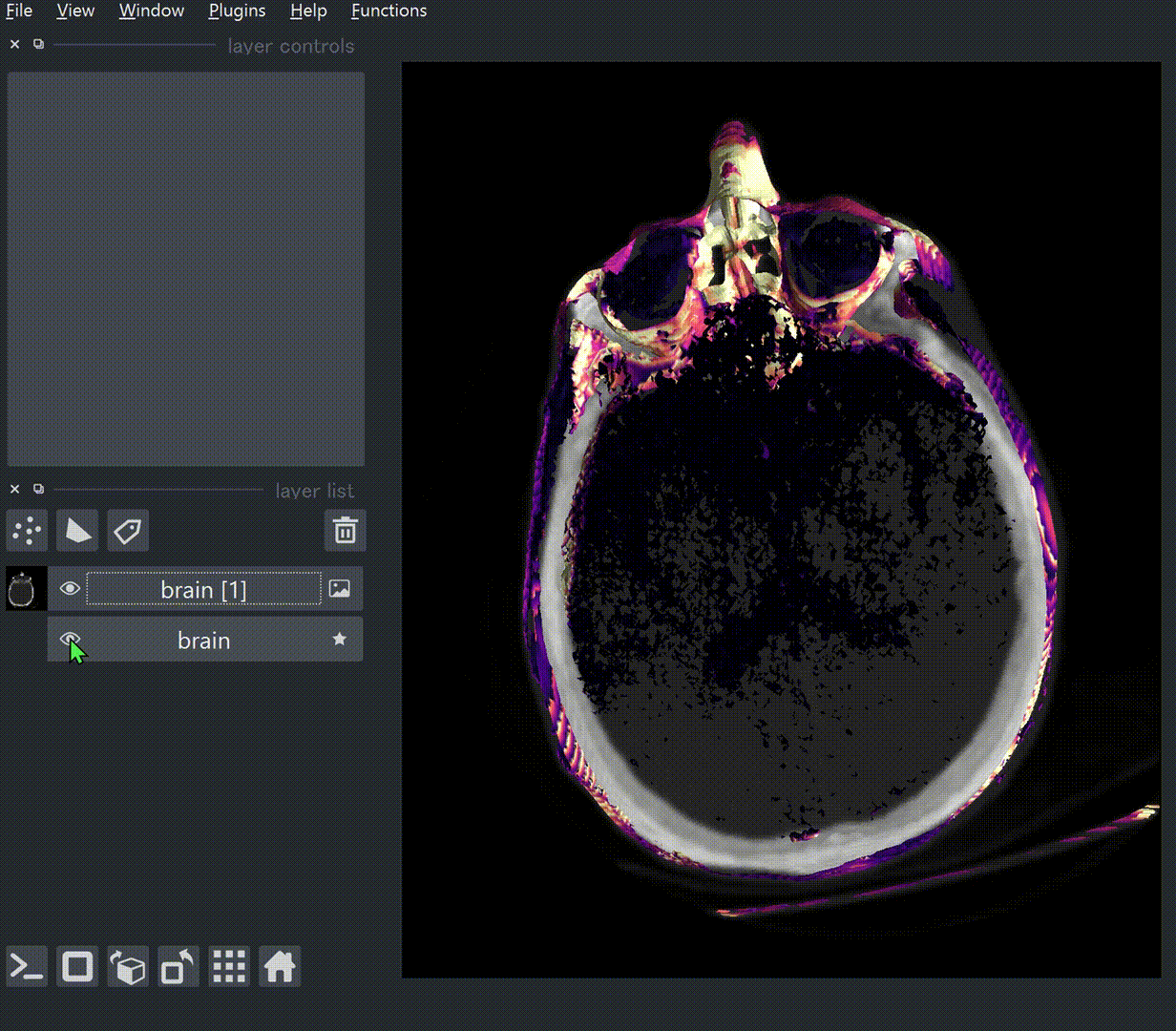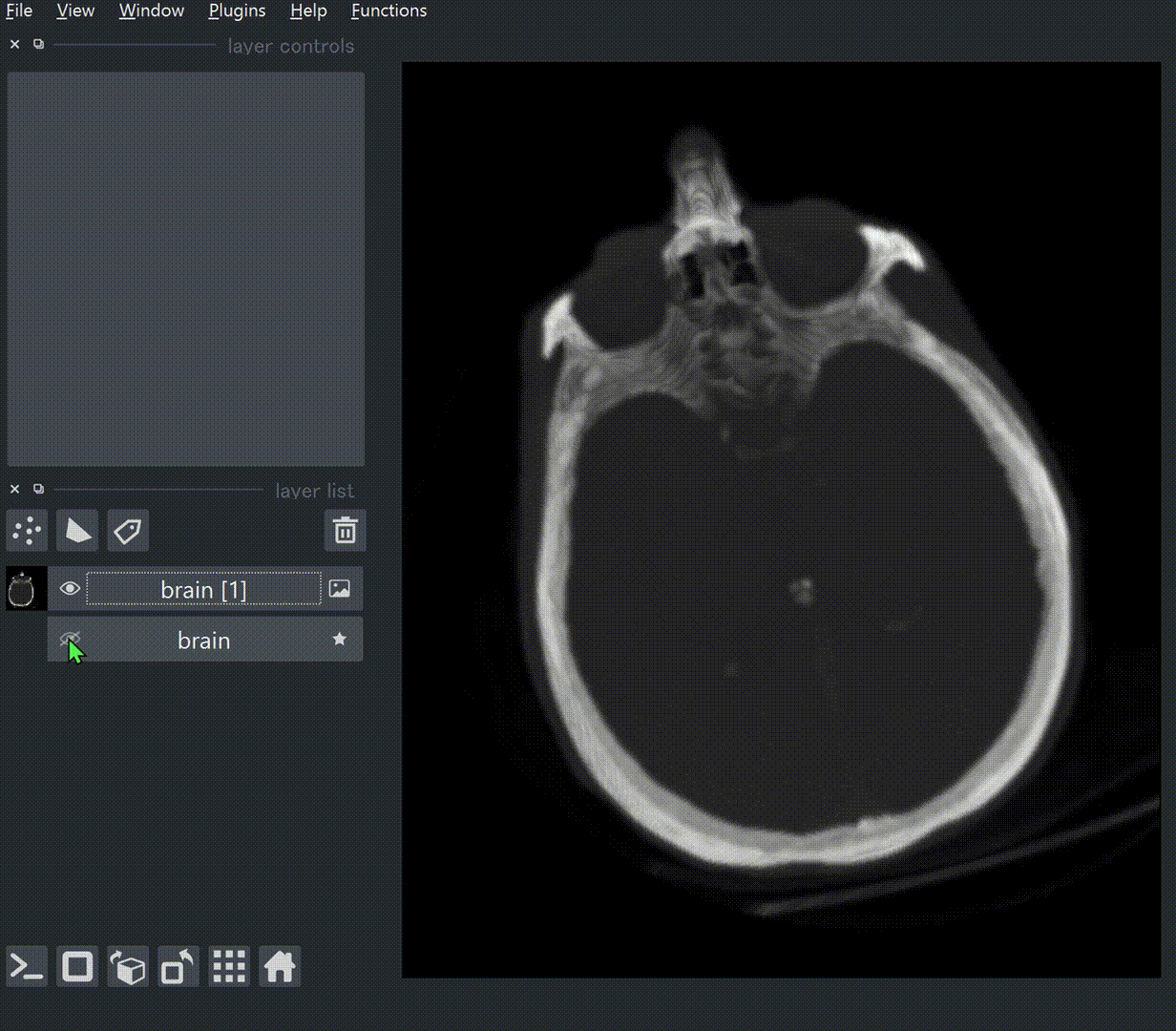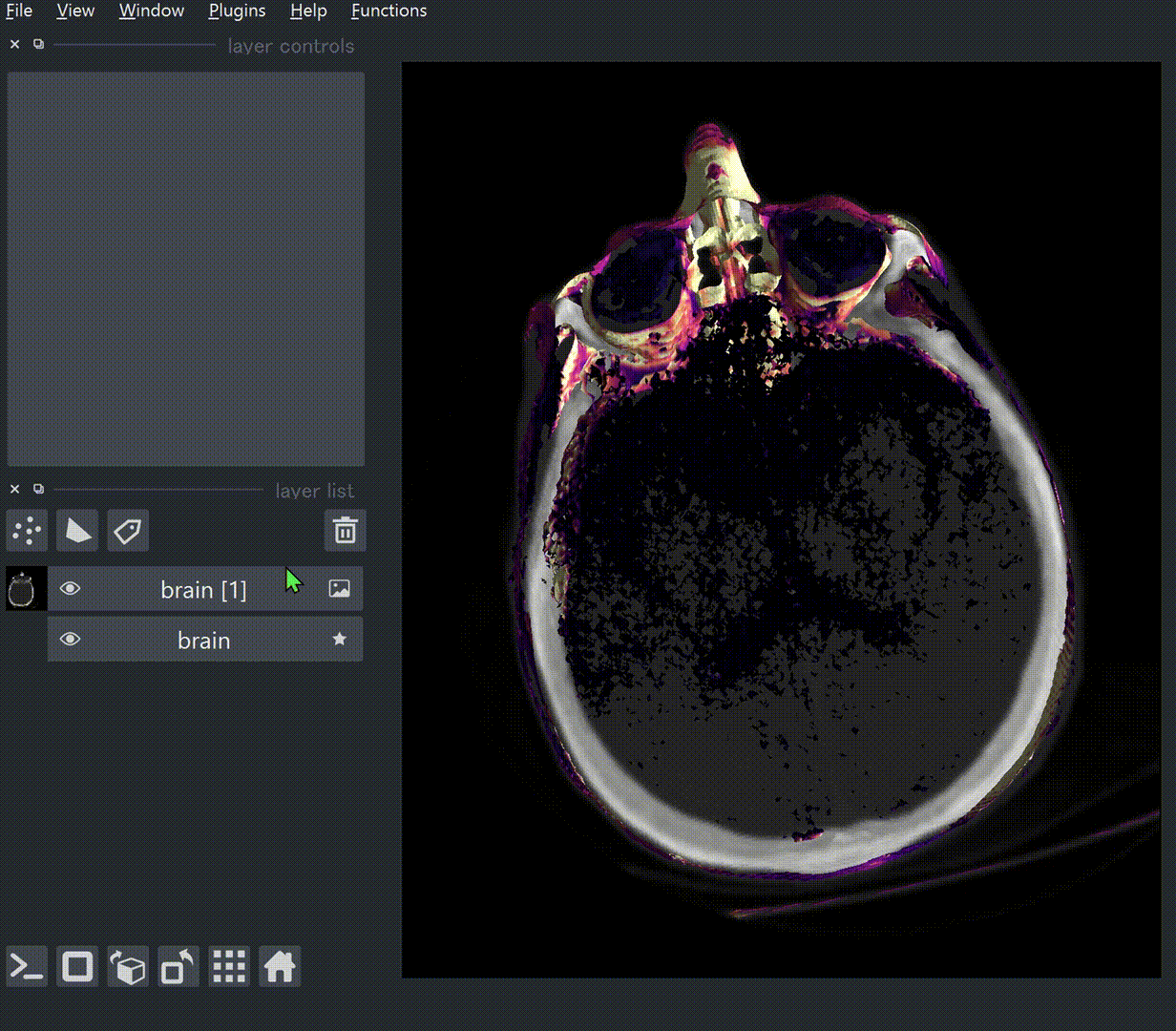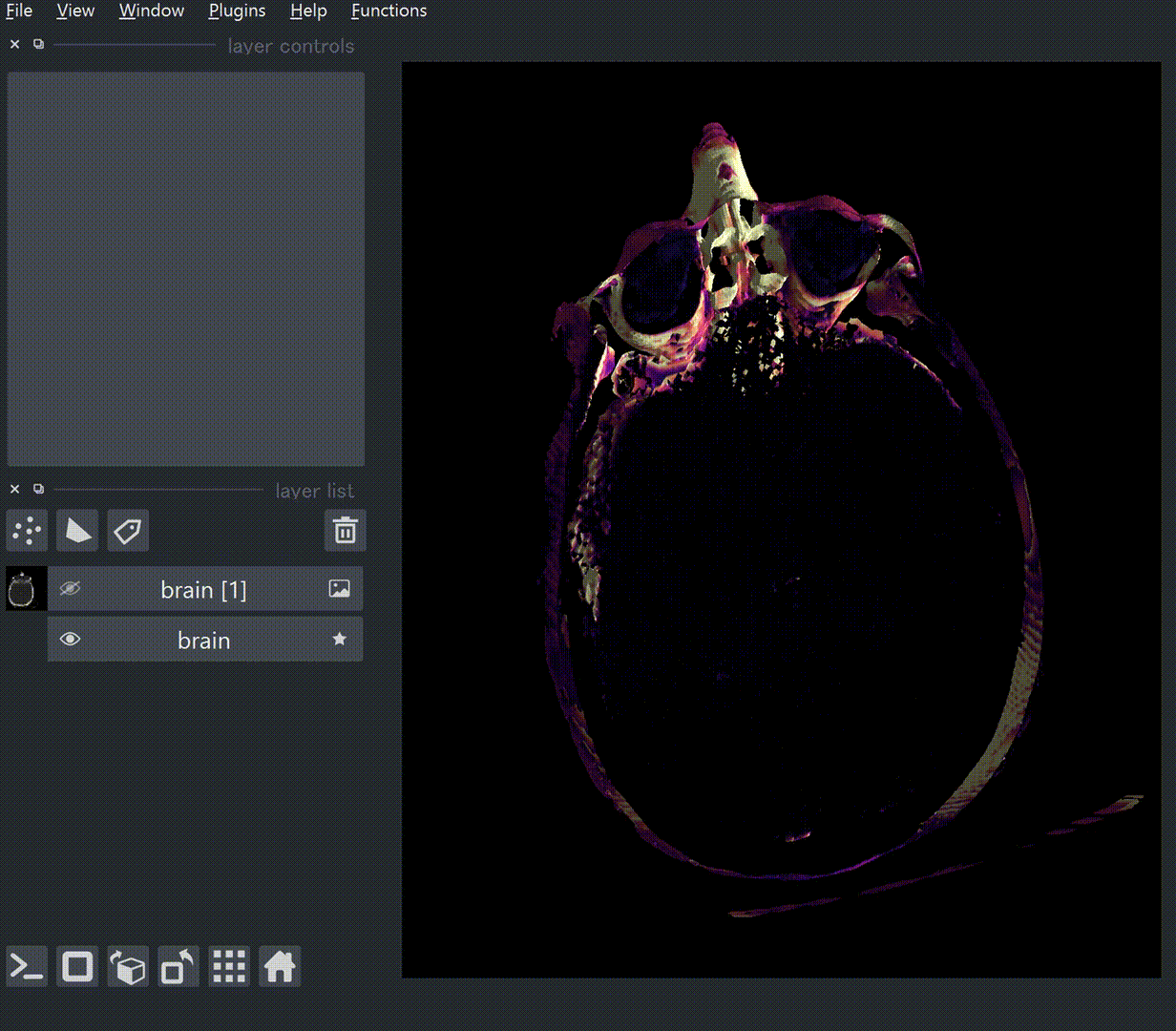
napariが得意とする3D表示をやってみます。今回はメニューバーFile > Open Sample > napari builtins > Binary Blobs (3D)とすることでサンプル画像を取得します。scikit-imageのサンプル画像を全部ダウンロードしている場合は"Brain (3D)"や"Cells (3D+2Ch)"など、もっとマシな画像が使えるかと思います。
デフォルトでは2D表示なので、下部のスライダーを動かして断面をみる形になります。3D関係の操作は"layer list"の下のボタンから行います。
3Dに切り替えると、"Binary Blobs (3D)"だとほぼ真っ白になってしまいます(バイナリ画像なので)。これは、レンダリングが"mip (max intensity projection)"になっているためで、バイナリ画像の場合"iso (iso-surface)"に設定すると見やすくなります。ぐるぐる回しましょう。
プラグインの導入
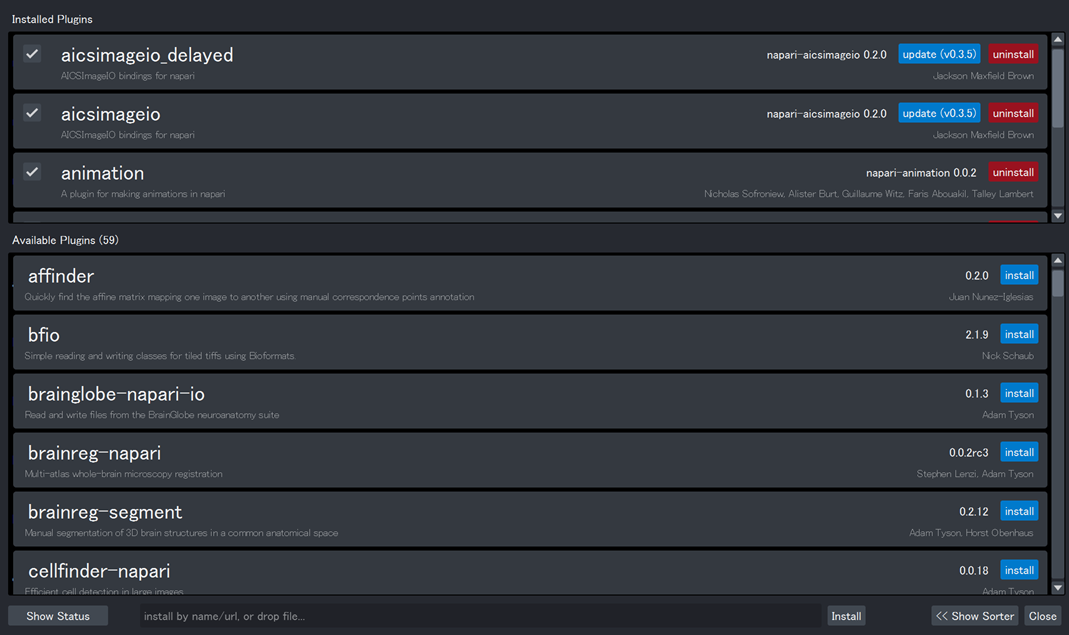
napariは機能追加のしやすさが売りなので、当然プラグインシステムが既に確立しています。上部のメニューバーのPlugins >> Install/Uninstall Package(s)...を選択すると、次の図に示すように新しいウィンドウに現在手に入るプラグインや更新可能なプラグインなどが表示され、そのまま手動でpipインストールできます。
簡単にアニメーションを作れるnapari-animationは特におすすめです。
スクリプトからのレイヤーの追加
続いては、スクリプトからレイヤーを追加していく方法について詳しく説明します。すでに述べたようにadd_imageを用いることでとりあえず画像をnapariに送ることができますが、実際には異なる色でマージした状態で送ったり、画像ではなく点や長方形を追加したりしたいこともあります。napariには多彩なレイヤーが用意されているので、状況に応じて使い分けられるとよいですね。
より詳細な情報はこちらを参照。
0. 各レイヤー共通のパラメータ
レイヤーはすべてLayerクラスを継承しており、レイヤーの種類にかかわらず、共通したプロパティがあります。これらはキーワード引数も共通しているので、初めにまとめてしまいます。
詳細を表示
name=None, metadata=None, scale=None, translate=None, rotate=None, shear=None,
affine=None, opacity=1, blending='translucent', visible=True, multiscale=False
- name str ... レイヤーの名前。他と被る場合は"XXX [1]"のように後ろに番号がつく。
- metadata dict ... レイヤーのメタデータ。補足・メモ用。
-
scale tuple of float ... 各軸のスケール。例えば
scale=(0.5, 0.2)なら、y軸で50%、x軸で20%のスケールがかかり、縦長のピクセルになる(行/列はy/xの順に対応することに注意!)。 -
translate tuple of float ... 各軸の平行移動。例えば
translate=(3,5)なら、下に3ピクセル、右に5ピクセル移動して画像が表示される。 - rotate float, tuple of float ... 反時計回り、$(0, 0)$のピクセルを中心とした回転。ラジアンではなく度数。tuple of floatで与えられた場合は3次元回転に対応する。
-
shear 1-D array ... せん断(正方形をひし形につぶすような変換)。2D画像の場合、
shear=[a]とするとx軸が$y=ax$方向に向いたように画像がつぶれる。 -
affine 2-D array ... アフィン変換行列。
scale,translate,rotate,shearはアフィン変換行列を分かりやすく分解したものなので、それらを指定するのとaffineを指定するのは数学的には同義となる。 - opacity float ... 非透明度、もしくはアルファチャネル。0で完全に透明、1で不透明になる。
- blending str ... 他のレイヤーとどのようにブレンドするか。opaque (不透明)、translucent (半透明)、additive (加算的) のいずれか。要するに複数のレイヤーが重なるときに、奥側のレイヤーを隠すかどうかを指定する。しかしなぜかtranslucentで半透明にならない。
- visible bool ... レイヤーを見える状態で追加するか否か。
-
multiscale bool ... マルチスケールで画像を表示するか否か (Google Mapで拡大倍率によって表示が不連続に変わるのを想像するとわかりやすい)。非常に大きな画像では拡大縮小の倍率変化が大きくなる。このとき、細部を見るためにズームインしているときは1ピクセル単位で表示する必要があるが、全体を見るためにズームアウトしているときは画像の表示のために全ピクセルを計算させる必要はない。このとき、
add_image([img, img[::4, ::4]], multiscale=True)のように与えれば、ズームアウトしていくと途中で勝手に4ピクセルごとの表示に切り替わる。
なお、レイヤーの有効な利用例がGitHubリポジトリのこちらにまとまっています。
1. Image layer
viewer.add_image(...)で配列を画像として追加します。
詳細を表示
add_image(data=None, *, channel_axis=None, rgb=None, colormap=None,
contrast_limits=None, gamma=1, interpolation2d='nearest', interpolation3d='linear', rendering='mip',
iso_threshold=0.5, attenuation=0.05, name=None, metadata=None, scale=None,
translate=None, rotate=None, shear=None, affine=None, opacity=1, blending=None,
visible=True, multiscale=None)
-
data array ... 画像データ。普通なら
numpy.ndarrayだが、メモリに乗らない大きな画像をdaskで渡してもちゃんと表示される。daskの使い方に関しては以下を参照。 -
channel_axis int ... チャネル軸。この軸に沿って画像が分割され、異なる色、異なるレイヤーで画像が追加される。例えばTIFFファイルではchannel, y, xの順に軸が並んでいるので、
channel_axis=0と指定することでチャネルごとに色付けされる。blendingも指定しなければ"additive"に切り替わる。 - rgb bool ... RGB画像として追加するか。この場合は軸はPNG画像などの順番であるy, x, colorに従う必要がある。
-
colormap str ... カラーマップ、もしくはLUT (look up table)。画像のピクセル値と色を対応させるもので、
napariが内部で使っているvispyで用意されているものを使うのが手っ取り早い。"gray", "plasma"など、matplotlibのカラーマップの代表的なものが用意されていると考えるとよい。 -
contrast_limits list ... 画像のコントラストの最小/最大。蛍光顕微鏡画像などでは外れ値があって周りが暗くなるので、
np.percentileなどで外れ値を一部サチュレートさせたコントラストを使うことになる。 - gamma float ... ガンマ値。ピクセル値とカラーマップを非線形に対応させるときに用いる。
- interpolation2d/interpolation3d str ... 2D/3Dモードでのピクセル間の補間の方法。画像をズームインし、スクリーンの画素よりも画像の1ピクセルが大きくなった時にどのように画像を表示するかを決める。例えば"nearest"はピクセルの正方形がそのまま拡大され、"bilinear"や"bicubic"ではなめらかに接続される。
- rendering str ... 3D表示のときのレンダリング方法。
-
iso_threshold float ...
rendering="iso"のときのパラメータ。 -
attenuation float ...
rendering="attenuated_mip"のときのパラメータ。
2. Labels layer
viewer.add_labels(...)で整数配列をラベルとして追加します。異なる整数は使える色を使いきるまで異なる色で表示され、0は背景となり透明になります。
詳細を表示
add_labels(data, *, colormap=None, features=None, name=None,
metadata=None, scale=None, translate=None, rotate=None, shear=None, affine=None,
opacity=0.7, blending='translucent', visible=True, multiscale=None)
-
data array ... ラベルデータ。
scipy.ndimage.labelなどの出力をそのまま用いるのが基本となる。
例えば2D画像imgに対し
from scipy import ndimage as ndi
binary_input = img>100
structure = [[0,1,0],[1,1,1],[0,1,0]]
labels, n_labels = ndi.label(binary_input, structure)
で得られる配列labelsを渡すことで可視化できる。
-
colormap ...
from napari.utils.colormapsのクラス・関数を用いて作成する。 -
features dict or DataFrame ... (背景を含む)各ラベルに与える何かしらの量。ラベルの上にマウスを合わせると表示されるので、画像と対応させながら参照したい情報を載せるとよい。
skimage.measure.regionprops_tableの結果と互換性があるが、こちらはデフォルトでは背景を含まないので注意 (→ scikit-imageの公式ドキュメント)。
3. Points layer
viewer.add_points(...)で座標のリストをポイントとして追加します。
詳細を表示
add_points(data=None, *, ndim=None, features=None, text=None, symbol='o', size=10,
edge_width=1, edge_color='black', edge_color_cycle=None, edge_colormap='viridis',
edge_contrast_limits=None, face_color='white', face_color_cycle=None,
face_colormap='viridis', face_contrast_limits=None, out_of_slice_display=False, name=None,
metadata=None, scale=None, translate=None, rotate=None, shear=None, affine=None,
opacity=1, blending='translucent', visible=True, shading='none')
- data array ... 点の座標のリスト。D次元のN点を追加する場合、(N, D)という形状になる。
- ndim int ... 空のレイヤーを作るときに、何次元にするかを指定する。
-
features dict or DataFrame ... 各点の何かしらの量。
Labelsと同様だが、Pointsレイヤーでは後述するtextやface_colormapと組み合わせてることで、さらに高度な利用ができる。 -
text str or dict ...
featuresとの組み合わせで、format文字列の形式でポイントにテキストを付随させる。例えば、xy平面上でランダムに点を配置し、座標を$(x, y)$の書式のテキストで追加する場合は次のようになる。イメージとしては、各点に関してtext.format(**properties)のようなコードが走っていると考えるとよい。
arr = np.random.random((8,2))*100 # 8点
viewer.add_points(
arr,
text="({x:.1f}, {y:.1f})",
features={"x": arr[:,1], "y": arr[:,0]}
)
- symbol str ... 点の形。デフォルトの"o"は円に対応する。
- size float ... 点の大きさ。
- edge_width float ... 枠線の太さ。
- edge_color/face_color str ... 枠線/塗りつぶしの色。
- edge_color_cycle/face_color_cycle ndarray or list ... 枠線/塗りつぶしのカラーサイクル。複数の色を順番に使っていく場合に用いる。
-
edge_colormap/face_colormap str ...
propertiesとの組み合わせで、各点に付随した値に応じて枠線/塗りつぶしの色を変えることができる。例えば点の座標に信頼度のようなものがあり、信頼度の高い点ほど赤色で明るくしたいとき、次のようなコードで実現できる。
arr = np.random.random((4, 2))*100 # 4点
features = {"confidence": [0.9, 0.3, 0.6, 0.1]} # 各点の信頼度
viewer.add_points(
arr,
features=features,
face_colormap="red",
face_color="confidence"
)
- edge_contrast_limits/face_contrast_limits ... 枠線/塗りつぶしの色のコントラスト値。
- out_of_slice_display bool ... Trueのとき、各点は少しずれたところの断面でも、少し小さい点として表示される(球を切るイメージ)。
-
shading str ... 影のつけ方。
"spherical"にすると立体的な球になる。
4. Shapes layer
viewer.add_shapes(...)により長方形、折れ線、円などのオブジェクトを追加します。手動での編集、テキストの追加も容易なので、PowerPointのように画像のアノテーションに使ったり、ImageJのROIのように使ったりできます。
オプションはPointsレイヤーとほぼ同じです。キーワード引数の詳細はそちらを参照してください。
詳細を表示
add_shapes(data=None, *, ndim=None, features=None, text=None,
shape_type='rectangle', edge_width=1, edge_color='black', edge_color_cycle=None,
edge_colormap='viridis', edge_contrast_limits=None, face_color='white',
face_color_cycle=None, face_colormap='viridis', face_contrast_limits=None,
z_index=0, name=None, metadata=None, scale=None, translate=None, rotate=None,
shear=None, affine=None, opacity=0.7, blending='translucent', visible=True)
-
data list or array ... 追加するオブジェクトの頂点の座標を指定する。
listで複数同時に追加することができる。頂点の座標の数は次元数やオブジェクトの種類によって変わる。 - shape_type str ... "line", "rectangle", "ellipse", "path", "polygon"のいずれか。
- z_index int or list ... 前後の配置を決める。大きい値ほど前に出る。
例えば長方形を追加する場合
viewer.add_shapes([[2,2],[2,10],[8,10],[8,2]], shape_type="rectangle")
のようになる。
5. Surface layer
viewer.add_surface(...)により、3D表面を表示できます。これは3D物体の境界を定量的に強調したり、地図と標高から立体的な俯瞰図を作成したりするときに用います。
詳細を表示
add_surface(data, *, colormap='gray', contrast_limits=None, gamma=1, name=None, metadata=None, scale=None, translate=None, rotate=None, shear=None, affine=None, opacity=1, blending='translucent', shading='flat', visible=True)
-
data tuple of arrays ... (頂点, 平面を形成する頂点の組, 値)の
tupleで指定する (最後の値は任意)。現実的には3D画像から例えばskimage.measure.marching_cubeを用いて生成することになる。
from skimage.meansure import marching_cubes
verts, faces, _, values = marching_cubes(image)
viewer.add_surface((verts, faces, values))
scikit-imageのサンプル画像である"brain"からSurfaceを生成するとこんな感じになります。
-
colormap, contrast_limits, gamma ...
Imageと同様。 - shading str ... "none", "flat", "smooth"のいずれか。影の付け方を指定する。
6. Track layer
viewer.add_tracks(...)により、時間的に移動している点に残像を付けることで軌跡を表現できます。惑星の運動や、蛍光輝点の追跡結果の表示に非常に便利です。特に、軌跡の分断・結合に対応しており、生細胞のトラッキングのように軌跡が増減する対象にも使えます。
詳細を表示
add_tracks(data, *, features=None, graph=None, tail_width=2, tail_length=30,
name=None, metadata=None, scale=None, translate=None, rotate=None, shear=None,
affine=None, opacity=1, blending='additive', visible=True, colormap='turbo',
color_by='track_id', colormaps_dict=None)
- data array 軌跡データ。D次元データの軌跡は(N, D+1)の形状をした配列で表される。2次元+時間の軌跡であれば、左の列から順にID、時間、y座標、x座標となる。IDが同じ点は一つの軌跡に属すると認識される。例えば時刻t=0-10で動く点とt=4-15で動く点が表現したければ、与えるべき配列は以下のような構成になる。
| ID | t | y | x |
|---|---|---|---|
| 0 | 0 | $y^0_0$ | $x^0_0$ |
| : | : | : | : |
| 0 | 10 | $y^0_{10}$ | $x^0_{10}$ |
| 1 | 4 | $y^1_4$ | $x^1_4$ |
| : | : | : | : |
| 1 | 15 | $y^1_{15}$ | $x^1_{15}$ |
-
features dict or DataFrame ... 各点でのプロパティ。各軌跡ではないので、各プロパティの長さは
dataの行数に一致する。時間経過に伴う軌跡の状態変化なども記述できるということ。 -
graph dict ... 軌跡の分離/融合を
dictで{子:親}の形で表現する。例えばgraph = {1: 0, 2: 0, 3: [1, 2]}であれば、点0が点1,2に分離し、その後点1,2が点3に融合する軌跡が描ける。詳細はこちらのコードで確認できる。 - tail_width, tail_length ... 軌跡の見せ方を変えるパラメータ。
- colormap str ... 軌跡の色付けに用いるカラーマップ。
-
color_by str ... 何で色付けするか。デフォルトではIDごとに色付けされるが、
featuresを与えていれば好みのプロパティで色付けできる。 -
colormaps_dict dict ... 異なるプロパティで異なるカラーマップを使いたいときに指定する。他のカラーマップ関係の引数と異なり、なぜか辞書の値は
napari.utils.Colormapしか受け付けない。
7. Vector layer
viewer.add_vectors(...)によりベクトル場を表示できます。add_imageと組み合わせてポテンシャル場・ベクトル場の時系列変化を可視化できるので、流体力学シミュレーションなどに向いているかと思います。
詳細を表示
add_vectors(data, *, features=None, edge_width=1, vector_style='triangle', edge_color='red',
edge_color_cycle=None, edge_colormap='viridis', edge_contrast_limits=None, length=1,
name=None, metadata=None, scale=None, translate=None, rotate=None, shear=None,
affine=None, opacity=0.7, blending='translucent', visible=True)
-
data array ベクトル場を表す配列。始点/終点を記述するパターン(
scipy.ndimage.map_coordinatesに似た様式)と、各点のベクトル値を与える、ベクトル場らしい記述パターンがある。D次元空間でのdataの形状は- 前者の場合 $(N, 2, D)$ であり、"N"はN本のベクトル、"2"は始点/終点に対応している。
- 後者の場合 $(N_1, ..., N_D, D)$ であり、例えば
data[0, 0, :]は2D平面上の点 $(0, 0)$ におけるベクトルの2成分に対応する。
- features dict or DataFrame ... 各ベクトルのプロパティ。これまで説明したレイヤーと同様。
-
edge_width/edge_color/edge_color_cycle/edge_colormap/edge_contrast_limits ... ベクトルの枠線の太さや色を決める。
Pointsレイヤーと同様。 - vector_style str ... ベクトルの終点をどう表示するか。
- length float ... ベクトルの長さを何倍するか。ベクトルの値が極端に大きかったり小さかったりするときに指定する。
スクリプトからレイヤーへのアクセス
napariのもう一つの強みは、手動で描いた点や図形の座標情報をnumpy.ndarrayとして受け取れる点です。これでPython上での画像解析の弱点を完全に克服できます。
レイヤーをスクリプトから指定して情報を得る
napari.Viewerは多次元画像の可視化に必要ないくつもの変数からなっています。例えば
-
axes(座標軸) -
camera(カメラの向きなど) -
layers(レイヤーリスト) -
scale_bar(スケールバー)
などがあります。
ここではlayersからレイヤーの情報を得ることについて解説します。layersはLayerListクラスのオブジェクトで、Layerオブジェクトを格納したPythonのlistのようなものです。したがって、一番下のレイヤーが欲しければ
layer = viewer.layers[0]
でアクセスします。もしくは、"XXX"という名前のレイヤーであれば
layer = viewer.layers["XXX"]
で指定してもOKです。
ここで得られる変数layerはLayerオブジェクトなので、配列データ、名前、メタデータなどすべて含まれています。それぞれ次のようにアクセスします。
layer.data # 配列データ
layer.name # 名前
layer.metadata # メタデータ
他にもレイヤーの種類によって様々な情報が得られますが、前章で登場したキーワード引数は、ほとんどすべてがそのままレイヤーオブジェクトの属性になっているので、難しくないと思います。
マウスで選択したレイヤーの情報を得る
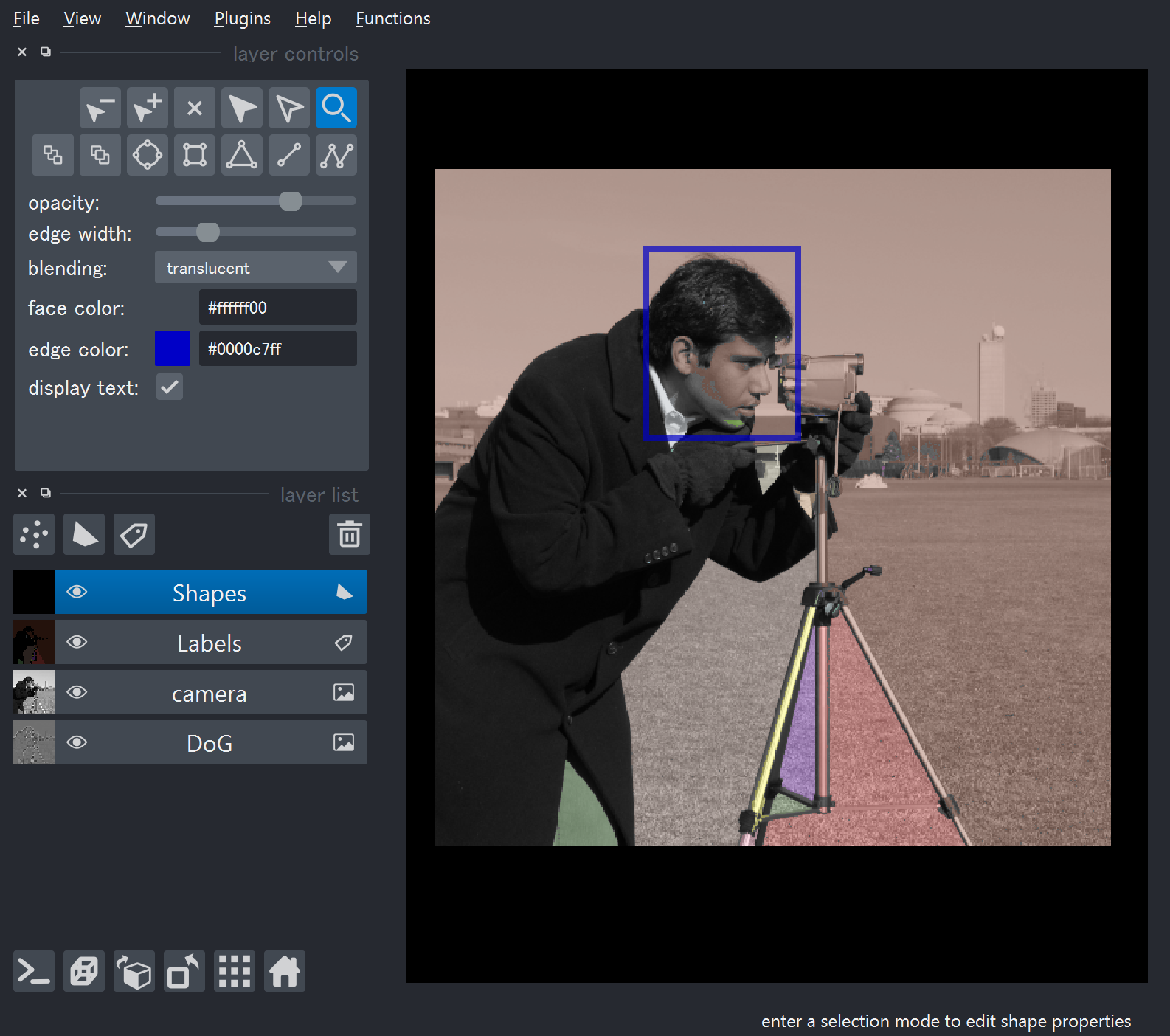
毎回レイヤーの順番や個数は変わりうるので、できればマウスでクリックしてレイヤーを指定したいです。例えば以下のように、サンプル画像にフィルタやラベル付けをし、顔の部分を手動で囲った状況で、長方形の座標を得たいとしましょう。レイヤーリストを見ると"Shapes"が選択されています。
これはLayerListクラスのselectionプロパティで得ることができます。
viewer.layers.selection
[Out] Selection({<Shapes layer 'Shapes' at 0x...>})
見ての通り選択されているレイヤーがPythonのsetのようなものに格納されているので、次のようにアクセスする必要があります。
viewer.layers.selection.pop() # OK。pop()なので選択は解除される。
list(viewer.layers.selection)[0] # OK。もとのsetは変わっていないので選択は解除されない。
viewer.layers.selection[0] # Error!
これで、コード自体は変えずに、状況に応じて手動で選択できるようになりました。
layer = viewer.layers.selection.pop()
layer.data[0] # 0番目の図形の座標
[Out]
array([[ 60.44640515, 159.68169239],
[ 60.44640515, 274.63154028],
[203.29670156, 274.63154028],
[203.29670156, 159.68169239]])
終わりに
読んでいただきありがとうございます。napariの多彩な機能は1回では紹介しきれないので、また次回に回します。予定としては
- よく使う自作関数をキーボードショートカットとして登録する
- マウスの動きを認識させてViewerでライブで解析する
- ウィジェットを追加する
などを書いていこうかと思います。他にもご要望があればコメント欄にどうぞ!
追記 (2021/08/09)
続きはこちらから。