なぜやるか
Chainer1.5から高速化されています。高速化の一因としてcythonを使用していることが挙げられていたので、exampleをcythonで書き換えたら、どれだけ早くなるのか単純な疑問が湧いたので、試してみました。
PCのスペック
OS:OS X Yosemite
CPU: 2.7GHz Intel Core i5
Memory:8GHz DDR3
条件
exampleのMnistを使用
学習回数:20回
データは予めダウンロードされた状態
可視化
プロファイラによる可視化
pycallgraphを使用
http://pycallgraph.slowchop.com
graphivzをインストール
http://www.graphviz.org/Download_macos.php
X11をインストール(Yosemiteの場合)
http://www.xquartz.org/
failed with error code 256が出た場合
https://github.com/gak/pycallgraph/issues/100
pycallgraphを使用する場合
python pycallgraph graphviz -- ./ファイル名.py
やろうと思うこと
1:通常処理の可視化、プロファイリング
2:単純なcython化
3:cdefによる静的な型設定
4:外部モジュールのcython化
初期の状態
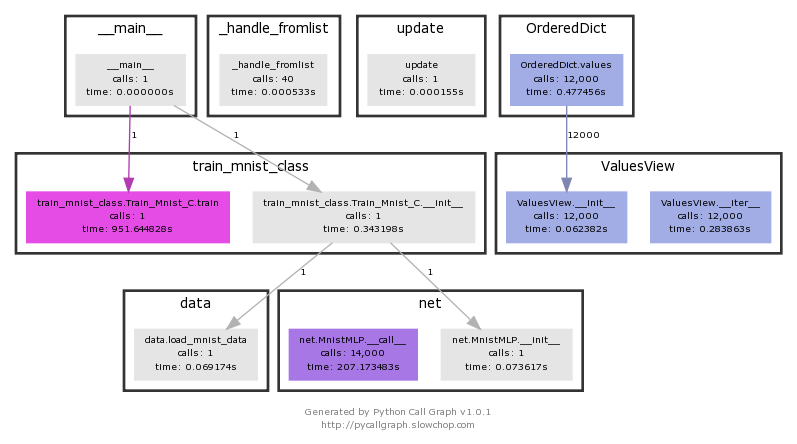
ビジュアライゼーションを行うとどの部分に時間がかかっているかを把握できます。
train_mnist.Train_Mnist.train
951秒かかっていることが分かります。
通常のプロファイルの結果が下記です。
ncalls: 呼び出し回数
tottime: この関数が消費した時間の合計
percall: tottime を ncalls で割った値
cumtime: 下位の関数を含むこの関数の (実行開始から終了までの) 消費時間の合計。この項目は再帰的な関数においても正確に計測されます。
percall: cumtime をプリミティブな呼び出し回数で割った値
今回はcythonの都合上、コードを変えているので上記の処理時間と異なります。本当はcythonでpycallgraphを使用したかったのですが、筆者の知識不足で使用できませんでした。使用方法知っている人いたら教えてください(通常の使用方法だとcython部分の処理が載りません)
755.154秒で終了しています。
実行方法
python -m cProfile
Profile.prof
37494628 function calls (35068627 primitive calls) in 755.154 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 448.089 0.006 448.651 0.006 adam.py:27(update_one_cpu)
114000 187.057 0.002 187.057 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 31.576 0.000 31.576 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 23.122 0.002 163.601 0.014 variable.py:216(backward)
2:8の法則に基づいて、もっとも処理に時間がかかっている部分に着目します。
adam.pyがほぼ大半の処理時間をとっており、numpyの行列演算が続いて処理時間を割いていることが分かります。
cython
cythonにおいてもグラフ化したかったのですが、筆者の知識が足りず、cythonの処理部分だけグラフ化できなかったのでプロファイリングしました。
結果は800秒と逆に遅くなっています
Profile.prof
37466504 function calls (35040503 primitive calls) in 800.453 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 473.638 0.007 474.181 0.007 adam.py:27(update_one_cpu)
114000 199.589 0.002 199.589 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.706 0.000 33.706 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.754 0.002 173.816 0.014 variable.py:216(backward)
28000 9.944 0.000 10.392 0.000
adam.pyとvariable.pyの処理部分がcython化する前より遅くなっています。
cythonにより変換したc言語とpythonの外部処理の連携で遅くなっている可能性があります。
cdef
cdefを用いて、予め静的な型を定義すると早くなるだろうと見込んでcdefの定義をしました。
事前準備
そのままmac上で使用するとエラーが出たので諸所の対応をしました。
cimport の使用を試みると下記のエラーが出る。
/Users/smap2/.pyxbld/temp.macosx-10.10-x86_64-3.4/pyrex/train_mnist_c2.c:242:10: fatal error: 'numpy/arrayobject.h' file not found
下記のディレクトリに
/usr/local/include/
下記のコマンドで探したヘッダーディレクトリをコピーまたはパスを通す
find / -name arrayobject.h -print 2> /dev/null
776秒となりました。
Profile.prof
37466756 function calls (35040748 primitive calls) in 776.901 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 458.284 0.006 458.812 0.006 adam.py:27(update_one_cpu)
114000 194.834 0.002 194.834 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 33.120 0.000 33.120 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 24.025 0.002 168.772 0.014 variable.py:216(backward)
単純なcython化よりは改善していますが、adam.pyとvariable.pyにあまり変化がないため、余計なC言語とPython言語の変換処理のため結果的にpython処理より遅くなっています。
adam.pyのcython化
もっとも処理に時間がかかる部分をadam.pyをcython化することによって高速化できないか試してみました。
結果として30秒ほど早くなる効果を見せました。
Profile.prof
37250749 function calls (34824741 primitive calls) in 727.414 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
72000 430.495 0.006 430.537 0.006 optimizer.py:388(update_one)
114000 180.775 0.002 180.775 0.002 {method 'dot' of 'numpy.ndarray' objects}
216000 30.647 0.000 30.647 0.000 {method 'fill' of 'numpy.ndarray' objects}
12000 21.766 0.002 157.230 0.013 variable.py:216(backward)
adam.pyを内包しているoptimizer.pyの処理時間がpython時より20秒ほど早くなっています。これが効いて早くなりました。
総括
単純にcython化して高速化を試みるのではなく、プロファイリングして、本当に効果がある所だけcython化すると効果が出る可能性があることが分かりました。
なんとなくするのではなく、仮説、可視化、検証のサイクルが重要ということが体験できたアドベントカレンダーでした。
実行に試したコードは下記に載せています。
https://github.com/SnowMasaya/chainer_cython_study
参考
Chainer: a neural network framework
https://github.com/pfnet/chainer