忙しくて論文をチェックしきれないとき、あるいはなにか新しいことに手を出すときにさらっと概要を(できれば既存知識とも結びつけて)説明してほしい、と思ったことがある人は多いんじゃないでしょうか。
今回は勉強もかねて、そういうときに使えそうなものがRDFを用いてつくれないか挑戦してみようと思います。
RDFについて
Resource Description Frameworkの略。S(Subject)、P(Predicate)、O(Object)という3つの値を使って有向グラフとして表現されるものです。データをつなげていくことができて、しかもクエリで知りたい情報を取り出せる仕組みもあります。
参考記事:
RDFに関する雑な説明 - Qiita
[直感RDF!! その2 -使いやすいRDFを作って,検索しよう。 - Qiita]
(http://qiita.com/maoringo/items/0d48a3d967a35581cc24)
論文データの準備
論文情報はNCBIが提供するPubMedならAPIで情報が取得できるのでそれを使ってみます。
提供されているAPIには以下の4種類があります。
- ESearch: 検索ワードに対する論文ID(PubMed ID)を返す
- ESummary: 論文IDに対するタイトルや著者名を返す
- EFetch: 論文IDに対するすべての情報を返す
- ESpell: 検索ワードのスペルチェックを行う
まず、ESearchで論文ID一覧を取得して、各論文IDに関する詳細を取得する必要があるようですね。ESpellについては今回は必要なさそうです。
参考記事
PubMed APIのまとめ
論文IDを取得してみよう
論文IDの取得にはESearchを使います。このURLがベースとなって
http://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=
"term="のあとに検索キーワードをいれればそのIDが返されるはずです。
例えば、検索キーワード: "cancer"(がん)でやってみます。
http://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term=cancer
ブラウザで上記URLを入力するとこのような結果が表示されます。
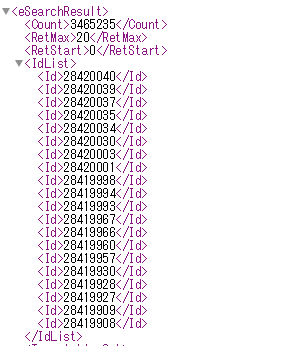
論文IDリストが取得できていますね。
ただ、毎回手動でやるのも大変だし、不要なものを消して論文IDだけにしたいですね。そこでpythonを使って書いていくことにします。
環境情報
- Windows10
- Python 3.6.0
- Anaconda 4.3.1
# coding: utf-8
import urllib.request
keyword = "cancer"
baseURL = "http://eutils.ncbi.nlm.nih.gov/entrez/eutils/esearch.fcgi?db=pubmed&term="
def get_id(url):#論文IDを取得する
result = urllib.request.urlopen(url)
return result
def main():
url = baseURL + keyword
result = get_id(url)
print(result.read())
if __name__ == "__main__":
main()
これを実行すると
% python get_id.py
<?xml version="1.0" encoding="UTF-8" ?><!DOCTYPE eSearchResult
PUBLIC "-//NLM//DTD esearch20060628//EN""https://eutils.ncbi.n
lm.nih.gov/eutils/dtd/20060628/esearch.dtd"><eSearchResult><Cou
nt>3465235</Count><RetMax>20</RetMax><RetStart>0</RetStart><IdL
ist><Id>28420040</Id><Id>28420039</Id><Id>28420037</Id>
....
改行なしで見にくいですが、さっきブラウザでやっときと同じ情報が取得できているのが確認できます。
論文IDだけを抜き出してくる
XMLは基本的に
<要素>内容</要素>
<要素="要素名" 属性="属性値">内容</要素>
のような構造になっています。例えば、論文IDなら
<Id>論文ID</Id>
のようになっているのが取得情報を見ればわかると思います。要素など不要な部分は取り除いて、必要な論文IDだけを抜き出してくるようにします。
XMLを扱うためのElementTreeというライブラリがあるのでそれを使用することにします。
from xml.etree.ElementTree import *
インポートしてからmainを以下のように書き換えます。
def main():
url = baseURL + keyword
result = get_id(url)
element = fromstring(result.read())
print(element.findtext(".//Id"))
まず、fromstring()でElementオブジェクトを作成します。その後のelement.findtext()は条件にマッチした最初の内容を返してくれるものです。今回は"Id"が欲しいのでそれを指定するのですが、".//Id"というように書く決まりがあります。
これを実行すると
% python get_id.py
28420040
最初の論文IDだけを抜き出してくることができました。最初の1つだけでなくマッチする内容をすべて抜き出してきてほしいときはelement.findall()を使って次のように書きます。
def main():
url = baseURL + keyword
result = get_id(url)
element = fromstring(result.read())
for e in element.findall(".//Id"):
print(e.text)
実行すると
% python get_id.py
28420040
28420039
28420037
28420035
...
うまくすべての論文IDだけを抜き出してくることができました。
今後の処理のことも考えて、"idlist_検索ワード.txt"というファイルをつくって取得したIDリストを保存するようにしておきます。
def main():
url = baseURL + keyword
result = get_id(url)
element = fromstring(result.read())
filename = "idlist_"+keyword+".txt"
f = open(filename, "w")
for e in element.findall(".//Id"):
f.write(e.text)
f.write("\n")
f.close()
論文IDから論文summaryを取得しよう
次に取得した論文IDを使ってsummaryを取得してみましょう。ESummaryのベースとなるURLは
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id=
です。論文ID取得のときと同じように"id="のあとに情報を取得したい論文IDをいれてあげます。例えば、先ほど取得した論文IDの最初のもの"28420040"をいれてみましょう。
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id=28420040
このURLをブラウザで入力するとこのように出版年月日や著者名、論文タイトルの情報が取得できました。

ここまでをPythonで書いてあげると
# coding: utf-8
import urllib.request
from xml.etree.ElementTree import *
keyword = "cancer"
idfile = "idlist_"+keyword+".txt"
baseURL = "https://eutils.ncbi.nlm.nih.gov/entrez/eutils/esummary.fcgi?db=pubmed&id="
def get_xml(url):#論文summaryを取得する
result = urllib.request.urlopen(url)
return result
def main():
idlist = []
f = open(idfile,"r")
for i in f.readlines():
idlist.append(i.strip())
f.close()
url = baseURL + idlist[0]
result = get_xml(url)
print(result.read())
if __name__ == "__main__":
main()
論文IDは保存したファイルから読み込む形式にしています。また、ここでは使っていませんが、この後すぐ使うので先にElementTreeライブラリもすでにインポートしています。実行すると論文ID取得のときのように、ブラウザで実行した場合の改行なしバージョンが出力されるはずです。
欲しい内容だけ抜き出してくる
あとは論文IDのときと同様欲しい内容の部分だけを抽出するだけです。しかし、論文IDのときと違ってAuthorやTitleはItem要素の属性になっています。そのため、論文IDのときのように
for e in element.findall(".//Item"):
print(e.text)
とすると、このように論文情報をすべて抜き出してきます。
% python get_summary.py
28420040
2017 Apr 18
2017 Apr 18
J Surg Oncol
None
Duan W
Liu K
Fu X
Shen X
...
これはこれで使えますが、AuthorやTitleなど欲しいものだけを抜き出してくる方法も知っておきましょう。
XMLテキストを渡してつくられるElementオブジェクトは辞書型オブジェクトになっていて、各要素にアクセスすることができるようになっています。いくつか例を挙げます。
print(element[0][3].text)
print(element[0][4][2].text)
print(element[0][6].text)
実行結果
2017 Apr 18
Fu X
Semi-end-to-end esophagojejunostomy after laparoscopy-assisted total gastrectomy better reduces stricture and leakage than the conventional end-to-side procedure: A retrospective study.
著者一覧を抜き出すならこんな感じです。
for i in range(len(element[0][4])):
print(element[0][4][i].text)
実行結果
Duan W
Liu K
Fu X
Shen X
...
また、要素(tag)や属性(key)の取得は
print(element[0][4].tag)
print(element[0][4].attrib)
print(element[0][4].keys())
実行結果
Item
{'Name': 'AuthorList', 'Type': 'List'}
['Name', 'Type']
のようにできます。覚えておくと便利だと思います。
論文の要旨を取得しよう
論文の情報を取得することはできましたが、タイトルや著者名だけではちょっと物足りません。論文に関するキーワードがついてればよかったんですが仕方ないですね。そこでEFetchを使って論文の要旨を取得しようと思います。
まずEFetchのベースとなるURLは
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&id=
すごい扱いずらそうな形式で返されてしまいました。調べてみるとretmodeというパラメーターでXML形式を指定できるようです。
URLを以下のようにして実行してみると
https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&id=28420040&retmode=xml
ここまではESearchとほとんど同じ要領で書くことができます。
# coding: utf-8
import urllib.request
from xml.etree.ElementTree import *
keyword = "cancer"
idfile = "idlist_"+keyword+".txt"
baseURL = "https://eutils.ncbi.nlm.nih.gov/entrez/eutils/efetch.fcgi?db=pubmed&id="
def get_xml(url):#論文情報を取得する
result = urllib.request.urlopen(url)
return result
def main():
idlist = []
f = open(idfile,"r")
for i in f.readlines():
idlist.append(i.strip())
f.close()
url = baseURL + idlist[0] + "&retmode=xml"
result = get_xml(url)
print(result.read())#取得結果をそのまま表示
if __name__ == "__main__":
main()
あとは、論文要旨がAbstractText要素なので論文IDを抽出した時の要領で
element = fromstring(result.read())
for e in element.findall(".//AbstractText"):
print(e.text)#論文要旨を表示
とすればできるはずです。
試してみると論文要旨だけをうまく抜き出してくることができました。
% python get_abstract.py
Laparoscopy-assisted total gastrectomy (LATG) has not
gained popularity due to the technical difficulty of e
sophagojejunostomy (EJ) and the high incidence of EJ-r
elated complications. Herein, we compared two types of
EJ for Roux-en-Y reconstruction to determine whether
...
こうして取得してきた論文要旨をうまく処理して知識をRDF化していけるならなかなかおもしろいものがつくれそうです。論文情報取得するまでで結構長くなってしまったので続きは次の記事にしたいと思います。
補足
一度保存したXMLファイルに対して処理を行う場合は
element = fromstring(result.read())
を
tree = parse("efetch_result.xml")
element = tree.getroot()
のように置き換えれば可能です。

