目的
東京都が公開している新型コロナウィルス(COVID-19)の陽性患者のデータを使ってPythonのグラフを描いてみました。
必要最低限のコードで書いていますので、これからPythonを使ってデータ解析を行ってみようと思っている人のご参考になれば幸いです。
東京都が毎日更新しているcsv形式の公開データを直接読み込むようにしてあるので、csvファイルをいちいちダウンロードしたりする必要がありません。
下記のPythonコードをご自身の実行環境(Jupyter Notebookなど)にコピーしておけば、毎回、最新情報のグラフが描画できるようになっています。
また日本全国版のcsvデータへのリンクを本記事の後半に付けておきましたので、そちらを使って練習して頂くとスキルが身につきやすいかと思います。
Python実行環境
この記事のPythonコードは、AnacondaがインストールされたWindows10マシンを使用し、Jupyter Labを使って動作確認しています。
データソース
csvデータ
今回グラフ化したデータは以下のcsvデータとなります。前日までの結果が毎日アップデートされています。
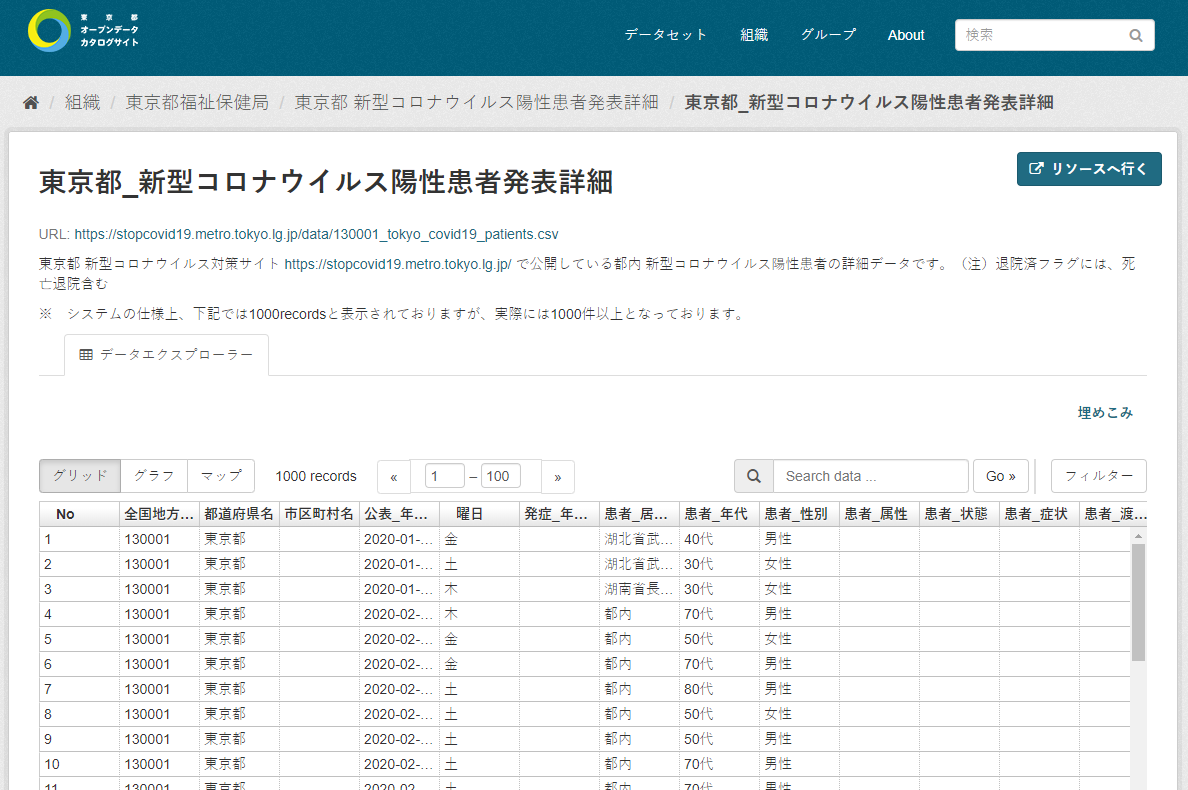
東京都_新型コロナウイルス陽性患者発表詳細(CSV形式)
ホームページ
以下はcsvデータへのリンクが掲載されているホームページです。
東京都_新型コロナウイルス陽性患者発表詳細
Pythonでグラフ作成
それではcsvデータを使ってPythonでグラフを描いていきます。
まずはデータ読み込み
まずは以下に記したPythonコードを使って、東京都のホームページに接続し最新のデータ(csv形式)の取得とpandasのDataFlameに変換を行います。
ここでのポイントは、csvファイルをローカルフォルダに保存せず、直接pandasのDataFlame(df)に変換している点です。
これにより、以下のコードを実行するだけで、毎日更新される最新版のcsvファイルをブラウザを開いてダウンロードする手間を省くことができます。
import requests
import pandas as pd
import io
# csvを直接pandasのdataframeに取り込む
url = 'https://stopcovid19.metro.tokyo.lg.jp/data/130001_tokyo_covid19_patients.csv'
r = requests.get(url).content
df = pd.read_csv(io.StringIO(r.decode('utf-8')))
df
無事データが読み込まれたらDataFrame(df)の中身が表示されるはずです。
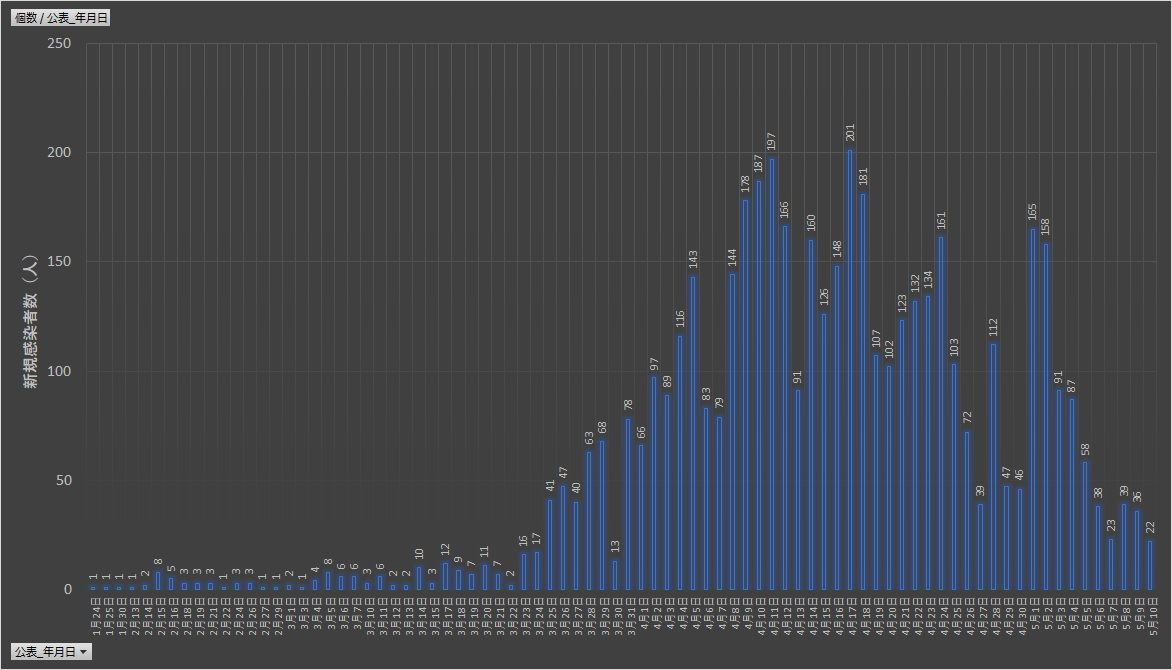
新規感染者の推移グラフ
上記で読み込んだDataFrame(df)を使ってグラフを描いていきます。まずは横軸が日付、縦軸が感染者数の棒グラフとなります。続けて以下のコードを実行してみましょう。
((5/15追記)) 元のcsvデータの順番が時系列にならばなくなったので、以下のコードの中央付近に公表_年月日の順番にデータを並び替えるコードを一行追記しました。
# グラフを書くためにmatplotlib.pyplotとseabornをインポートする
import matplotlib.pyplot as plt
import seaborn as sns
%matplotlib inline
# グラフを書く
plt.figure(figsize=(13,7)) # グラフのサイズを定義
sns.set(font='Yu Gothic', font_scale = 1.2) # 日本語が文字化けするのでフォントを指定
df = df.sort_values('公表_年月日') #公表_年月日の順番にデータを並び替える(5/15追記)
sns.countplot(data=df, x='公表_年月日') # Seabornを使って感染者数の集計グラフを作成する。
plt.title('COVID-19新規感染者数の推移@東京都')
plt.xticks(rotation=90, fontsize=10) # x軸の日時が重なるので90°回転させて表示
plt.ylabel('感染者数(人)') # y軸のラベルを'感染者数'と表示
以下のようなグラフが描画されましたでしょうか。
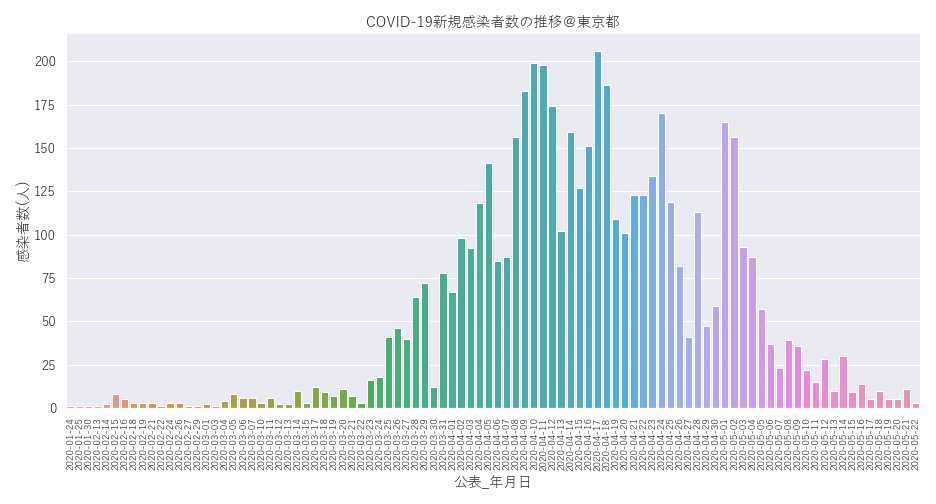
だいぶ収束してきた感じですが、この先いったいどうなるのやら。。。
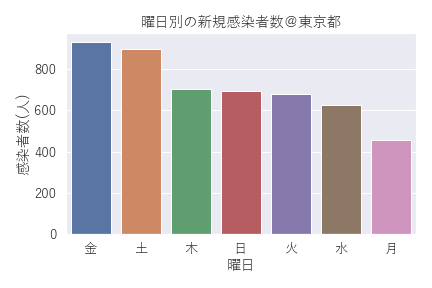
曜日別の感染者数のグラフ
ちなみに同じ棒グラフを使って、横軸を曜日別にしてみると、
# 感染者数の曜日別のグラフを描く
sns.countplot(data=df, x="曜日") # グラフを描く
plt.title('曜日別の新規感染者数@東京都') # グラフタイトルを表示
plt.ylabel('感染者数(人)') # 縦軸のタイトルを表示
曜日を並び替えるには、下記のように書き換えてみて下さい。
# グラフの横軸を並び替えて再度グラフを描く
list_weekday = ['月','火','水','木','金','土','日'] # 横軸の順番を示すリストを作る
sns.countplot(data=df, x="曜日",order=list_weekday) # グラフを描く
plt.title('曜日別の新規感染者数@東京都') # グラフタイトルを表示
plt.ylabel('感染者数(人)') # 縦軸のタイトルを表示
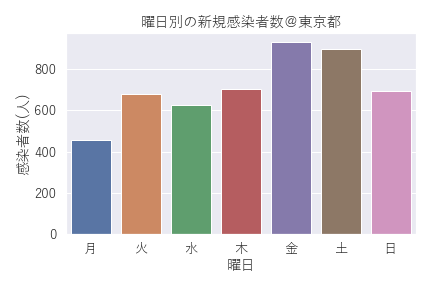
無事、曜日順に綺麗に並び替えられました。週末の金曜と土曜の数が多く、日曜と月曜の数が少ないようです。
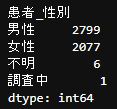
感染者数の男女比のグラフ
続いて男女比は、、、
# 感染者数の男女別のグラフを描く
sns.countplot(data=df, x="患者_性別") # グラフを描く
plt.title('男女別の新規感染者数@東京都') # グラフタイトルを表示
plt.ylabel('感染者数(人)') # 縦軸のタイトルを表示
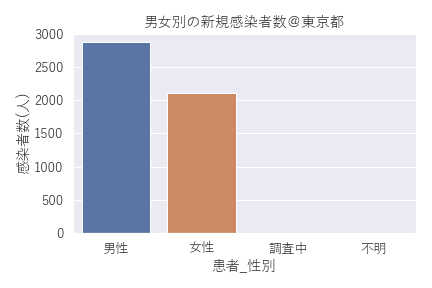
こちらは連日報道されている通り男性の方が多い傾向ですが、、、
それよりも「男性」と「女性」以外に「調査中」と「不明」の項目もデータに含まれていることが分かりました。
こういった予期せぬ発見はデータ解析においてよくある話ですが、
念のためpivot_tableで患者_性別のデータを集計してみましょう。元データから下記の1行で集計できます。
# 患者_性別のデータの集計を行う
df.pivot_table(index='患者_性別',aggfunc='size').sort_values(ascending=False)
つまり患者_性別の項目には、
「男性」と「女性」以外にも6つの「不明」と1つの「調査中」が混ざっている模様です。
データを解析していると予期せぬ項目が含まれていたりするのはよくある話なので、
グラフの可視化だけでなくデータの集計や前処理のテクニックも覚えておくことはとても大切です。
年代別の感染者数のグラフ
続いて年代別は、、、
list_age = ['10歳未満','10代','20代','30代','40代','50代','60代','70代','80代','90代','100歳以上','不明']
sns.countplot(data=df, x="患者_年代", order=list_age)
plt.xticks(rotation=90)
plt.ylabel('感染者数(人)')
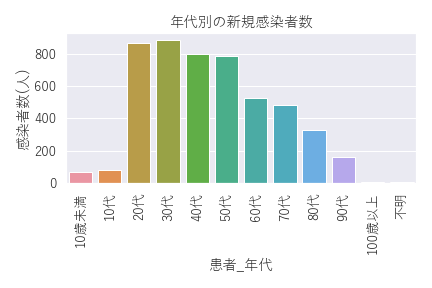
こうやって見ると60代以上の高齢者の割合はもちろんですが、人口の割に20代と30代の感染者数が多そうに見えますね。
(人口の割には40代と50代の割合が少ないと表現した方が良いのかもしれませんが。)
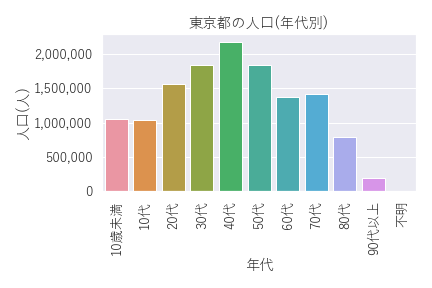
東京都の年代別人口のグラフ(参考)
参考までに以下に東京都の年代別人口(令和2年1月1日現在)のグラフ1を示しておきます。
| 年代 | 総人口 | 男性人口 | 女性人口 |
|---|---|---|---|
| 10歳未満 | 1,048,921 | 536,920 | 512,001 |
| 10代 | 1,029,680 | 526,065 | 503,615 |
| 20代 | 1,557,966 | 779,053 | 778,913 |
| 30代 | 1,842,086 | 939,710 | 902,376 |
| 40代 | 2,177,935 | 1,108,561 | 1,069,374 |
| 50代 | 1,832,946 | 946,158 | 886,788 |
| 60代 | 1,373,395 | 688,654 | 684,741 |
| 70代 | 1,414,012 | 645,774 | 768,238 |
| 80代 | 794,805 | 304,309 | 490,496 |
| 90代以上 | 185,849 | 47,609 | 138,240 |
| 不明 | 1 | 0 | 1 |
年代別人口当たりの感染者数(男女合計)
そして上記の年代別の感染者数を年代別人口で割り算して10万人当たりの感染者数を比較したグラフが以下となります。
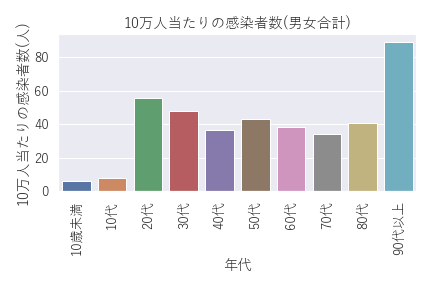
ちょっと意外でした。。。90代以上が圧倒的で、次いで20代、30代、、、そして、40代から80代まではあまり差が無いという傾向のようです。
年代別人口当たりの感染者数(男女別)
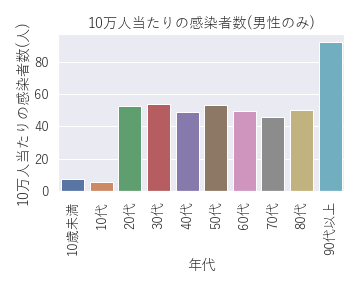
そして男性と女性に分けてみると。
 |
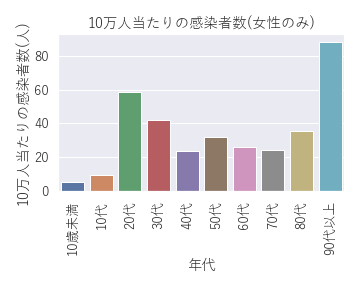 |
| これまた意外な結果です。てっきり20代の感染者は男性が多いのかと思っていましたが、20代と30代の感染者が多い傾向を示しているのは女性でした。原因は分かりませんが少し気になる結果です。 |
年代別と日付のヒートマップ
そして年代別と日付のヒートマップを見てみると、、、
# 公表_年月日と患者_年代の列でピボットテーブルを作成
df_pivot = df[['公表_年月日','患者_年代']].pivot_table(index='公表_年月日',columns='患者_年代',aggfunc='size')
# 患者_年代の各項目をリスト化(ヒートマップの縦軸に使用)
list_age = ['10歳未満','10代','20代','30代','40代','50代','60代','70代','80代','90代','100歳以上','不明']
plt.figure(figsize=(6,16)) # グラフのサイズを定義
plt.yticks(fontsize = 10) # y軸のフォントサイズを定義
sns.heatmap(df_pivot[list_age], annot = True, annot_kws={"size": 10}, linewidth = .1) # ヒートマップを描画
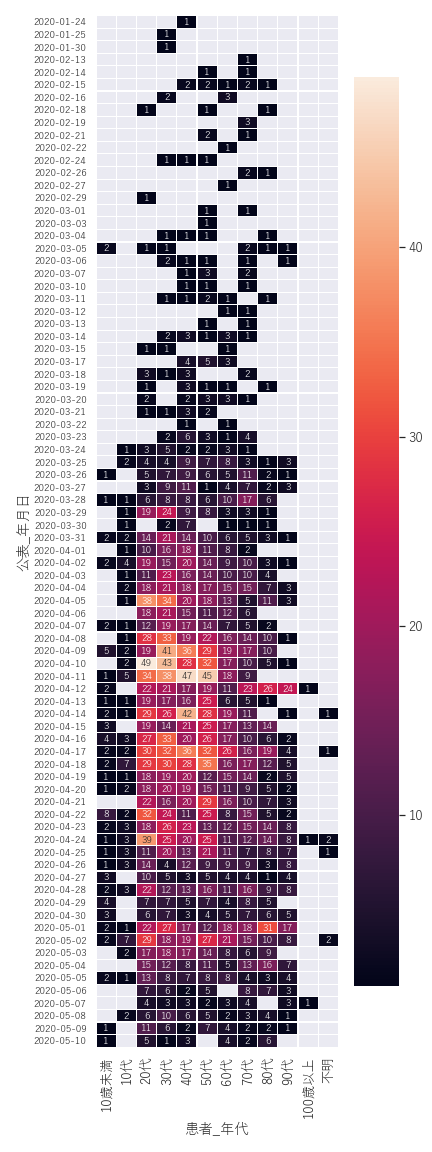
見た目はそれっぽいですが「だからなんなんだ」って感じですね。。。(-_-;)
他にも情報が引き出せそうなのでぼちぼち解析を続けてみようと思います。
データのトリミングについて
そういえば生データ(csv)の中身を全く確認していませんでしたが、csvデータは冒頭のコードでDataFrame(df)に変換済みですので、以下のコマンドでデータの中身を再度表示させて見ましょう。
df

データは4,883行(2020.5.12時点)あるのですが、空欄を示すnanがたくさん含まれているようです。ここで念のため、各列に含まれるユニークな値を見てみます。以下のコードを実行してみて下さい。
# csvデータを格納したデータフレーム(df)の列名と各列に格納されているユニークな値を抽出する。
for i in df.columns: # 各列について繰り返す
print('列名:' + i) # 列の名前を出力する
print('ユニーク値の数:' + str(len(df[i].unique()))) # 各列のユニークな値の数を数える
print('ユニーク値:' + str(df[i].unique())) # 各列のユニークな値を抽出する
print('///////////////////////////////////////////') # 区切り線
結果が長いので以下に折りたたんで格納しておきました。
実行結果(クリックして下さい)
列名:No ユニーク値の数:4987 ユニーク値:[ 1 2 3 ... 10109 10110 10111] /////////////////////////////////////////// 列名:全国地方公共団体コード ユニーク値の数:1 ユニーク値:[130001] /////////////////////////////////////////// 列名:都道府県名 ユニーク値の数:1 ユニーク値:['東京都'] /////////////////////////////////////////// 列名:市区町村名 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:公表_年月日 ユニーク値の数:84 ユニーク値:['2020-01-24' '2020-01-25' '2020-01-30' '2020-02-13' '2020-02-14' '2020-02-15' '2020-02-16' '2020-02-18' '2020-02-19' '2020-02-21' '2020-02-22' '2020-02-24' '2020-02-26' '2020-02-27' '2020-02-29' '2020-03-01' '2020-03-03' '2020-03-04' '2020-03-05' '2020-03-06' '2020-03-07' '2020-03-10' '2020-03-11' '2020-03-12' '2020-03-13' '2020-03-14' '2020-03-15' '2020-03-17' '2020-03-18' '2020-03-19' '2020-03-20' '2020-03-21' '2020-03-22' '2020-03-23' '2020-03-24' '2020-03-25' '2020-03-26' '2020-03-27' '2020-03-28' '2020-03-29' '2020-03-30' '2020-03-31' '2020-04-01' '2020-04-02' '2020-04-03' '2020-04-04' '2020-04-05' '2020-04-06' '2020-04-07' '2020-04-08' '2020-04-09' '2020-04-10' '2020-04-11' '2020-04-12' '2020-04-13' '2020-04-14' '2020-04-15' '2020-04-16' '2020-04-17' '2020-04-18' '2020-04-19' '2020-04-20' '2020-04-21' '2020-04-22' '2020-04-23' '2020-04-24' '2020-04-25' '2020-04-26' '2020-04-27' '2020-04-28' '2020-04-29' '2020-04-30' '2020-05-01' '2020-05-02' '2020-05-03' '2020-05-04' '2020-05-05' '2020-05-06' '2020-05-07' '2020-05-08' '2020-05-09' '2020-05-10' '2020-05-11' '2020-05-12'] /////////////////////////////////////////// 列名:曜日 ユニーク値の数:7 ユニーク値:['金' '土' '木' '日' '火' '水' '月'] /////////////////////////////////////////// 列名:発症_年月日 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:患者_居住地 ユニーク値の数:7 ユニーク値:['湖北省武漢市' '湖南省長沙市' '都内' '都外' nan '調査中' '―'] /////////////////////////////////////////// 列名:患者_年代 ユニーク値の数:13 ユニーク値:['40代' '30代' '70代' '50代' '80代' '60代' '20代' '10歳未満' '90代' '10代' '100歳以上' '不明' '-'] /////////////////////////////////////////// 列名:患者_性別 ユニーク値の数:4 ユニーク値:['男性' '女性' '調査中' '不明'] /////////////////////////////////////////// 列名:患者_属性 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:患者_状態 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:患者_症状 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:患者_渡航歴の有無フラグ ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:備考 ユニーク値の数:1 ユニーク値:[nan] /////////////////////////////////////////// 列名:退院済フラグ ユニーク値の数:2 ユニーク値:[ 1. nan] ///////////////////////////////////////////少なくとも以下の列についてはすべてが空欄(nan)であるようです。
- 「市区町村名」
- 「発症_年月日」
- 「患者_属性」
- 「患者_状態」
- 「患者_症状」
- 「患者_渡航歴の有無フラグ」
- 「備考」
また「全国地方公共団体コード」と「都道府県名」はすべて同じ値となっており、データ解析において意味をなさないものとなっています。
こういった不要なデータはあらかじめデータから除いておくのが望ましいです。必要な項目だけ抜き出して新たなデータフレーム(df_extract)を作成します。以下のコードを実行して下さい。
# 不要な列をトリミングする(必要な列のみを抽出する)
df_extract = df[['No','公表_年月日','曜日','患者_居住地','患者_年代','患者_性別','退院済フラグ']]
df_extract = df_extract.set_index('No') # 「No」の列をindexに設定する。
df_extract
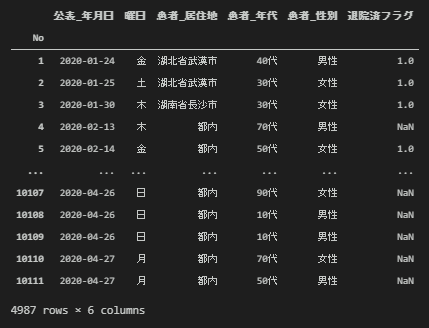
これでだいぶスッキリしました。データを解析する上で不要なデータを適切に判断して除外するトリミング作業もとても重要なスキルだと思います。
練習用データ
今回は東京都のデータでしたが、
ジャッグジャパン株式会社が全国版のcsvデータを公開しています。
https://dl.dropboxusercontent.com/s/6mztoeb6xf78g5w/COVID-19.csv
データ数も多くPythonを使ったデータ解析の練習には丁度良いと思います。
ほぼ同じ流れで進められますので、ご興味ある方は是非ご自身で取り組んでみてはいかがでしょう。
おまけ
実はExcelでもほぼ同じ事ができる(しかも比較的簡単に)
以下は同じデータを使ってExcelで描いたピボットグラフです。
実は本記事で紹介したヒートマップを含めほぼ同じ事がExcelでも簡単にできます。
私もPythonが大好きで、なんでもPythonでという気持ちは山々なのですが、何のためのデータ解析か、誰のためのデータ解析かということを考えると、ExcelでできることはExcelでやる、ExcelでできることはPythonではやらないというのを基本スタイルにしてゆくべきなんだと日々思っております。
Excelで描画した新規感染者の推移グラフ
Excelで描画したヒートマップ(もどき)
以上、最後までご精読いただきありがとうございました。
自らのスキルアップも兼ねて継続的にアップデートを進めていこうと思います。