- 製造業出身のデータサイエンティストがお送りする記事
- 今回は製造業で使える異常検知手法を実装し整理しました。
はじめに
最近では製造業の現場でも機械学習を活用した取組みが増え始めていると思います。
今回は、異常検知プロジェクトで活用した多変量統計的プロセス管理(MSPC)を整理しました。
製造業における異常検知とは
製造業では、予防保全という言葉があります。予防保全とは、生産ラインにおける機械設備の故障、不具合発生、性能低下などを未然に防ぐ保全方法を指しており、設備が壊れて生産ラインがストップすることを防ぐ目的で行っております。
他にも通常操業から異常な製品が生産されることを未然に防ぐために操業の異常検知などもあります。
今回、ご紹介する異常検知手法(MSPC)はどちらにも活用できる手法となっております。
多変量統計的プロセス管理(MSPC)とは
MSPCで使用するデータは、使用するデータは正常データのみで学習を行います。
一般的に機械学習などで分類を行う際は、正常データと異常データを同程度のサンプルを集めないと適切な分類ができないと思われがちですが、実際の現場では異常データはほとんど無く、正常データのみたくさんある状況です。
(実際、「異常データ=設備が故障する」と言うことなので、工場の生産がストップした際のデータになります。そんなデータが少ないのは当たり前です。そのために、製造現場では予防保全に力を入れており、少し過剰なぐらいのレベルで保全活動を行っております。)
基本的な異常検知の考え方は、多次元空間内に正常データが存在する領域を設定し、その領域から逸脱したデータを観測した際に異常と判定します。そのため、正常データのみ学習することで異常検知モデルを構築する事ができます。
次に正常状態と異常状態の境界となる管理限界の設定の仕方について説明します。
少し天下り的になりますが、通常、変数間には何かしらの関係(相関関係)があり、正常データが含まれる領域を適切に定める際に変数間の関係性を捉える必要があります。つまり、2変数なら楕円、n変数なら超楕円を用意する必要があります。
MCPCでは変数間の関係性を考慮する手法として、主成分分析(PCA)を活用してマハラノビス距離を計算します。PCAを活用して超楕円を構築し、管理限界を設定してそこからデータが逸脱するのかどうかを判断する手法がMSPCになります。
MSPCでは、PCAを活用して各主成分の分散を1に規格化し、マハラノビス距離を算出する際に全ての主成分を分散1に規格化しません。主要な主成分のみを分散1に規格化して、原点からの距離を算出します。この距離の2乗は$Hotelling'sT^2$統計量と呼ばれております。一方、主要ではない主成分は、Q統計量(二乗予測誤差:Squared Prediction Error)と呼ばれ、主要な主成分で張られる部分空間からの距離(予測誤差)の二乗で定義されます。
MSPCのまとめ
MSPCの手順を下記に整理します。
- 正常データを取得
- 正常データをPCAを活用する
- 主要な主成分の数を決める
- 主要な主成分は$T^2$統計量を算出し、その他の主成分は$Q$統計量を算出する。
- $T^2$統計量、$Q$統計量の管理限界を設定する
MSPCの実装
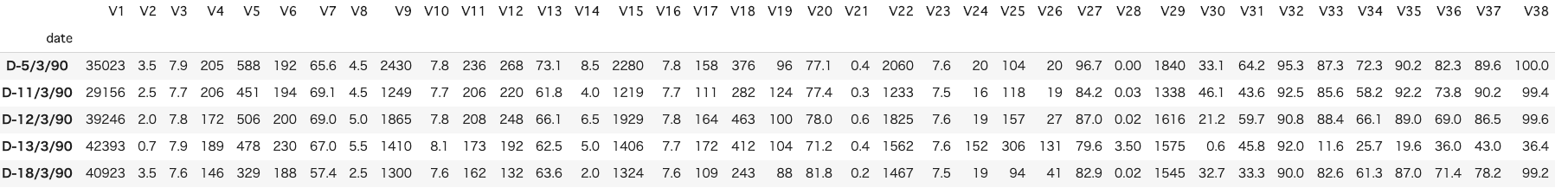
今回、サンプル用にUCI機械学習リポジトリのWater Treatment Plantというデータセットを使用しました。
正常データを100件、残りの279件を評価データとして正常状態からどれだけ逸脱しているのかを見ました。
pythonのコードは下記の通りです。
# 必要なライブラリーのインポート
import pandas as pd
import numpy as np
from matplotlib import pyplot as plt
%matplotlib inline
from sklearn.preprocessing import StandardScaler
from scipy import stats
from sklearn.neighbors.kde import KernelDensity
from sklearn.decomposition import PCA
df = pd.read_csv('watertreatment_mod.csv', encoding='shift_jis', header=0, index_col=0)
df.head()
インプットデータは上記のような感じです。
次に学習データと評価データを分割し、標準化を行っていきます。
# 分割ポイント
split_point = 100
# 学習データ(train)と評価データ(test)に分割
train_df = df.iloc[:(split_point-1),]
test_df = df.iloc[split_point:,]
# データを標準化
sc = StandardScaler()
sc.fit(train_df)
train_df_std = sc.transform(train_df)
次に主成分分析を行って主要な主成分を決めていきます。
今回は累積寄与率が95%までの部分を主要な主成分としました。
# PCA
pca = PCA()
pca.fit(train_df_std)
# 累積寄与率のグラフ
plt.figure()
variance = pca.explained_variance_ratio_
variance_total = np.zeros(np.shape(variance)[0])
plt.bar(range(np.shape(variance)[0]), variance)
for i in range(np.shape(variance)[0]):
variance_total[i] = np.sum(variance[0:i+1])
plt.plot(variance_total)
NumOfScore = np.min(np.where(variance_total>0.95))
x1=[NumOfScore,NumOfScore]
y1=[0,1]
plt.plot(x1,y1,ls="--", color = "r")
pca = PCA(n_components = NumOfScore)
pca.fit(train_df_std)
次にQ統計量を求めます。
# Q統計量
scores = pca.transform(train_df_std)
residuals = pca.inverse_transform(scores)-train_df_std
dist = np.sqrt(np.sum(np.power(residuals,2),axis=1))/(np.shape(train_df_std)[1])
正常データから管理限界を設定しなければいけないため、カーネル密度推定を用いて管理限界を設定しました。
# コントロールリミット(カーネル密度推定)
cr = 0.99
X = dist.reshape(np.shape(dist)[0],1)
bw= (np.max(X)-np.min(X))/100
kde = KernelDensity(kernel='gaussian', bandwidth=bw).fit(X)
X_plot = np.linspace(np.min(X), np.max(X), 1000)[:, np.newaxis]
log_dens = kde.score_samples(X_plot)
plt.figure()
plt.hist(X, bins=X_plot[:,0])
plt.plot(X_plot[:,0],np.exp(log_dens))
prob = np.exp(log_dens) / np.sum(np.exp(log_dens))
calprob = np.zeros(np.shape(prob)[0])
calprob[0] = prob[0]
for i in range(1,np.shape(prob)[0]):
calprob[i]=calprob[i-1]+prob[i]
cl = X_plot[np.min(np.where(calprob>cr))]
次にテストデータが正常データの領域からいつだつしていないのかを確認します。
# データを標準化
test_df_std = sc.transform(test_df)
# Test Data
newscores = pca.transform(test_df_std)
newresiduals = pca.inverse_transform(newscores)-test_df_std
newdist = np.sqrt(np.sum(np.power(newresiduals,2),axis=1))/(np.shape(test_df_std)[1])
最後にQ統計量(学習とテストデータ含む)とカーネル密度推定で算出した管理限界を設定します。
# Q統計量のプロット
SPE = np.r_[dist,newdist]
plt.figure()
x = range(0,np.shape(df.index)[0],100)
NewTimeIndices = np.array(df.index[x])
x2 = [0, np.shape(SPE)[0]]
y2 = [cl,cl]
plt.title('SPE')
plt.plot(SPE)
plt.xticks(x,NewTimeIndices,rotation='vertical')
plt.plot(x2,y2,ls="-", color = "r")
plt.xlabel("Time")
plt.ylabel("SPE")
# contribution plot
plt.figure()
total_residuals= np.r_[residuals,newresiduals]
CspeTimeseries = np.power(total_residuals,2)
cspe_summary=np.zeros([np.shape(CspeTimeseries)[0],10])
本当はここから寄与プロットを算出し、管理限界を超えた際にどの説明変数が影響して異常になったのかを算出します。
また、$T^2$管理図も同様に管理し、管理限界を逸脱した際にどの説明変数が影響しているのか確認することができます。
MSPCを用いることによって、38変数あるセンサーデータも2つの管理図で監視する事ができ、アラームが鳴った際にどのセンサーが異常なのかを特定することができますので、製造現場での監視負荷も下がります。
また、変数同士の関係性が線形でないなら、PCAの代わりにカーネルPCAを活用することも可能です。
スクリプトの纏め
今までのスクリプトを整理します。
# 必要なライブラリーのインポート
import pandas as pd
import numpy as np
from matplotlib import pyplot as plt
%matplotlib inline
from sklearn.preprocessing import StandardScaler
from scipy import stats
from sklearn.neighbors.kde import KernelDensity
from sklearn.decomposition import PCA
def preprocess_sc(df, split_point):
"""データを学習データと評価データに分割し、標準化を行う
Parameters
----------
df (pd.DataFrame) : データセット
split_point (float) : データの分割ポイント(今回は恣意的に決定するため必要)
Returns
-------
train_df_std (pd.DataFrame) : 標準化後の学習データ
test_df_std (pd.DataFrame) : 標準化後の評価データ
"""
# 学習データ(train)と評価データ(test)に分割
train_df = df.iloc[:(split_point-1),]
test_df = df.iloc[split_point:,]
# データを標準化
sc = StandardScaler()
sc.fit(train_df)
train_df_std = sc.transform(train_df)
test_df_std = sc.transform(test_df)
return train_df_std, test_df_std
def pca_q(train_df_std, test_df_std):
"""主成分分析を行いQ統計量を求める
Parameters
----------
train_df_std (pd.DataFrame) : 標準化後の学習データ
test_df_std (pd.DataFrame) : 標準化後の評価データ
Returns
-------
dist (np.array) : 学習データのQ統計量
newdist (np.array) : 評価データのQ統計量
"""
# PCA
pca = PCA()
pca.fit(train_df_std)
# 累積寄与率の計算と可視化
plt.figure()
variance = pca.explained_variance_ratio_
variance_total = np.zeros(np.shape(variance)[0])
plt.bar(range(np.shape(variance)[0]), variance)
for i in range(np.shape(variance)[0]):
variance_total[i] = np.sum(variance[0:i+1])
plt.plot(variance_total)
NumOfScore = np.min(np.where(variance_total>0.95))
x1=[NumOfScore,NumOfScore]
y1=[0,1]
plt.plot(x1,y1,ls="--", color = "r")
pca = PCA(n_components = NumOfScore)
pca.fit(train_df_std)
# Q統計量
scores = pca.transform(train_df_std)
residuals = pca.inverse_transform(scores)-train_df_std
dist = np.sqrt(np.sum(np.power(residuals,2),axis=1))/(np.shape(train_df_std)[1])
# Test Data
newscores = pca.transform(test_df_std)
newresiduals = pca.inverse_transform(newscores)-test_df_std
newdist = np.sqrt(np.sum(np.power(newresiduals,2),axis=1))/(np.shape(test_df_std)[1])
return dist, newdist
def cl_limit(dist, cr=0.99):
"""管理限界の計算を行う
Parameters
----------
dist (np.array) : Q統計量
cr (float) : 管理限界の境界
Returns
-------
cl (float) : 管理限界の境界点
"""
cr = 0.99
X = dist.reshape(np.shape(dist)[0],1)
bw= (np.max(X)-np.min(X))/100
kde = KernelDensity(kernel='gaussian', bandwidth=bw).fit(X)
X_plot = np.linspace(np.min(X), np.max(X), 1000)[:, np.newaxis]
log_dens = kde.score_samples(X_plot)
plt.figure()
plt.hist(X, bins=X_plot[:,0])
plt.plot(X_plot[:,0],np.exp(log_dens))
prob = np.exp(log_dens) / np.sum(np.exp(log_dens))
calprob = np.zeros(np.shape(prob)[0])
calprob[0] = prob[0]
for i in range(1,np.shape(prob)[0]):
calprob[i]=calprob[i-1]+prob[i]
cl = X_plot[np.min(np.where(calprob>cr))]
return cl
def spe_plot(df, dist, newdist, cl):
"""Q統計量を可視化する
Parameters
----------
df (pd.DataFrame) : データセット
dist (np.array) : 学習データのQ統計量
newdist (np.array) : 評価データのQ統計量
cl (float) : 管理限界の境界点
Returns
-------
train_df_std (pd.DataFrame) : 標準化後の学習データ
test_df_std (pd.DataFrame) : 標準化後の評価データ
"""
# Q統計量のプロット
SPE = np.r_[dist,newdist]
plt.figure()
x = range(0,np.shape(df.index)[0],100)
NewTimeIndices = np.array(df.index[x])
x2 = [0, np.shape(SPE)[0]]
y2 = [cl,cl]
plt.title('SPE')
plt.plot(SPE)
plt.xticks(x,NewTimeIndices,rotation='vertical')
plt.plot(x2, y2, ls="-", color = "r")
plt.xlabel("Time")
plt.ylabel("SPE")
# contribution plot
plt.figure()
total_residuals= np.r_[residuals,newresiduals]
CspeTimeseries = np.power(total_residuals,2)
cspe_summary=np.zeros([np.shape(CspeTimeseries)[0],10])
def main():
# データセットの読込み
df = pd.read_csv('watertreatment_mod.csv', encoding='shift_jis', header=0, index_col=0)
train_df_std, test_df_std = preprocess_sc(df, split_point)
dist, newdist = pca_q(train_df_std, test_df_std)
cl = cl_limit(dist, cr=0.99)
spe_plot(df, dist, newdist, cl)
if __name__ == "__main__":
main()
さいごに
最後まで読んで頂き、ありがとうございました。
今回は、製造現場で必要とされる異常検知手法(MSPC)について実装しました。
実際の現場では、正常状態の既定方法や管理限界の設定などチューニング要素は多数ありますが、現場で運用しながら設定していくのが良いと思います。
訂正要望がありましたら、ご連絡頂けますと幸いです。