#はじめに
*インフォマティクス初学者がRDKit環境を構築するまでの流れの記録です。
初めまして。
メーカーで材料の研究開発に従事している技術者です。
最近はインフォマティクス系にも関わり、試行錯誤している最中です。
先週ケモインフォマティクス勉強会に参加し、興味が沸いたのでRDKit環境の構築を行いました。
RDKitについては下記参考にさせていただきました。[1]
(詳細記載されておりますので、RDKitの説明については割愛いたします。)
[1] https://github.com/Mishima-syk/py4chemoinformatics
しかしながら、初学者である私は環境構築に非常に苦戦しました。。。
そこで備忘録として記載します。
(初学者のため、誤りがあれば御指摘いただけると幸いです。)
#実施環境
・MacBook Pro (Retina, 13-inch, Early 2015)
・macOS Catalina 10.15.4
#RDKit環境構築
##1. JupyterLab環境の構築
インフォマティクスに取組む際、下記参考にさせていただきました。[2]
そのときからJupyterLabを利用しています。
JupyterLab環境についての全体的な説明については割愛いたします。
[2] http://datawokagaku.com/startjupyternote/
##2. RDKitのインストール @JupyterLab
JupyterLabを起動し、RDKitのインストールを実施します。
まず、仮想環境の構築です。
conda create -y -n py4chemoinformatics
と入力します。
py4chemoinformaticsは他の名称でも問題ないです。
conda create -n py4chemoinformaticsだけの入力では出力時に"Proceed ([y]/n)?"と確認されるのでyで続行する必要があります。
-yの追記により確認動作を省略し、仮想環境構築を進めることができます。
出力画面にて
"Note: you may need to restart the kernel to use updated packages."
と表示されますが、
Kernelタブ内のRestart Kernelを選択すればOKです。
次にRDKitをインストールします。
conda install -c conda-forge rdkit
と入力し実行すると、インストールが完了します。
この時に以下のようなエラーが発生しました。
"ImportError: libXrender.so.1: cannot open shared object file: No such file or directory"
これはlibXrenderがインストールされていないことによるものだそう。
そこで、
!sudo apt-get install -y libxrender1
と入力し実行することでlibXrenderをインストールすることができました。
さらに進めてみると以下のようなエラーが発生しました。
"ImportError: libXext.so.6: cannot open shared object file: No such file or directory"
これはlibXextがインストールされていないことによるものだそう。
そこで、先ほどの要領で、
!sudo apt-get install -y libxext-dev
と入力し実行することでlibXextをインストールすることができました。
上記に関しては下記参考にさせていただきました。[3]
[3] https://omohikane.com/python3_open_cv_libsm/
##3. RDKitによる構造式描画
RDKitを実際に使ってみます。
今回は基本的な構造式描画を行いました。
from rdkit import Chem
from rdkit import IPythonConsole #構造描画の設定
from rdkit.Chem import Draw
from rdkit import IPythonConsoleにて構造描画の設定をしているようです。
構造を入力するために今回はSMILES記法を利用しました。
*SMILES:Simplified Molecular Input Line Entry System
構造情報を文字列に変換した記述方法
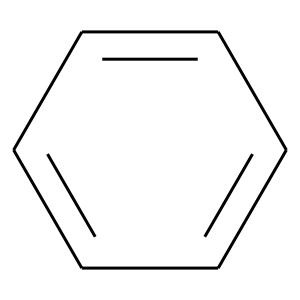
では実際にフェノールの構造式を出力してみます。
mol = Chem.MolFromSmiles("c1ccccc1")
mol
実行した出力結果がこちらです。
何と簡単に描画できることでしょう!
いつも市販ソフトウェアを利用して描画していますが、フリーで描画できてしまうことに感動を覚えました。
下記サイトでは様々な分子構造の記載方法が紹介されています。[4]
[4] https://magattaca.github.io/RDKit_unofficial_translation_JP/Supplementary_Text_jp.html
#終わりに
RDKitのインストール方法について記載いたしました。
環境構築に非常に手間取りましたが(今でも環境面で苦戦すること多数です。。。)、先人の知見の蓄積を参考にさせていただきながら、挑戦していければと思います。
備忘録としてまた記載していきます。
#参考リスト
[1] RDKit全般について
https://github.com/Mishima-syk/py4chemoinformatics
[2] JupyterLabを使う
http://datawokagaku.com/startjupyternote/
[3] ImportErrorの対処法
https://omohikane.com/python3_open_cv_libsm/
[4] PythonでRDKitを始めよう
https://magattaca.github.io/RDKit_unofficial_translation_JP/Supplementary_Text_jp.html