はじめに
Aidemy Premiumのカリキュラムの一環で、受講修了条件を満たすために公開しています。
2024年4月からAidemy Premiumにてデータ分析講座を受講しました。
その成果物として、与えられた患者データから薬剤を選択する予測モデルを構築してみました。
このページはデータ確認編です。(モデル構築・予測編はこちら)
この記事でわかる・できること
- 教師あり学習(分類)の流れがわかる
この記事の対象者
- データ分析初心者
動作環境・データセット
- OS バージョン
- Windows10 Pro 22H2
- 実装環境
- google colaboratory
- データセット
- 【kaggle】Drug.csv
データセット内容
今回のデータセットは、患者情報(年齢、性別、血圧、コレステロール値、ナトリウム、カリウムなど)と、医師が処方した薬剤から構成されています。内容は以下の通りです。
| index | Age | Sex | BP | Cholesterol | Na | K | Drug |
|---|---|---|---|---|---|---|---|
| 0 | 23 | F | HIGH | HIGH | 0.792535 | 0.031258 | drugY |
| 1 | 47 | M | LOW | HIGH | 0.739309 | 0.056468 | drugC |
| 2 | 47 | M | LOW | HIGH | 0.697269 | 0.068944 | drugC |
| 3 | 28 | F | NORMAL | HIGH | 0.563682 | 0.072289 | drugX |
| 4 | 61 | F | LOW | HIGH | 0.559294 | 0.030998 | drugY |
データの確認
データの概要
#Pandasのimport
import pandas as pd
#データの読み込み
df = pd.read_csv("Drug.csv")
# データの形を表示
print(f'Df_shape : {df.shape}\n')
# 各columnのデータ型を表示
print(f'{df.dtypes} \n')
#先頭5つを可視化
display(df.head())
#数値データの統計量を確認
display(df.describe())
#カテゴリカルデータの統計量を確認
display(df.describe(exclude='number'))
# データの形を表示
Df_shape : (200, 7)
# 各columnのデータ型を表示
Age int64
Sex object
BP object
Cholesterol object
Na float64
K float64
Drug object
dtype: object
#先頭5つを可視化
Age Sex BP Cholesterol Na K Drug
0 23 F HIGH HIGH 0.792535 0.031258 drugY
1 47 M LOW HIGH 0.739309 0.056468 drugC
2 47 M LOW HIGH 0.697269 0.068944 drugC
3 28 F NORMAL HIGH 0.563682 0.072289 drugX
4 61 F LOW HIGH 0.559294 0.030998 drugY
#数値データの統計量を確認
Age Na K
count 200.000000 200.000000 200.000000
mean 44.315000 0.697095 0.050174
std 16.544315 0.118907 0.017611
min 15.000000 0.500169 0.020022
25% 31.000000 0.583887 0.035054
50% 45.000000 0.721853 0.049663
75% 58.000000 0.801494 0.066000
max 74.000000 0.896056 0.079788
#カテゴリカルデータの統計量を確認
Sex BP Cholesterol Drug
count 200 200 200 200
unique 2 3 2 5
top M HIGH HIGH drugY
freq 104 77 103 91
- count項目から欠損値なし
- Drug項目から、unique:5に対しtop:drugYの割合が大きい
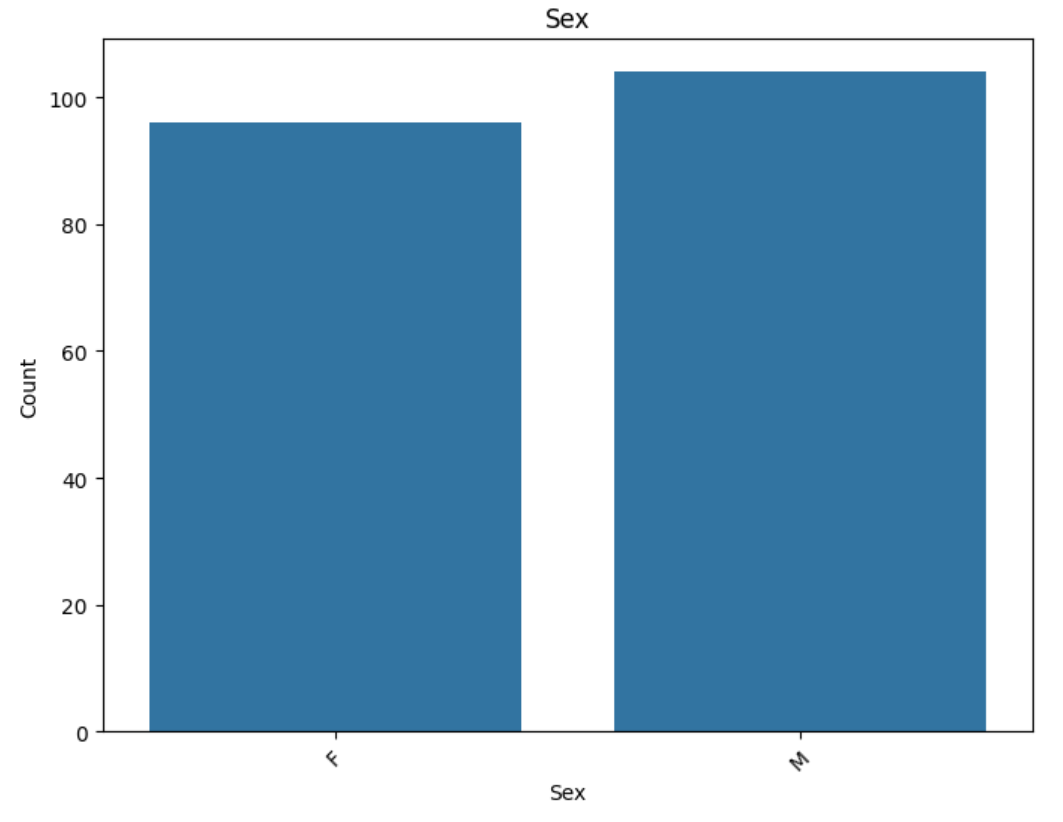
- Sex項目から、男女比は約半々
データ分布の可視化
#各カラムごとに度数分布表を作成する関数定義
def print_frequency_table(column):
print("\nFeature:", column)
print("Frequency table:")
frequency_table = df[column].value_counts()
total_samples = len(df)
for category, count in frequency_table.items():
percentage = (count / total_samples) * 100
print(f"{category}: Count={count}, Percentage={percentage:.2f}%")
# カテゴリカルデータにおける度数分布表を表示させる
print("\nData type and frequency table for qualitative features:")
for column in df.select_dtypes(include=['object']):
print_frequency_table(column)
# カテゴリカルデータのユニーク値を表示
print("\nUnique values in categorical columns:")
for column in df.select_dtypes(include=['object']):
print(f"{column}: {df[column].unique()}")
Data type and frequency table for qualitative features:
Feature: Sex
Frequency table:
M: Count=104, Percentage=52.00%
F: Count=96, Percentage=48.00%
Feature: BP
Frequency table:
HIGH: Count=77, Percentage=38.50%
LOW: Count=64, Percentage=32.00%
NORMAL: Count=59, Percentage=29.50%
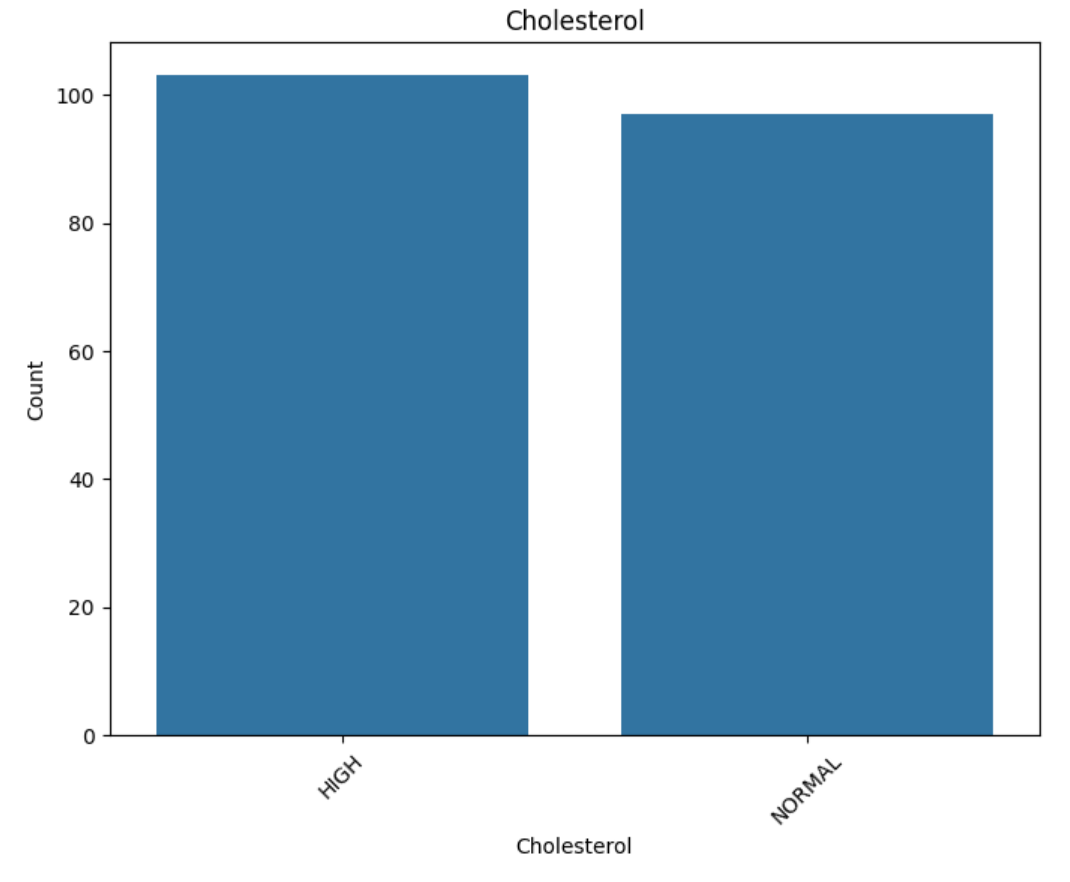
Feature: Cholesterol
Frequency table:
HIGH: Count=103, Percentage=51.50%
NORMAL: Count=97, Percentage=48.50%
Feature: Drug
Frequency table:
drugY: Count=91, Percentage=45.50%
drugX: Count=54, Percentage=27.00%
drugA: Count=23, Percentage=11.50%
drugC: Count=16, Percentage=8.00%
drugB: Count=16, Percentage=8.00%
Unique values in categorical columns:
Sex: ['F' 'M']
BP: ['HIGH' 'LOW' 'NORMAL']
Cholesterol: ['HIGH' 'NORMAL']
Drug: ['drugY' 'drugC' 'drugX' 'drugA' 'drugB']
わかりにくいのでグラフで可視化することに
#グラフによるカテゴリカルデータの可視化
import seaborn as sns
def plot_feature(feature):
if df[feature].dtype == 'object': # Categorical feature
plt.figure(figsize=(8, 6))
sns.countplot(x=feature, data=df)
plt.title(f'{feature}')
plt.xlabel(feature)
plt.ylabel('Count')
plt.xticks(rotation=45)
for feature in df.columns[:]:
plot_feature(feature)
グラフから
- Sex,BP,Cholesterolはユニーク値ごとに大きな偏りはなさそう
- ターゲット変数(Drug)はdrugYが極端に多く、drugA,B,Cは少なめ
- DrugはAge,K,Naに影響されそう
# `Age`のボックスプロット作成
df.boxplot(column=['Age'], figsize=(8, 6))
plt.title('Box Plot of Age')
plt.ylabel('Value')
plt.xlabel('Feature')
plt.show()
# 'K'のヒストグラム作成
plt.figure(figsize=(8, 6))
plt.hist(df['K'], bins=20, color='lightgreen', edgecolor='black')
plt.title('Histogram of Potassium (K)')
plt.xlabel('Potassium (K)')
plt.ylabel('Frequency')
plt.grid(True)
plt.show()
# 'Na'のヒストグラム作成
plt.figure(figsize=(8, 6))
plt.hist(df['Na'], bins=20, color='skyblue', edgecolor='black')
plt.title('Histogram of Sodium (Na)')
plt.xlabel('Sodium (Na)')
plt.ylabel('Frequency')
plt.grid(True)
plt.show()
グラフから
- Ageは15~74歳で大きなばらつきはなさそう
- Na,Kは全体的にやや高めか
データ相関の可視化
# カテゴリカルデータとDrugの相関図を作成する関数定義
def plot_feature_vs_target(feature):
if df[feature].dtype == 'object':
plt.figure(figsize=(8, 6))
sns.countplot(x=feature, hue='Drug', data=df)
plt.title(f'{feature} vs Drug')
plt.xlabel(feature)
plt.ylabel('Count')
plt.legend(title='Drug', loc='upper right')
plt.xticks(rotation=45)
# %をグラフ上に表示させる
total_count_all = df.shape[0]
for drug in df['Drug'].unique():
total_count = df[df['Drug'] == drug].shape[0]
for i, p in enumerate(plt.gca().patches):
if p.get_height() > 0:
if i % len(df['Drug'].unique()) == df['Drug'].unique().tolist().index(drug):
plt.text(p.get_x() + p.get_width() / 2.,
p.get_height() + 0.5,
f'{p.get_height() / total_count_all * 100:.1f}%',
ha='center', va='bottom')
# 各カラムごとに反復処理
for feature in df.columns[:-1]:
plot_feature_vs_target(feature)
グラフから
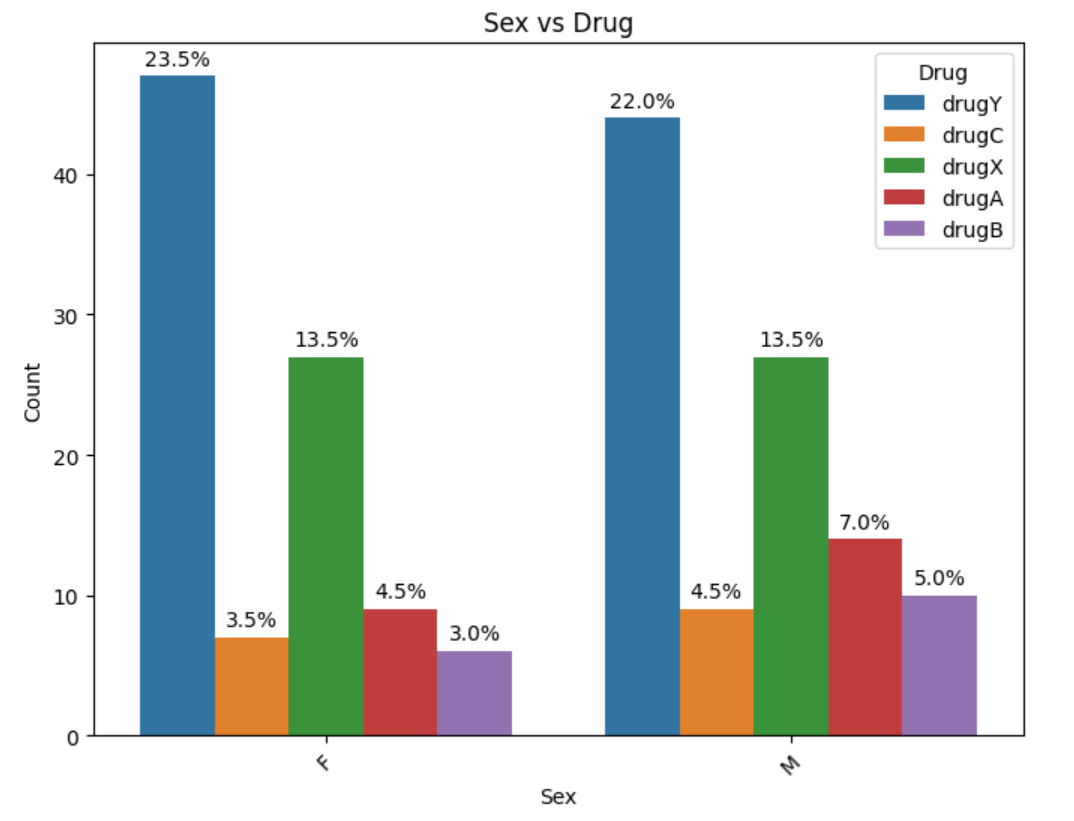
- Sex vs Drugから、女性の方がややdrugA~C高めか
- BP vs Drugから、BPのユニーク値ごとにDrug分布が大きく異なり、DrugはBPの影響を強く受けそう
- Cholesterol vs Drugから、BPほどではないがユニーク値ごとに分布が異なる
from scipy.stats import chi2_contingency
# DrugとSexのクロス集計表を作成
contingency_table = pd.crosstab(df['Drug'], df['Sex'])
# カイ二乗検定を実行し、カイ二乗値、p値、自由度、期待度数を取得
chi2, p, dof, expected = chi2_contingency(contingency_table)
print("カイ二乗値:", chi2)
print("p値:", p)
print("自由度:", dof)
print("期待度数:", expected)
print("Contingency Table:")
display(contingency_table)
# 期待度数の最小値が5未満の場合の警告を表示
expected_min_count = expected.min().min()
if expected_min_count < 5:
print("WARNING: Expected frequencies in the contingency table are below 5. Consider merging categories or using another test.")
# データ数が40未満の場合の警告を表示
sample_size = len(df)
if sample_size < 40:
print("WARNING: Sample size is below 40. Consider increasing sample size for more reliable results.")
カイ二乗分布表より、発生確率0.05未満のカイ二乗値は9.4877
今回算出されたカイ二乗値は2.119248418109203であり、9.4877よりも低い値
よって「2つの指標は互いに独立している」という帰無仮説は棄却されず、
DrugとSexは互いに独立である

カイ二乗検定の超基本を確率分布から考える。| econoshift.com
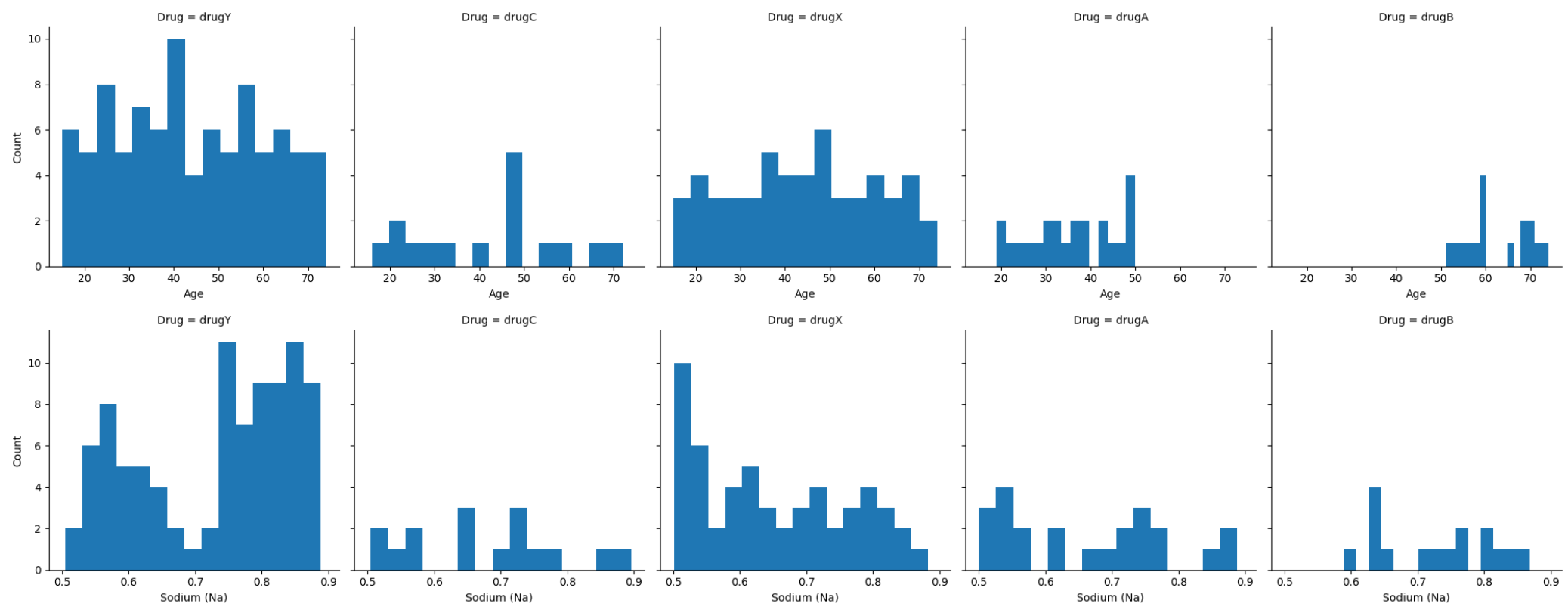
# AgeのヒストグラムをFacetGridにマップする
g = sns.FacetGrid(df, col='Drug', height=4)
g.map(plt.hist, 'Age', bins=15)
g.set_axis_labels('Age', 'Count')
g.set_titles('Drug = {col_name}')
plt.show()
# NaのヒストグラムをFacetGridにマップする
g = sns.FacetGrid(df, col='Drug', height=4)
g.map(plt.hist, 'Na', bins=15)
g.set_axis_labels('Sodium (Na)', 'Count')
g.set_titles('Drug = {col_name}')
plt.show()
# KのヒストグラムをFacetGridにマップする
g = sns.FacetGrid(df, col='Drug', height=4)
g.map(plt.hist, 'K', bins=15)
g.set_axis_labels('Potassium (K)', 'Count')
g.set_titles('Drug = {col_name}')
plt.show()
from sklearn.preprocessing import LabelEncoder
# DrugをLabelEncoder()を使用して数値に変換
label_encoder = LabelEncoder()
df['Drug'] = label_encoder.fit_transform(df['Drug'])
# 数値データ
numerical_features = ['Age', 'Na', 'K']
# 数値データのピアソン相関係数を計算。
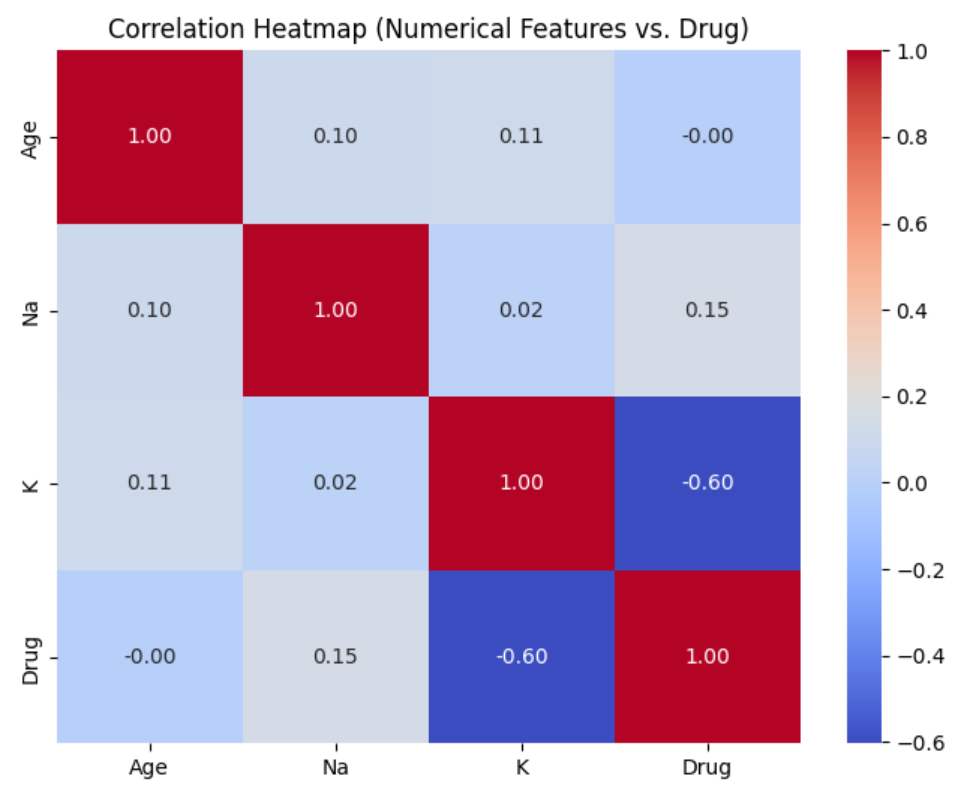
correlation_matrix = df[numerical_features + ['Drug']].corr()
# ヒートマップを使って相関関係を視覚化
plt.figure(figsize=(8, 6))
sns.heatmap(correlation_matrix, annot=True, cmap='coolwarm', fmt=".2f")
plt.title('Correlation Heatmap (Numerical Features vs. Drug)')
plt.show()
DrugとKの間に負の相関があることを確認
from scipy.stats import pearsonr, spearmanr
# すべての数値特徴のペアについて、ピアソンおよびスピアマンの相関係数とp値を計算
correlation_results = {}
for i, feature1 in enumerate(numerical_features):
for j, feature2 in enumerate(numerical_features):
if i < j: # 重複するペアを避ける
# ピアソンの積立相関係数とp値を計算
correlation_coefficient_pearson, p_value_pearson = pearsonr(df[feature1], df[feature2])
# スピアマンの順位相関係数とp値を計算
correlation_coefficient_spearman, p_value_spearman = spearmanr(df[feature1], df[feature2])
correlation_results[(feature1, feature2)] = {
'Pearson': (correlation_coefficient_pearson, p_value_pearson),
'Spearman': (correlation_coefficient_spearman, p_value_spearman)
}
# 計算結果を表示する
for (feature1, feature2), results in correlation_results.items():
print(f"Correlation between {feature1} and {feature2}:")
pearson_coefficient, pearson_p_value = results['Pearson']
spearman_coefficient, spearman_p_value = results['Spearman']
print("Pearson correlation:")
print(f"- Coefficient: {pearson_coefficient:.2f}")
print(f"- P-value: {pearson_p_value:.4f}")
if pearson_p_value < 0.05:
print(" (Statistically significant)")
else:
print(" (Not statistically significant)")
print("Spearman rank correlation:")
print(f"- Coefficient: {spearman_coefficient:.2f}")
print(f"- P-value: {spearman_p_value:.4f}")
if spearman_p_value < 0.05:
print(" (Statistically significant)")
else:
print(" (Not statistically significant)")
print()
Correlation between Age and Na:
Pearson correlation:
- Coefficient: 0.10
- P-value: 0.1573
(Not statistically significant)
Spearman rank correlation:
- Coefficient: 0.09
- P-value: 0.1900
(Not statistically significant)
Correlation between Age and K:
Pearson correlation:
- Coefficient: 0.11
- P-value: 0.1160
(Not statistically significant)
Spearman rank correlation:
- Coefficient: 0.11
- P-value: 0.1230
(Not statistically significant)
Correlation between Na and K:
Pearson correlation:
- Coefficient: 0.02
- P-value: 0.8075
(Not statistically significant)
Spearman rank correlation:
- Coefficient: -0.01
- P-value: 0.9417
(Not statistically significant)
Age,Na,Kの間には相関はない
plt.figure(figsize=(8, 6))
sns.regplot(x='Na', y='K', data=df, scatter_kws={'alpha':0.5})
plt.title('Scatter Plot of Na vs K')
plt.xlabel('Sodium (Na)')
plt.ylabel('Potassium (K)')
plt.grid(True)
plt.show()
class_names_full = {
0: "drugA",
1: "drugB",
2: "drugC",
3: "drugX",
4: "drugY"
}
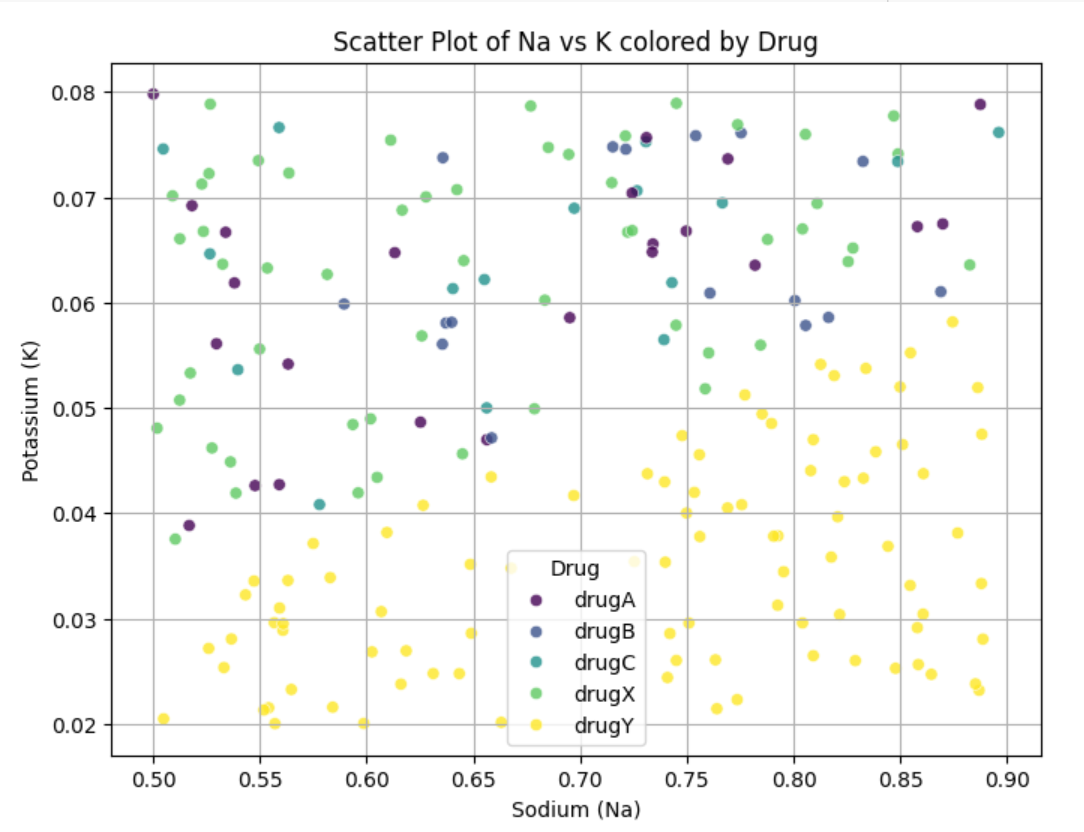
plt.figure(figsize=(8, 6))
scatter = sns.scatterplot(x='Na', y='K', hue='Drug', data=df, palette='viridis', alpha=0.8)
plt.title('Scatter Plot of Na vs K colored by Drug')
plt.xlabel('Sodium (Na)')
plt.ylabel('Potassium (K)')
plt.grid(True)
# Update legend labels
handles, labels = scatter.get_legend_handles_labels()
scatter.legend(handles, [class_names_full[int(label)] for label in labels], title='Drug')
plt.show()
Na,KとdrugYとの関係には相関がありそう(Na/K比に注目)
新たな特徴量の作成と可視化
# 各データについてNa/K比を計算し、新しい列 Na/K Ratio に追加
df['Na/K Ratio'] = df['Na'] / df['K']
# Drugのカラム名を数値から元に戻す
df['Drug'] = df['Drug'].map(class_names_full)
# Na/K 比率の分布を視覚化
plt.figure(figsize=(8, 6))
sns.boxplot(x='Drug', y='Na/K Ratio', data=df, color='skyblue')
plt.title('Na/K Ratio by Drug')
plt.xlabel('Drug')
plt.ylabel('Na/K Ratio')
plt.show()
# drugYとその他のdrug間で Na/K比の平均に有意差があるかを検定(t検定)
from scipy.stats import ttest_ind
drugY_ratio = df[df['Drug'] == 'drugY']['Na/K Ratio']
other_drugs_ratio = df[df['Drug'] != 'drugY']['Na/K Ratio']
# 有意水準
alpha = 0.05
t_statistic, p_value = ttest_ind(drugY_ratio, other_drugs_ratio)
print(f'T-test: t-statistic = {t_statistic}, p-value = {p_value}')
# 判定
if p_value < alpha:
print("Statistically significant")#有意差あり
else:
print("Not statistically significant")#有意差なし
drugYとその他のdrug間で Na/K比の平均に有意差あり
import numpy as np
import warnings
# FutureWarning を無効化(新しいバージョンで発生する可能のある警告を無視)
warnings.simplefilter(action='ignore', category=FutureWarning)
# Drug列がdrugYの行のみをフィルタリングし、新しいDataFrame`drugY_df`に保存
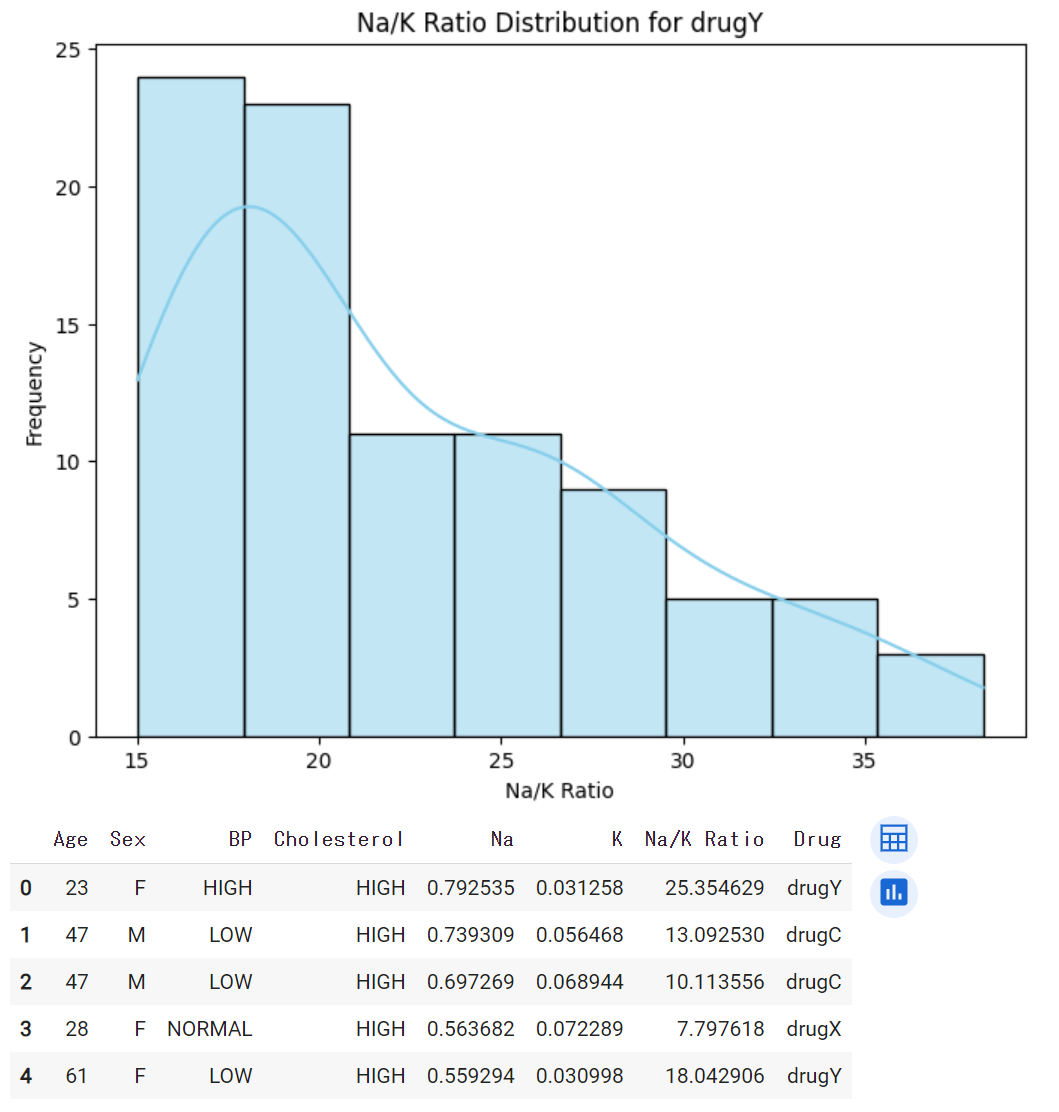
drugY_df = df[df['Drug'] == 'drugY'].copy() # 元のDataFrameを変更しないようにコピーを作成
# drugYのNa/K比の分布を可視化
plt.figure(figsize=(8, 6))
sns.histplot(drugY_df['Na/K Ratio'], kde=True, color='skyblue')
plt.title('Na/K Ratio Distribution for drugY')
plt.xlabel('Na/K Ratio')
plt.ylabel('Frequency')
plt.show()
# Drug列を最終行に移動
drug_column = df.pop('Drug')#Drug列を切り取り
df['Drug'] = drug_column#新たな列として追加
# 先頭5行を表示
display(df.head())
数値データのカテゴリカル変数化
from sklearn.preprocessing import OrdinalEncoder, LabelEncoder
# カテゴリカルデータとそのクラスの定義
sex_classes = ['F', 'M']
bp_classes = ['LOW', 'NORMAL', 'HIGH']
cholesterol_classes = ['NORMAL', 'HIGH']
drug_classes = ['drugA', 'drugB', 'drugC', 'drugX', 'drugY']
# Sex,BP,Cholesterolを数値データに変換
ordinal_encoder = OrdinalEncoder(categories=[sex_classes, bp_classes, cholesterol_classes])
df[['Sex', 'BP', 'Cholesterol']] = ordinal_encoder.fit_transform(df[['Sex', 'BP', 'Cholesterol']])
# ターゲット変数Drugを数値に変換
label_encoder = LabelEncoder()
df['Drug'] = label_encoder.fit_transform(df['Drug'])
# DataFrameの確認
display(df.head())
まとめ
- SexとDrugは互いに独立
- BP,Cholestelol,K,Na/K RatioとDrugとの間に相関あり
Sexを削除したデータセットでモデルの学習を行う
(モデル構築・予測編はこちら)