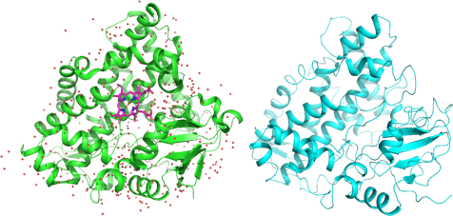RaptorXはタンパク質の構造予測コンテストCASPでAlphaFold以前からずっとこの研究で有名な豊田工業大学シカゴ校のJinbo Xu先生たちが作成しているツール群。
RaptorX-3DModelingは2020年10月12日にJinbo Xu先生らが発表した論文Improved protein structure prediction by deep learning irrespective of co-evolution informationの内容をソフトウェア化したもの。
RaptorXのウェブサイトではこれをウェブサーバー上から実行することができるらしいが、相当時間がかかるみたいなので、彼らの公開しているGitHubからソースコードをダウンロードしてきて実行することができればそちらの方が良いそうだ。
ここでは https://github.com/j3xugit/RaptorX-3DModeling/ からcloneしてきたRaptorX-3DModelingを実行し、その性能を評価することを試みます。
インストール方法
マシン環境
OSは
- GPUつきCentOS 7系または8系
- GPUつきUbuntu 18.04または20.04
- (macOS)
のいずれかが必要。そしてHH-suite動作のための500GB以上のディスク容量が必要。一応macOSでも原理的には動作すると思いますが、ディスク容量的に厳しいと思うのであまりおすすめしません。
HH-Suite 3.3のインストール
事前にHH-Suite 3.3をインストールし、UniRef30_2020_06_hhsuite.tar.gzを解凍して適当なディレクトリに配置しておく。この解凍されたデータベースファイルはなぜか読み込み許可のパーミッションがついていないことが多々あるので注意。
以下の例では/home/database/uniref30_2020_03/にデータベースを解凍しておいたものとします。
CCMPred動作準備
CCMPredはHH-Suiteから生成したMSAからコンタクトマップを生成するためのツール。ただ、すでにGitHubのRaptorX-3DModelingの中に組み込まれているので新たにダウンロードする必要はありません。しかし、動作に必要ないくつかのライブラリがマシン環境によっては入っていないことがあるので、先にこれをインストールしておきます。
# ccmpred: error while loading shared libraries: libncurses.so.5: cannot open shared object file: No such file or directoryのエラー回避
dnf -y install ncurses-compat-libs parallel # CentOS 8の場合
sudo apt -y install ncurses-devel parallel # Ubuntu/Debian系の場合
brew install ncurses parallel # macOSの場合
anacondaを使ってPython2.7環境を用意する
RaptorX-3DModelingは現在まだPython2.7環境依存だそうで、さらにいくつかのPythonモジュールに依存しているため、素直にAnacondaを入れておいた方が楽かもしれないです。ちなみにminicondaではモジュールが足りなかった。AnacondaでPython2.7の仮想環境を構築し、その状態でcondaとpipを使ってbiopython, scipy,numpy, mkl, theano, pygpuなど必要なモジュールをさらにインストールします。
# Linux用のAnacondaをダウンロードする。Mac用は探して入れてきてください。
wget https://repo.anaconda.com/archive/Anaconda2-2019.10-Linux-x86_64.sh
chmod +x Anaconda2-2019.10-Linux-x86_64.sh
./Anaconda2-2019.10-Linux-x86_64.sh
# ここで指示に従ってAnaconda 2のインストールを進める。
# 詳しくは https://www.python.jp/install/anaconda/unix/install.html などを読みましょう。
# 一度ログアウトして再ログインし、conda initが動くかを確かめる
conda init
# conda initが動いたら次に進む
conda config --set auto_activate_base false
conda create --name RaptorX python=2
## conda activateを使って(RaptorX)専用の仮想環境にする
conda activate RaptorX
## RaptorXのPython2.7が起動すると、Terminalの表示が切り替わる。
which python2.7
## 上のwhich python2.7の返り値が~/anaconda2/envs/RaptorX/bin/python2.7となっていることを確認したら次へ進む
## pipとcondaで必要なパッケージを入れる。
## condaの後に-yを付けていると途中の選択肢を自動でyesにしてくれる
pip install biopython==1.76 pillow
conda install numpy scipy mkl -y
conda install theano pygpu -y
conda install -c anaconda msgpack-python -y
## shared_ndarrayのインストール
git clone https://github.com/crowsonkb/shared_ndarray.git
cd shared_ndarray/
python2.7 setup.py install
PyRosetta4のインストール
PyRosetta4はタンパク質デザイン&モデリングソフトウェアRosettaのタンパク質構造生成部分をPythonで行えるようにした派生パッケージ(?)らしい。http://www.pyrosetta.org/dow からダウンロードできるようになっているが、User, Passが求められるのでそれを入力します。……ただ、なぜか最近うまく手続きをこなしてもユーザー名とパスを提示してくれないことがあるみたいなので、一応書いておくと、pose と foldtreeです。
解凍などのコマンドは以下の通り。
tar jxvf PyRosetta4.Release.python27.linux.release-269.tar.bz2
mv PyRosetta4.Release.python27.linux.release-269 PyRosetta4_python27_269
cd PyRosetta4_python27_269/setup
## 必ず先程のcondaが起動している状態でpython2.7 setupを行う
python2.7 setup.py install
CuDNN 7.6.5のインストール
CUDA 10.2.89にNVIDIAのサイトからダウンロードしてきたcudnn 7.6.5をインストールする。cudnn 8では動作しない。
tar zxvf cudnn-10.2-linux-x64-v7.6.5.32.tgz
cp -rp cuda/include/cudnn* /usr/local/cuda-10.2/include/
cp -rp cuda/lib64/* /usr/local/cuda-10.2/lib64/
cp -rp cuda/NVIDIA_SLA_cuDNN_Support.txt /usr/local/cuda-10.2/
RaptorX-3DModeling本体のインストール
例としてホームディレクトリ/home/ag_smith上にインストールすると仮定します。
また、Jinbo先生のウェブサイトから学習済みのモデルをダウンロードし、/home/ag_smith/RaptorX-3DModeling内のDL4DistancePrediction4とDL4PropertyPredictionにそれぞれ配置します。Distanceの方は794MBくらいのファイルサイズになっていますがもう一方は意外と小さいファイルサイズです。これらを解凍して配置します。
cd /home/ag_smith #RaptorX-3DModelingを置きたい任意のディレクトリに移動する
git clone https://github.com/j3xugit/RaptorX-3DModeling.git
# RaptorX-3DModelingのPATH設定ファイルraptorx-external.shを編集してHH-Suiteの場所を指定する。
# この中のHHDIRとHHDBをそれぞれHH-suiteのインストール場所に適切に変更しておく。
# 以下はその例
# export HHDIR=/home/apps/hh-suite/3.3
# export PATH=$HHDIR/bin:$HHDIR/scripts:$PATH
# export HHDB=/home/database/uniref30_2020_03/UniRef30_2020_03
# 学習済みモデルをJinbo Xu先生のウェブサイトからダウンロードし、適切なPATH上に置く
# ModelingHomeを自身のRaptorX-3DModelingがある場所に設定する
export ModelingHome=/home/ag_smith/RaptorX-3DModeling/
cd $ModelingHome/DL4DistancePrediction4
curl http://raptorx.uchicago.edu/download/bW9yaXdha2lAYmkuYS51LXRva3lvLmFjLmpw/39/ -o RXDeepModels4DistOri-FM.tar.gz
tar zxvf RXDeepModels4DistOri-FM.tar.gz
cd $ModelingHome/DL4PropertyPrediction
curl http://raptorx.uchicago.edu/download/bW9yaXdha2lAYmkuYS51LXRva3lvLmFjLmpw/43/ -o RXDeepModels4Property.tar.gz
tar zxvf RXDeepModels4Property.tar.gz
以上で設定は完了です。
モデリングの実行
まず任意のfasta形式ファイルを用意します。例としてPDB: 2O6PのChain Aの配列(の一部)を2o6pA.fastaとして使います。
>2O6P_1|Chains A,B|Iron-regulated surface determinant protein C|Staphylococcus aureus subsp. aureus (158879)
NAADSGTLNYEVYKYNTNDTSIANDYFNKPAKYIKKNGKLYVQITVNHSHWITGMSIEGHKENIISKNTAKDERTSEFEVSKLNGKIDGKIDVYIDEKVNGKPFKYDHHYNITYKFNGPTDVAGAN
次に実行スクリプトを用意します。
# !/bin/sh
# >>> ここからcondaの設定読み込み >>>
# !! Contents within this block are managed by 'conda init' !!
__conda_setup="$('/home/ag_smith/anaconda2/bin/conda' 'shell.bash' 'hook' 2> /dev/null)"
if [ $? -eq 0 ]; then
eval "$__conda_setup"
else
if [ -f "/home/ag_smith/anaconda2/etc/profile.d/conda.sh" ]; then
. "/home/ag_smith/anaconda2/etc/profile.d/conda.sh"
else
export PATH="/home/ag_smith/anaconda2/bin:$PATH"
fi
fi
unset __conda_setup
# <<< ここまでcondaの設定読み込み <<<
# RaptorX仮想環境の起動
conda activate RaptorX
# RaptorX-3DModelingのPATH設定
export ModelingHome=/home/ag_smith/RaptorX-3DModeling
. $ModelingHome/raptorx-path.sh
. $ModelingHome/raptorx-external.sh
# RaptorXFolderの実行
cd $ModelingHome
Server/RaptorXFolder.sh -g -1 \
-l 1050 \
-m 9 \
-n 20 \
-r 1 \
-t 1 \
2o6pA.fasta
condaの設定読み込みについては、お使いのマシン環境に合わせてPATHを変更してください(特に/home/ag_smithは変更必須です)。次にconda activate RaptorXを入力して仮想環境を起動し、RaptorX-3DModelingのPATH設定を読み込みます。
そして最後にServer/RaptorXFolder.shを引数付きで実行することでタンパク質構造予測が始まります。最後の行に、予測したい配列ファイルの名前を指定します(例として2o6pA.fastaを入れています)。
使用方法の詳細についてはRaptorX-3DModelingのディレクトリ内部でServer/RaptorXFolder.shを実行すると表示されます。またはGitHubの解説を読んでください。-nはモデルするタンパク質構造の個数、-rは0ならば構造最適化しない、1ならば構造最適化する、といった具合です。
計算時間
計算時間は入力したアミノ酸配列の長さに依存します。150アミノ酸程度ですと特徴量の生成計算におよそ20〜30分、650アミノ酸と長いものですと4時間くらいです。あとはその特徴量を使って生成するモデルの個数(つまり上記シェルスクリプトの-nの値)に依存しますが、20個程度ですとおよそ30分〜1時間くらいです。
性能評価
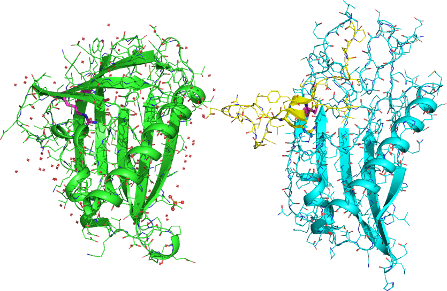
Case 1: IsdC (PDB: 2O6P)
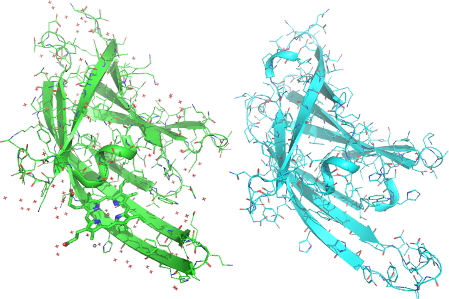
緑が結晶構造、青がRaptorXの作り出した20個のモデル構造のうち最良だったもの。RMSD = 1.9Åと非常に精度が高い。活性部位(ヘム結合部分)もきれいに再現されている。30分程度で計算終了。
ほぼ完璧ですね。
Case 2: LFS (PDB: 5GTE)
タマネギを切った時に飛び出てくる催涙成分PTSOを合成するタンパク質LFS。プレスリリース記事
RMSDは3.6Å程度。触媒活性を持つ残基R71, E88(図左の紫色)が乗っているβシートの構造予測周辺(黄色)では、周辺のアミノ酸との距離情報がどうやらほとんど取得できなかったみたいで、PyRosettaが作り出すモデル構造ごとに位置が大きく変化しており、精度良いモデリングがまったくできていませんでした。図に載せているのは20個作り出したうち、もっとも(運良く)正解に近い位置にいたものを示しています。
Case 3: CYP154C2 (PDB: 6L69)
シトクロムP450の一種のCYP154C2。400アミノ酸長。
シトクロムP450系は自然界に類縁配列が非常に多いことからMSAの取得が容易だったと思われる。さらにヘムの結合部位が常に共通しており、類縁配列も非常に多く存在しているためか、概形およびヘムの結合部位周辺のモデリング精度は高かった。RMSD = 2.53Å。ただ、基質が結合するポケット部位周辺はやはり少し不安定。
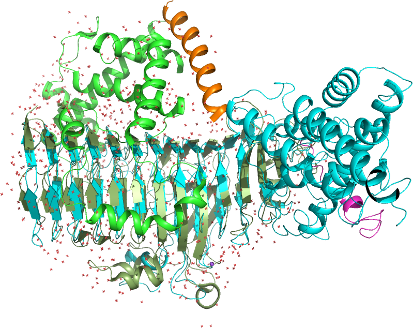
Case 4: GH136 lacto-N-biosidase (PDB: 6KQT)
このタンパク質の全長は2つの構造ドメインに分かれ、αヘリックスが多いN末端ドメイン(残基1-180)とαヘリックスリンカー(残基203-226、オレンジ色)、βシートが何個も並ぶβ helix foldと呼ばれる領域(残基242-663、ErLnb136II)という構成になっています。
C末端側のβ helix foldドメインとても精度よく予測できていますが、N末端側のヘリックスドメインは位置関係が大きくずれている上に、それぞれのヘリックス部分で重ねてもうまく重なりませんでした。ただこれについては、オレンジ色のαヘリックス型リンカーを境に2つの構造ドメインから成り立っているため、それらの位置関係は独立しているため、大きな問題ではないと思います。ただ、このαヘリックスリンカーは予測ではうまくαヘリックスになっていなかった(紫色)上、αヘリックスドメイン自体のトポロジーもうまく結晶構造と一致しなかったことについてはよくわかっていません。
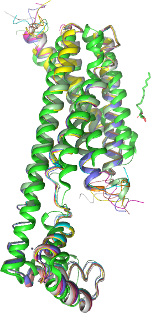
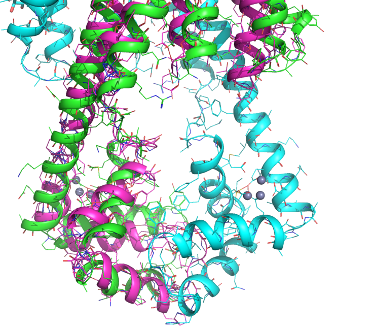
Case 5: Iron transporter VIT1 (PDB: 6IU3)
Iron transporter VIT1 with zinc ions from Saccharomyces cerevisiae。
膜タンパク質かつ2量体で機能するタイプのタンパク質の例として選んできました。234アミノ酸長。
緑と青が2量体を形成する正解の結晶構造で、紫色がRaptorX-3DModelingによるモデル構造です。
概形はこのケースであってもかなり精度良く再現できていると思われますが、肝心のZnイオン(灰色のボール、計6個見える)が結合する部位(下図)を構築するヘリックスの相対的な位置はあと一歩と言ったところでしょうか。
まとめ
タンパク質の概形を30分〜数時間以内で、しかもたった20個しかモデルを出力していないにもかかわらず、かなり精度良く構造を予測してくれることがわかりました。BioRxivの論文によれば、CASP13のターゲットタンパク質だったものについても、このRaptorX-3DModelingはAlphaFoldを上回る精度で構造を出力してくれるとアピールしています。2020年の12月にはCASP14コンテストの結果が発表されると言っておりますが、このRaptorX-3DModelingが優勝するのか、それとも他のグループが出した予測器がこれを上回るのか見どころですね。