ワシントン大学のDavid Baker研究室が中心となっている開発しているRosetta Program Suiteはde novoタンパク質モデリングで目覚ましい成果を上げていますが、それの要となっているのがFragment Assemblyと呼ばれる手法で、既知のタンパク質構造情報から、アミノ酸を3-merまたは9-merごとに分割し(フラグメント)、それらを組み合わせてタンパク質を作り上げるということをしています。Rosettaのプログラムの1つであるAbinitioRelaxなども、このフラグメント情報を活用することで構造空間探索を絞り込むことができ、少ない時間でもっともらしい部分構造を提示することができるようになっています。
Rosettaのfragment pickingなどに使われるフラグメントの元ネタのファイルのことはvallと呼ばれ、通常、ユーザーとしてこのフラグメントを集めたライブラリファイルであるvall.jul19.2011.gzは、Rosettaをインストールしたときに/path/to/Rosetta/tools/fragment_toolsに格納されています。
今回のお話は、このフラグメントファイルを自作してアップデートしようというものです。
動機としては、このvall.jul19.2011.gzを解凍してみればわかるのですが、2011年7月19日に作成したまま、このフラグメントライブラリの更新が止まってしまっているからなのです(なんで止まってるんだろう?)。2011年7月19日から2018年9月30日の間にProtein Data Bank (PDB) に登録されているタンパク質の構造の数は66,889個となっており、この7年間でこのライブラリが作られた当時の約2倍弱までタンパク質構造情報が増えています。このため、最新版に更新することで新しいモチーフなどが採用されるようになれば、Rosettaのモデル構築や、AbinitioRelaxなどのde novo構造予測に置いて、もっと精度の高い結果をもたらすことが期待されます。
Rosetta 3.9から、このフラグメントライブラリの作成を支援してくれるツールpdb2vallがfragment_toolsディレクトリに公開されました(たぶん3.8まではなかったような気がします)。しかしながら、このpdb2vall、処理自体はかなり明快なのですがかなり無茶苦茶な実装をしている上に、全然ユーザーフレンドリーではないので初見殺しの要素が満載です。そこで、これらを解決してフラグメントを作るまでの記録をここに綴ります。
fragment_toolsディレクトリについて
以下の項では上記のようにRosettaフラグメントを作るまでの記録を綴っているのですが、結果としてかなりの部分を補修する必要があったので、この記事を見ている方々がいちいち全部手動で修正していくのはきっと面倒だと思います。
そこで、せっかちな方は https://github.com/BILAB/fragment_tools/ をまるごとダウンロードして使ってください。
git clone https://github.com/BILAB/fragment_tools.git
補修にあたっては@omi_UTさんが多大な貢献をしてくださいました(特に0カラム補修部分)。
使用するときには、下記の「BLASTのnrデータベースの構築」、「pdb_seqres.txt, ss_dis.txtの入手」、「BioPerlのインストール」、「DEPTHのインストール」、「pdb2vall.cfgの設定」、「環境変数設定」が必要となります。
前提:フラグメントデータベースとvallファイルについて
/path/to/Rosetta/tools/fragment_toolsに存在するvallではじまるファイルたちがフラグメントデータベースです。特に、vall.jul19.2011.gz(466540884
byte)がフラグメントデータベースの本体です。2008verもあります。
中身を見てみるとこんな感じ
# VALL - created by David E Kim
# PDB source: Pisces server cullpdb_pc60_res3.0_R0.3_d110719_chains16802.3820
# NR Protein Database 06-19-2011
# PDBs idealized and relaxed with coordinate constraints
#
# pdb code+chain
# AA one-letter aminio acid code
# SS DSSP secondary structure assignment from original PDB coordinates (E,H,L)
# seqpos pdb_seqres.txt position
# bfactor avg N,CA,C bfactor
# CA_x CA x coord
# CA_y CA y coord
# CA_z CA z coord
# CB_x CB x coord
# CB_y CB y coord
# CB_z CB z coord
# CEN_x centroid x coord
# CEN_y centroid y coord
# CEN_z centroid z coord
# Phi phi torsion
# Psi psi torsion
# Omega omega torsion
# dssp_phi dssp phi torsion
# dssp_psi dssp psi torsion
# dssp_sa dssp solvent accessibility
# n_ali number of alignments used for PSSM
# p{AA} Residue type AA frequency from psiblast binary checkpoint file (with pseudocounts)
# ps{AA} Residue type AA frequency from structure profiles (with pseudocounts)
#
#pdb AA SS seqpos bfactor CA_x CA_y CA_z CB_x CB_y CB_z CEN_x CEN_y CEN_z Phi Psi Omega dssp_phi dssp_psi dssp_sa n_ali pA pC pD pE pF pG pH pI pK pL pM pN pP pQ pR pS pT pV pW pY psA psC psD psE psF psG psH psI psK psL psM psN psP psQ psR psS psT psV psW psY
7odcA S L 2 38.15 -27.18 -11.43 -31.31 -28.59 -11.05 -30.93 -29.05 -11.04 -30.64 0.000 133.529 -176.999 360.000 131.300 75 500 0.151 0.005 0.129 0.059 0.008 0.031 0.010 0.012 0.174 0.020 0.007 0.157 0.018 0.042 0.030 0.105 0.033 0.018 0.002 0.009 0.060 0.005 0.045 0.076 0.008 0.259 0.006 0.041 0.048 0.050 0.007 0.018 0.013 0.017 0.096 0.082 0.099 0.068 0.003 0.007
7odcA S E 3 38.43 -24.67 -8.57 -31.13 -23.30 -9.21 -31.11 -22.89 -9.54 -31.26 -156.125 166.203 176.182 -152.900 163.500 74 500 0.051 0.005 0.020 0.049 0.078 0.234 0.036 0.038 0.022 0.040 0.018 0.056 0.015 0.052 0.017 0.120 0.082 0.036 0.004 0.032 0.054 0.004 0.033 0.137 0.051 0.032 0.021 0.041 0.085 0.023 0.018 0.016 0.066 0.018 0.017 0.134 0.175 0.047 0.034 0.010
7odcA F E 4 39.41 -23.86 -5.07 -32.37 -24.95 -4.06 -32.01 -26.36 -3.58 -32.56 -142.134 158.100 -176.901 -140.400 155.800 24 500 0.036 0.008 0.010 0.014 0.333 0.024 0.019 0.033 0.013 0.102 0.012 0.011 0.010 0.011 0.012 0.064 0.020 0.086 0.008 0.144 0.062 0.025 0.008 0.030 0.089 0.034 0.005 0.146 0.011 0.161 0.011 0.007 0.031 0.027 0.009 0.017 0.033 0.202 0.003 0.068
7odcA T E 5 40.59 -20.74 -3.05 -33.14 -19.86 -3.57 -34.30 -19.60 -4.01 -34.58 -123.727 136.074 174.838 -124.700 142.500 102 500 0.090 0.007 0.028 0.051 0.015 0.027 0.031 0.094 0.021 0.174 0.013 0.057 0.027 0.017 0.017 0.090 0.137 0.076 0.003 0.022 0.024 0.003 0.043 0.255 0.008 0.015 0.015 0.017 0.022 0.025 0.013 0.078 0.056 0.059 0.055 0.092 0.161 0.028 0.002 0.048
7odcA K E 6 39.31 -19.93 0.60 -32.47 -21.22 1.25 -31.96 -23.04 2.24 -32.38 -166.604 152.765 179.364 -170.400 148.100 63 500 0.033 0.006 0.073 0.111 0.025 0.052 0.025 0.025 0.112 0.039 0.014 0.180 0.032 0.040 0.048 0.038 0.028 0.041 0.005 0.082 0.048 0.010 0.019 0.065 0.051 0.013 0.061 0.087 0.053 0.113 0.013 0.011 0.010 0.033 0.049 0.075 0.084 0.130 0.038 0.033
7odcA D L 7 40.06 -17.07 2.86 -31.37 -17.52 3.50 -30.06 -17.77 4.42 -29.68 46.445 42.105 171.655 42.700 51.500 53 500 0.049 0.005 0.188 0.114 0.010 0.035 0.022 0.014 0.089 0.075 0.008 0.067 0.069 0.063 0.052 0.080 0.038 0.019 0.003 0.019 0.065 0.008 0.133 0.040 0.038 0.147 0.055 0.009 0.064 0.042 0.009 0.162 0.015 0.013 0.028 0.067 0.027 0.016 0.008 0.059
(中略)
7odcA L H 416 37.59 -33.47 2.61 -24.75 -33.35 2.27 -23.26 -34.37 2.24 -22.11 -54.708 -52.456 -176.514 -56.300 -48.400 96 500 0.089 0.010 0.054 0.128 0.063 0.019 0.025 0.080 0.045 0.203 0.035 0.018 0.011 0.029 0.022 0.031 0.045 0.062 0.003 0.021 0.096 0.004 0.036 0.093 0.018 0.040 0.026 0.030 0.162 0.082 0.007 0.032 0.012 0.064 0.158 0.062 0.039 0.027 0.002 0.019
7odcA M H 417 38.99 -29.96 3.86 -25.51 -28.70 3.01 -25.51 -27.22 2.70 -24.61 -56.274 -44.526 179.408 -54.500 -46.700 21 500 0.022 0.018 0.008 0.016 0.030 0.011 0.006 0.130 0.013 0.278 0.139 0.008 0.015 0.018 0.021 0.026 0.023 0.095 0.009 0.092 0.089 0.021 0.010 0.028 0.062 0.029 0.006 0.130 0.028 0.231 0.043 0.023 0.009 0.011 0.026 0.047 0.018 0.056 0.021 0.096
7odcA K L 418 42.66 -30.80 4.37 -29.19 -31.20 3.26 -30.17 -30.65 1.97 -31.75 -63.576 0.000 0.000 -61.500 360.000 212 500 0.061 0.003 0.018 0.104 0.016 0.022 0.010 0.011 0.137 0.055 0.011 0.025 0.013 0.181 0.207 0.055 0.029 0.028 0.015 0.013 0.054 0.032 0.049 0.112 0.008 0.049 0.036 0.013 0.205 0.049 0.033 0.044 0.015 0.099 0.082 0.056 0.022 0.041 0.003 0.009
3hl4A G L 40 28.22 11.07 25.41 13.04 9999.99 9999.99 9999.99 11.07 25.41 13.04 0.000 -112.026 160.819 360.000 -109.300 123 500 0.123 0.005 0.101 0.055 0.007 0.213 0.032 0.023 0.043 0.026 0.011 0.049 0.021 0.015 0.015 0.094 0.130 0.017 0.002 0.007 0.049 0.004 0.072 0.028 0.008 0.078 0.034 0.040 0.152 0.049 0.007 0.068 0.044 0.072 0.155 0.052 0.021 0.064 0.002 0.008
3hl4A L L 41 22.05 11.01 27.29 9.76 9.87 28.22 9.32 8.59 27.99 8.50 -115.585 147.901 -172.091 -122.000 151.500 148 500 0.036 0.005 0.013 0.027 0.066 0.093 0.006 0.161 0.016 0.154 0.010 0.029 0.024 0.029 0.030 0.106 0.038 0.100 0.003 0.025 0.048 0.033 0.013 0.045 0.011 0.046 0.020 0.093 0.052 0.112 0.062 0.043 0.073 0.027 0.110 0.076 0.059 0.023 0.003 0.050
3hl4A R L 42 20.16 14.25 28.13 7.96 15.17 26.94 7.70 16.85 25.16 8.09 -119.774 -31.580 -176.785 -125.600 -21.700 219 500 0.052 0.077 0.068 0.165 0.015 0.018 0.017 0.029 0.052 0.021 0.016 0.038 0.040 0.054 0.154 0.079 0.046 0.025 0.003 0.031 0.083 0.016 0.083 0.061 0.021 0.092 0.006 0.019 0.050 0.043 0.006 0.063 0.049 0.043 0.070 0.078 0.103 0.045 0.023 0.056
(中略)
3hl4A Y H 213 63.08 9.48 64.25 15.54 9.29 64.10 14.03 10.06 63.39 12.63 -65.661 -46.053 -179.341 -56.800 -42.800 196 500 0.027 0.028 0.017 0.026 0.027 0.016 0.066 0.017 0.068 0.031 0.007 0.014 0.014 0.021 0.189 0.027 0.058 0.026 0.006 0.309 0.123 0.004 0.042 0.040 0.020 0.047 0.017 0.043 0.052 0.175 0.038 0.022 0.010 0.086 0.064 0.052 0.045 0.037 0.014 0.067
3hl4A D H 214 66.84 7.44 67.32 16.45 5.92 67.44 16.34 5.20 67.78 15.70 -62.464 -40.210 176.278 -65.300 -45.400 124 500 0.056 0.004 0.230 0.111 0.015 0.026 0.011 0.038 0.090 0.131 0.008 0.069 0.025 0.051 0.044 0.033 0.021 0.032 0.003 0.009 0.078 0.004 0.077 0.078 0.020 0.119 0.006 0.025 0.128 0.074 0.035 0.123 0.011 0.031 0.074 0.038 0.044 0.033 0.002 0.007
3hl4A V L 215 70.29 8.28 66.84 20.12 7.29 66.25 21.14 6.83 66.42 21.62 -101.834 0.000 0.000 -97.200 360.000 167 500 0.123 0.005 0.086 0.133 0.009 0.042 0.014 0.041 0.116 0.058 0.021 0.025 0.018 0.064 0.044 0.054 0.098 0.049 0.003 0.009 0.130 0.004 0.019 0.077 0.036 0.107 0.060 0.016 0.098 0.025 0.032 0.093 0.013 0.044 0.046 0.056 0.046 0.089 0.003 0.010
3n9xA R L 11 44.45 111.46 13.83 97.40 112.76 14.49 96.97 114.50 16.22 96.60 0.000 118.656 178.458 360.000 120.100 277 500 0.056 0.005 0.031 0.074 0.011 0.037 0.011 0.014 0.124 0.037 0.010 0.038 0.075 0.060 0.258 0.043 0.044 0.026 0.054 0.013 0.053 0.004 0.045 0.025 0.038 0.184 0.061 0.010 0.050 0.019 0.006 0.069 0.080 0.092 0.069 0.055 0.044 0.014 0.004 0.090
3n9xA E L 12 47.12 109.18 12.38 94.72 107.66 12.55 94.70 106.11 11.99 95.12 -83.520 143.638 176.270 -78.700 143.600 151 500 0.052 0.005 0.050 0.159 0.040 0.035 0.018 0.015 0.051 0.030 0.009 0.031 0.090 0.071 0.065 0.083 0.060 0.026 0.006 0.127 0.050 0.004 0.029 0.075 0.040 0.043 0.036 0.041 0.061 0.100 0.020 0.041 0.063 0.045 0.056 0.075 0.086 0.093 0.019 0.026
3n9xA N L 13 48.86 110.41 11.92 91.16 110.60 10.60 90.43 111.37 9.91 90.25 -60.339 149.894 -179.525 -51.700 146.200 110 500 0.070 0.006 0.049 0.068 0.017 0.067 0.056 0.057 0.068 0.049 0.035 0.115 0.020 0.033 0.027 0.046 0.093 0.109 0.003 0.013 0.060 0.004 0.015 0.138 0.052 0.063 0.005 0.060 0.054 0.108 0.016 0.030 0.108 0.043 0.030 0.041 0.042 0.101 0.019 0.018
(以下省略)
これの#で始まるコメント行の情報を読む限り、Pisces server cullpdb_pc60_res3.0_R0.3_d110719_chains16802.3820と書かれていることから、Piscesサーバーで2011年7月19日にPDB databaseをMaximum percentage identity 60%、解像度3.0 Å以下、R-Value <
0.3の条件(その他はデフォルトパラメータで良い?)で処理したものだと思われます。PISCESサーバーはDunbrack Labのページにあって、下図のようなフォーマットをしています。このPiscesサーバーでPDBエントリ全体を入力して、冗長性を排除しチェインごとに区切ったPDB IDのリストをゲットします。
これをやってみたところ、2018年9月末の時点で28000個くらいになりました……。まあ全部作るのはなかなか時間がかかりますが……とりあえずはまずvallファイルを1つ作ることからやってみましょう。
vallファイル作成のために必要となる計算処理
上記vallファイルを紐解いてみると、かなりの数のパラメータが必要とされていることがわかります。このうち特に処理上で重要となるのは
- 古いblastに同梱されているプログラム
blastpgpによる各タンパク質チェイン構造に対するPSSM計算結果- ちなみに現行のBLAST+では
blastpgpはpsiblastに置き換わっている。 - **2018年9月13日に古いblastはNCBIの公式ウェブサイトから削除され、ダウンロードできなくなった。**Linuxであれば
fragment_tools/pdb2vall/pdb_scriptsディレクトリにスタンドアローンで動くblastpgpが含まれているため、これを利用して処理を行うことが可能であるが、その他のOSではおそらく不可なので、psiblastを用いた計算に置き換える必要がある。
- ちなみに現行のBLAST+では
- DEPTHを使ったsolvent accessible surface areaやらなんやらの計算
- dsspを使った二次構造アサイン
- Rosettaの
idealizeとrelaxを用いた簡単な構造最適化計算処理
です。これらをひとまとめにしてやってくれるのが上記pdb2vallプログラムです。これを動かすために、以下で記す下準備をしておきましょう。
BLASTのnrデータベースの構築
vall作成処理はまずタンパク質構造ファイル&チェインIDに対して、そのアミノ酸配列に対するPSSM計算から始まります。この計算はblastpgpや現行のBLAST+ではpsiblastのプログラムが担っていますが、これをローカル環境で行うためには、BLASTのnon-redundantな配列データベースからnrデータベースをダウンロードしてきて、データベースを構築する必要があります。やり方の例は以下の通り
mkdir -p /path/to/database/blast && cd /path/to/database/blast
# nr.gzのダウンロード(注:37GB以上)
wget ftp://ftp.ncbi.nih.gov/blast/db/FASTA/nr.gz
# nr.gzの解凍(注:94GB以上)
gzip -f -d -c nr.gz > nr
# データベースの作成。legacyなblastのコマンドでは formatdb -t local_nr_base_filt -i local_nr_base_filt -p T に対応。max_file_sz指定がそろそろ必要(デフォルトは1GB)。
makeblastdb -title nr -dbtype prot -in nr -max_file_sz 2GB
こんな感じ。これをローカルマシンまたはワークステーションの共有ディレクトリに置いておきます。
pdb_seqres.txt, ss_dis.txtの入手
最新版の全PDBの配列リストであるpdb_seqres.txtと、その二次構造アサインの情報であるss_dis.txtをダウンロードする必要があります。pdb2vallディレクトリ内部の決められた箇所にディレクトリを作成し、そこにこの2つのファイルを設置しておく必要があります。
cd /path/to/Rosetta/tools/fragment_tools/pdb2vall/database/rcsb_data/
mkdir derived_data
# pdb_seqres.txt.gzの入手
curl ftp://ftp.wwpdb.org/pub/pdb/derived_data/pdb_seqres.txt.gz -O
gunzip pdb_seqres.txt.gz
mv pdb_seqres.txt derived_data
# ss_dis.txtのダウンロードと展開をしているだけの単純なスクリプトの実行
chmod +x get_ss_dis.sh
./get_ss_dis.sh
pdb_seqres.txtはこんな感じの内容。
>101m_A mol:protein length:154 MYOGLOBIN
MVLSEGEWQLVLHVWAKVEADVAGHGQDILIRLFKSHPETLEKFDRVKHLKTEAEMKASEDLKKHGVTVLTALGAILKKKGHHEAELKPLAQSHATKHKIPIKYLEFISEAIIHVLHSRHPGNFGADAQGAMNKALELFRKDIAAKYKELGYQG
>102l_A mol:protein length:165 T4 LYSOZYME
MNIFEMLRIDEGLRLKIYKDTEGYYTIGIGHLLTKSPSLNAAAKSELDKAIGRNTNGVITKDEAEKLFNQDVDAAVRGILRNAKLKPVYDSLDAVRRAALINMVFQMGETGVAGFTNSLRMLQQKRWDEAAVNLAKSRWYNQTPNRAKRVITTFRTGTWDAYKNL
>102m_A mol:protein length:154 MYOGLOBIN
MVLSEGEWQLVLHVWAKVEADVAGHGQDILIRLFKSHPETLEKFDRFKHLKTEAEMKASEDLKKAGVTVLTALGAILKKKGHHEAELKPLAQSHATKHKIPIKYLEFISEAIIHVLHSRHPGNFGADAQGAMNKALELFRKDIAAKYKELGYQG
アミノ酸配列がチェインごとにまとめられています。一方でss_dis.txtはこんな感じ。
>101M:A:sequence
MVLSEGEWQLVLHVWAKVEADVAGHGQDILIRLFKSHPETLEKFDRVKHLKTEAEMKASEDLKKHGVTVLTALGA
ILKKKGHHEAELKPLAQSHATKHKIPIKYLEFISEAIIHVLHSRHPGNFGADAQGAMNKALELFRKDIAAKYKEL
GYQG
>101M:A:secstr
HHHHHHHHHHHHHHGGGHHHHHHHHHHHHHHH GGGGGG TTTTT SHHHHHH HHHHHHHHHHHHHHHH
HHTTTT HHHHHHHHHHHHHTS HHHHHHHHHHHHHHHHHH GGG SHHHHHHHHHHHHHHHHHHHHHHHHT
T
>101M:A:disorder
---------------------------------------------------------------------------
---------------------------------------------------------------------------
----
>102L:A:sequence
MNIFEMLRIDEGLRLKIYKDTEGYYTIGIGHLLTKSPSLNAAAKSELDKAIGRNTNGVITKDEAEKLFNQDVDAA
VRGILRNAKLKPVYDSLDAVRRAALINMVFQMGETGVAGFTNSLRMLQQKRWDEAAVNLAKSRWYNQTPNRAKRV
ITTFRTGTWDAYKNL
>102L:A:secstr
HHHHHHHHH EEEEEE TTS EEEETTEEEESSS TTTHHHHHHHHHHTS TTB HHHHHHHHHHHHHHH
HHHHHH TTHHHHHHHS HHHHHHHHHHHHHHHHHHHHT HHHHHHHHTT HHHHHHHHHSSHHHHHSHHHHHHH
HHHHHHSSSGGG
>102L:A:disorder
---------------------------------------------------------------------------
---------------------------------------------------------------------------
-------------XX
pdb_seqres.txtの配列に対応した二次構造情報とdisorder情報が載っているようです。
BioPerlの設定
pdb2vallの処理の中ではBioPerlを必要とするため、これをインストールします。macOS、CentOS 7 いずれもCPANを使った方法が楽だと思います。ただし、ちょっとやり方が異なります。
参考: https://bioperl.org/INSTALL.html
macOSの場合はHomebrewでexpatを入れておいた後、perlbrewでPerlをインストールし、そのPerlに対応したBioperlを入れておきます。
# expatを先にHomebrewで入れておく。
brew install expat
# perlbrewとBioperlのインストール
curl -kL http://install.perlbrew.pl | bash
source ~/perl5/perlbrew/etc/bashrc # ←これを手持ちの~/.bashrcにも追記しておく
# perl 5.26.2のインストール
# perlbrew availableでダウンロード可能なバージョンを確認
perlbrew install 5.26.2
perlbrew install-cpanm
# cpanに追加モジュールのインストール
cpan
cpan[1]> install Algorithm::Munkres Array::Compare Convert::Binary::C Data::Stag Graph GraphViz Math::Random Bio::AlignIO
# ここでCtrl-dなどでcpan を抜ける
# Bio::Perlのインストール
cpanm Bio::Perl --force # --forceをつけないとうまくインストールされないものがある
CentOS 7の場合でシステムのperl(/usr/bin/perl)にCPANを入れるようにするには
yum -y install perl-CPAN perl-CPANPLUS
perl -MCPAN -e shell
## ここで環境構築を色々聞かれるけれど適当にyesにしました
# 以下cpan[1] >みたいな状態で入力すべきもの
install CPAN
reload cpan
install Module::Build
o conf commit MB
o conf commit
install Algorithm::Munkres Array::Compare Convert::Binary::C Data::Stag Graph GraphViz Math::Random Bio::AlignIO
force install Bio::Perl
# cpanここまで
こんな感じ。CentOS 7のときはなんか途中で色々Yesとか答えないといけなかったような。あと、Bio::PerlはいずれのOSでもforceをつけないとうまくいかないっぽいです。
DEPTHのインストール
タンパク質内部のSASA計算のためにRosettaはDEPTHというプログラムを使って測定しているらしい(?)。一応pdb2vallに同梱されていますが、だいたいの環境の場合、うまく動作しないみたいなので、自前でインストールし直した方がいいです。
http://cospi.iiserpune.ac.in/depth/htdocs/download.htmlからダウンロード
wget http://cospi.iiserpune.ac.in/depth/htdocs/program/depth-2.0.tar.gz
tar zxvf depth-2.0.tar.gz
mv depth-2.0 /path/to/your/directory
cd depth-2.0/bin
make
QUICK_STARTを見るとなんやかんやさらにパラメータの設定が必要らしいですが、ここでは使わないので省略します。
注意点として、**makeまたはこの後のmake installをしたあとにこのディレクトリを移動させてはいけません。**内部で絶対PATHが決め撃ちされるからです。しかも、このDEPTH、エラー処理がうまく働いていないみたいなので、動作に失敗すると無限ループに入ります。pdb2vallではこのDEPTHのPATHを指定して使うことになりますが、DEPTHの前段階で死んでいるとDEPTHが無限に終わらないループに入るのできちんとインストールしておきます。
pdb2vall.cfgの設定
/path/to/Rosetta/tools/fragment_tools/pdb2vallに存在するコンフィグファイルであるところのpdb2vall.cfgを書き換えます。ここには使う計算コア数やblastのnrデータベース、各種プログラムのPATHを書き記しておきます。
[pdb2vall]
rosetta_path = /usr/local/package/Rosetta/Rosetta3.9_shared
fragment_picker_num_cpus = 1
blastpgp_num_cpus = 1
blastpgp = /path/to/fragment_tools/pdb2vall/pdb_scripts/blastpgp
bl2seq = /path/to/fragment_tools/pdb2vall/pdb_scripts/bl2seq
formatdb = /path/to/fragment_tools/pdb2vall/pdb_scripts/formatdb
nr = /raid1/share/database/blast/db.20180921/nr
depth = /usr/local/package/depth-2.0/bin/DEPTH
depth_num_cpus = 1
コア数を指定する箇所が3箇所ほどありますが、ぶっちゃけそんなにコア数を上げても大差ない気がするので、全部1で良いと思います。
環境変数設定など
以下の4つの環境変数を、pdb2vall.pyを動かす前に設定しておく必要があります。これらはタンパク質構造データ(PDBフォーマット)のダウンロードと、BLAST処理を行うときに重要な環境変数です。以下はその例。
export PDB_DIR="/raid1/share/database/PDB_uncompressed/divided_dot_pdb/pdb"
#export INET_HOST="remotehost"
export BLASTDB=/raid1/share/database/blast/db.20181003/nr
export BLASTMAT=/raid1/share/database/blast/matrices
PDB_DIRは適当になにか設定しておきます。実際には、pdb_scripts/get_pdb_new.pyに存在するdef download_pdb 内の関数を参照してもらえばわかるのですが、取得に失敗するとwgetでPDBファイルをウェブ経由で取得しようとし始め、たいていは問題なく取得してくれます。もし、サーバー内にミラーリングデータが有るのであれば、それを利用するようにしているのが環境変数PDB_DIRとINET_HOSTです。まあ詳しくは上の関数部分の処理をよく読んでください。
BLASTDBには先述のBLASTのnrデータベースのPATHを、BLASTMATには置換行列のデータディレクトリを指定しておきます。これは必須で、ftp://ftp.ncbi.nlm.nih.gov/blast/matrices/のmatricesディレクトリをまるごとダウンロードしてどこかに置いておけばOKです。
その他細かな設定項目
pdb2vall処理をちょっと使いやすくするために、いくつか簡単な変更を加えます。
database/rcsb_data/update_rcsb_data.pl の書き換え
このファイルはPDBファイルデータを自動的にrsyncでアップデートしようとするが、毎度自動的にやられても困るので、5行目くらいに exit(0); を追記。これで何もしなくなります。
structure_profile_scripts/make_sequence_fragments.pl の書き換え
29 行目くらいの"$ROSETTA_PATH/main/source/bin/fragment_picker.boost_thread.linuxgccrelease";
の.linuxgccreleaseを削除しておきます(Linux以外の環境の場合のみ必要)。
sequence_profile_scripts/run_psiblast_filtnr_tight.pl の書き換え
62行目にある(-s $DB) or die "ERROR! NR database $DB does not exist\n";はなぜかよく左辺の判定に失敗してしまうので、これまでの設定を踏まえていればここはコメントアウトしても問題ないようなので、先頭に#をつけて保存しておく。
structure_profile_scripts/create_checkpoint_from_fasta_alignment.pl の書き換え
Bioperlで設定したものを使うために、1行目の#!/usr/bin/perlを#!/usr/bin/env perlに変更する(Linux以外の環境の場合のみ必要)。
pdb_scripts/get_pdb_new.pyとfetch_raw_pdb.pyの書き換え
この2つはPDBフォーマットのタンパク質構造情報を取得することを担うスクリプトです。上でも少し書きましたが、get_pdb_new.pyの方は処理の最初の方でblastpgpを行うときにPDBを取得するタイミングで使用します。後者のfetch_raw_pdb.pyはblastpgpによってcheckpointファイルが作成された後、structure_profileを作成するタイミングで使用します。
……なんでこの2つを分けて使っているのかよくわかりませんし、効果が重複している上に設定項目が独立になっているので意外とややこしいです。しかも、前者はラボ内PDBデータベースを構築していなくてもwgetを使って取得しようとしてくれるのですが、後者にはその機能はないようなので、ラボ内PDBデータベースがない場合には適当にコマンドを作って上げる必要があります。
pdb2vall.pyの書き換え
本体のこのプログラムですが、python2で書かれたままです。まずは62, 63行目にあるidealization_app,
relax_appについて、.linuxgccreleaseがついていると環境によっては動かないので、この文字をまるごと削除するか、環境に合わせた適切なものに変えましょう。
pdb_scripts/blastpgp -a 1 -t 1 -j 2 -h 0.0000000001 -e 0.0000000001 -b 0 -k 0 -d nr -M BLOSUM62 -o 7odcA.1.blast -C 7odcA.1.chk -Q 7odcA.1.matrix -i 7odcA.fasta
これの動作を確認しておきます。
dsspの動作確認
pdb2vall/structure_profile_scripts/dsspに存在するdsspが動くかどうかを、/path/to/dsspと打ってみて、動作するかどうかを確認しておきます。もしexec format errorと表示されるなら、これは使用不能です(macOSによくある)。
使えない場合は、macOSの場合はbrew install xsspでインストールできるmkdsspコマンドをここにシンボリックリンクとして貼っておけば代用することができます。
# 元のdsspバイナリのリネーム
mv /path/to/pdb2vall/structure_profile_scripts/dssp/dssp /path/to/pdb2vall/structure_profile_scripts/dssp/dssp.bak
# Homebrewでxsspのインストール。この中にmkdssp, mkhsspコマンドがある。
brew install xssp
# 新しくインストールしたmkdsspコマンドを上と同じ場所にdsspという名前で置く
ln -s /usr/local/bin/mkdssp /path/to/pdb2vall/structure_profile_scripts/dssp/dssp
pdb2vall.pyでフラグメントファイルを作成
以上の準備が終わったらようやく本題のフラグメントファイル作成です。とは言ってもこれ自体は非常にシンプルで、以下のコマンドを入力すればできます。
# フラグメントを作りたいPDBファイルのチェイン名を入力。必ず最初の4文字は小文字、最後のchain IDは大文字。
pdbid=2e7dA
# 適当なディレクトリを先に作っておいたほうが良い
mkdir -p ${pdbid} && cd ${pdbid}
python2.7 /path/to/fragment_tools/pdb2vall/pdb2vall.py -p ${pdbid}
以上の準備が整っていればきっとできます。環境がしっかり整っているマシンならば1つあたり4〜6時間くらいでできます。
このフラグメント作成処理は、後述のようにblastpgpによるPSSM作成処理(sequence profile作成処理)と、DEPTH処理以降のstructure profile作成処理の2つに大別できます。このうち前半のblastpgpによるPSSM作成は、その計算時間が計算環境に大きく依存します。これはpsiblastなどを配列解析をやったことがある人にはよく知られた現象なのですが、処理速度を上げる簡単な方法としては、
- nrデータベースはHDDよりもSSD上にある方がベター
- RAMがたくさん積んである環境がベター
ということを覚えておきましょう。反対に、後半のstructure profile作成処理はこの2項目は全く関係せず、CPUのシングルコア性能のみで処理を行う(マルチコア処理にもできるけれどあまり作成速度に大差ない)という特徴があります(←しかも10年前のCPUでもそんなに大差ない)。よって、もし大量にフラグメントファイルを作りたいけれど、計算環境が限られているという場合には、前半のblastpgp処理部分だけ速度が出る環境で行い、後半は別のマシンで行うというふうに分割するとトータルで作成速度が上がります。
とはいえ最初のうちは準備不足のために、エラー落ちしてしまうことが多々あるでしょう。以下にその処理の流れを書いておきます。
pdb2vall.pyの処理の流れ
やっている内容は以下のコマンドを順に処理しているだけです。例としてPDBの7odcのchain Aをとると、
-
get_pdb_new.pyを使って7odc.pdbのPDBファイルをダウンロード - 1段階目の
blastpgp処理。thresholdは0.0000000001。 - 2段階目の
blastpgp処理。thresholdは0.00001 -
7odcA.2.chkから7odcA.checkpoint(チェックポイント 1) -
structure_profile_checkpointディレクトリを作成し、structure_profile_scripts/make_depthfile.py 7odcA.pdbを実行し、DEPTH処理を開始する。この後しばらくはDEPTH実行のログ。**DEPTH処理は1コアでもさすがに1〜2分で終わるはずなので、このDEPTH処理が終わらない場合はDEPTH無限ループに入っている可能性がある。**この場合は、/path/to/DEPTH -thread 1 -i structure_profile_checkpoint/7odcA.pdb -o structure_profile_checkpoint/7odcA.depthを実行してみたときに、中間ファイルが現れていないと無限ループ状態である可能性が高い。処理が終わると、pdb2vall/structure_profile_checkpointの中にインプットの-pで指定したPDB_ID+Chainのディレクトリが作成され、そこに7odcA/7odcA.depth-residue.depthというファイルが作成される。 - DEPTHが終わったら
fragment_picker処理を行い、7odcA.50.9mers.ali.fastaを始めとした9-merファイルを作成しようとする。 -
blastpgpを再度呼び出し、7odcA.50.9mers.ali.fasta.new.blastから7odcA.50.9mers.ali.fasta.new.blast.chkを作成する。さらに、ここから7odcA.50.9mers.ali.fasta.new.blast.checkpointファイルを作成する。ちなみに、このときにデータベースはplaceholder_seqs/placeholder_seqsを参照しているが、これはダミーデータベースで、要は処理をとにかく行うための見せかけだけのデータベースらしい。(チェックポイント 2) - 上記処理が終了したら、Rosettaのプログラムである
idealizeとrelaxを順に使って構造情報を少し最適化し、7odcA_0001.pdbと7odcA_0001_0001.pdbを作り出す。残基が長いと少し処理に時間がかかる。 -
7odcA_0001_0001.pdbに対しdsspを使って二次構造情報を取得する - 以上の情報を用いて、Rosettaフラグメントファイルの
7odcA.vallを書き出し、処理を終了する。
こんな感じ。
vallファイルに含まれる0カラムの補修
これでだいたいできたはずなんですが……なぜかいくつかのパラメータの列が0で埋まっています。具体的には以下の7つの列です。
0行列になっている残りのパラメータ
- B-factor
- CB_x, CB_y, CB_z
- CEN_x, CEN_y, CEN,z
- n_ali (number of alignments used for PSSM)
これはオリジナルのpdb2vall.pyの最後の行を見てみると、なんと0出力するように決め撃ちされています。しかし幸いにも元の既成品のフラグメントライブラリにはこれらに何の値を埋め込めばよいかの説明が書かれているので、0出力が嫌ならばここを修正することが可能です。……でもなんで0にされてるんだろう?もしかして不必要だからなのか……?
まあ気味が悪いので、これらを修正したいところです。この説明をいちいちするのはめんどくさいので、これを補修したスクリプト https://github.com/BILAB/fragment_tools/blob/master/pdb2vall/pdb2vall.py を置いておきますので勝手に理解してください(投げやり)。omi_UTくんありがとう。
これでRosettaフラグメントライブラリ作成についてのお話はだいたい終わりです……でももうちょっとだけ余談が続くんじゃ
(余談) blastpgpをBLAST+のpsiblastでやるには
はじめに書いたように、legacyなblastpgpは2018年9月13日から配信が完全に打ち切られ、もうダウンロードが不可能になってしまいました。そこで、現在も開発とサポートが行われ、しかもより早くて正確だという仕様になっている後続のBLAST+のpsiblastに処理を置き換えたいというのは当然の流れです。Linux OSの方はfragment_toolsに同梱されているblastpgpがスタンドアローンで動くので、まあいいんですけど、やっぱり後々のことを考えて、psiblastを使ってRosettaフラグメント作成をしたいという気持ちは残ります。そこで、その処理を考察してみます。
まずは最新のBLAST+をインストールする必要がありますが、CentOS 7の場合は
# download the RPM package of ncbi blast+ 2.7.1 for CentOS 7
wget ftp://ftp.ncbi.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.7.1+-1.x86_64.rpm
wget ftp://ftp.ncbi.nih.gov/blast/executables/blast+/LATEST/ncbi-blast-2.7.1+-1.x86_64.rpm.md5
# ncbi-blast-2.7.1+-1.x86_64 requires these 3 programs
yum -y install perl-List-MoreUtils perl-Digest-MD5 perl-Archive-Tar
# install the RPM package
mkdir -p /usr/local/package/blastplus/2.7.1
rpm -Uvh --prefix=/usr/local/package/blastplus/2.7.1 ncbi-blast-2.7.1+-1.x86_64.rpm
でインストールできます。macOSの場合はbrew install blast。
このBLAST+モジュールに同梱されているlegacy_blast.plコマンドを使うと、blastpgpでの処理をpsiblastに変換表示させることができるのでそれを見てみると、blastpgp処理はpsiblastでは以下のようになると思われる。
変換処理コマンド
legacy_blast.pl blastpgp -i 7odcA.fasta (...other options...) --print_only
psiblastでのコマンド
# !/bin/sh
INPUT=7odcA
BLASTDB=/path/to/database/blast/db.20181003/nr
export BLASTMAT="/path/to/database/matrices"
psiblast -db ${BLASTDB} \
-query ${INPUT}.fasta \
-matrix BLOSUM62 \
-num_iterations 2 \
-evalue 0.0000000001 \
-num_threads 1 \
-inclusion_ethresh 0.0000000001 \
-num_alignments 0 \
-comp_based_stats 1 \
-out ${INPUT}.1.blast \
-out_ascii_pssm ${INPUT}.1.matrix \
-out_pssm ${INPUT}.1.chk
psiblast -db ${BLASTDB} \
-in_pssm ${INPUT}.1.chk \
-num_iterations 2 \
-evalue 0.00001 -num_threads 1 \
-inclusion_ethresh 0.00001 \
-num_alignments 0 -comp_based_stats 1 \
-out ${INPUT}.2.blast \
-out_ascii_pssm ${INPUT}.2.matrix \
-out_pssm ${INPUT}.2.chk
たぶんこれであっているはず。
この7odcA.2.chkから7odcA.checkpointファイルに変換するには新しく専用のスクリプトが必要になるのですが、これは我々のgithubのfragment_toolsのpdb2vall/sequence_profile_scripts/convert_asciichk_to_checkpoint.pyを使えば、python3 convert_asciichk_to_checkpoint.py -p 7odcAとすることでうまいこと変換してくれるはずです(7odcA.2.chkがある同ディレクトリ上で実行してください)。
blastpgpとpsiblastでのcheckpointファイルの値の比較
blastpgpとpsiblastでどれくらい値が変わってくるかを確かめるために、PDB: 16pkA, 16vpA, 1a0aAで最後の${input}.2.chkから作り出した${input}.checkpointファイルの行列成分の差分をとってみます。blastpgpのバージョンは2.2.26を、psiblastは2.7.1を使いました。図は@omi_UTくんが提供してくれました。
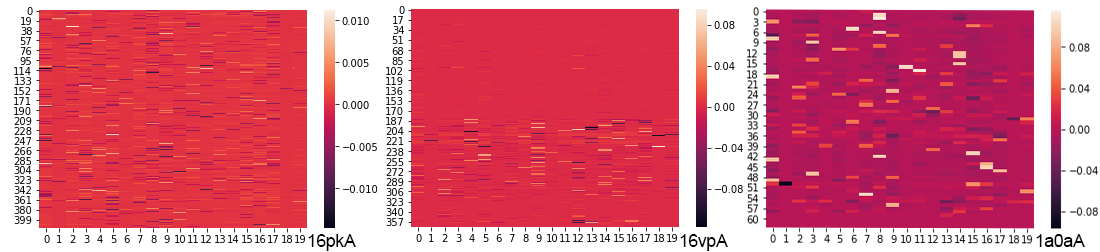
だいたいあっているようなところもありますが、1a0aAなんかは結構値がずれている箇所があることがわかります。これを考察してみたんですが、二者のアルゴリズムのblastでヒットしている配列やe-valueの値が変わってきているので、クエリ配列によっては取得する類縁配列に大きな差があることがわかります。これ以上の詳しい考察はおそらくアルゴリズム自体の違いになってしまうので割愛しますが、一応psiblastは、以前のblastpgpよりも速くて正確とか言ってるので、まあこっちを使っても問題はない気がします。ただ、Rosettaフラグメントを改めて作るときにはどちらかのアルゴリズムに統一したほうがいいかもしれません。
完全にpsiblastに置き換えてpdb2vall.pyを作成するために、現在GitHubのhttps://github.com/BILAB/fragment_tools/tree/master/pdb2vall においてblastplus_~~で始まる各種スクリプトを開発しています。完全に問題なく動くまでもうちょっとだけ補修が必要なのですが、そこはComing Soon...とさせてください。
おわりに
Rosettaフラグメントライブラリの作成手順を色々分解して遊んでいる中でRosettaへの理解も少しずつ深まってきました。これからもRosettaについて気付いたことがあればたびたび記事を投稿していきたいと思います。