最近プログラミング言語のJuliaを勉強し始めていて、JuliaからPyCall.jlを使ってPythonのModellerをimportして使えたらいいな〜と思ったので、その時の作業記録を残します。また、pycallやpyimportを使う必要性があったことについても述べます。
Modellerのサンプルスクリプトは公式のものを含め色々存在していますが、ここでは私のMDシミュレーションのチュートリアル記事であるMDシミュレーションのチュートリアル〜PDB: 1LKEの場合〜で使われているalign.pyをJuliaから使えるようにしてみます。動作に必要な1LKE_truncated.pdb, alignment.aliは事前に用意しておきます。
動作環境
- Julia 1.5.3
- Conda v1.5.1
- PyCall v1.92.2
元となるpythonのコード
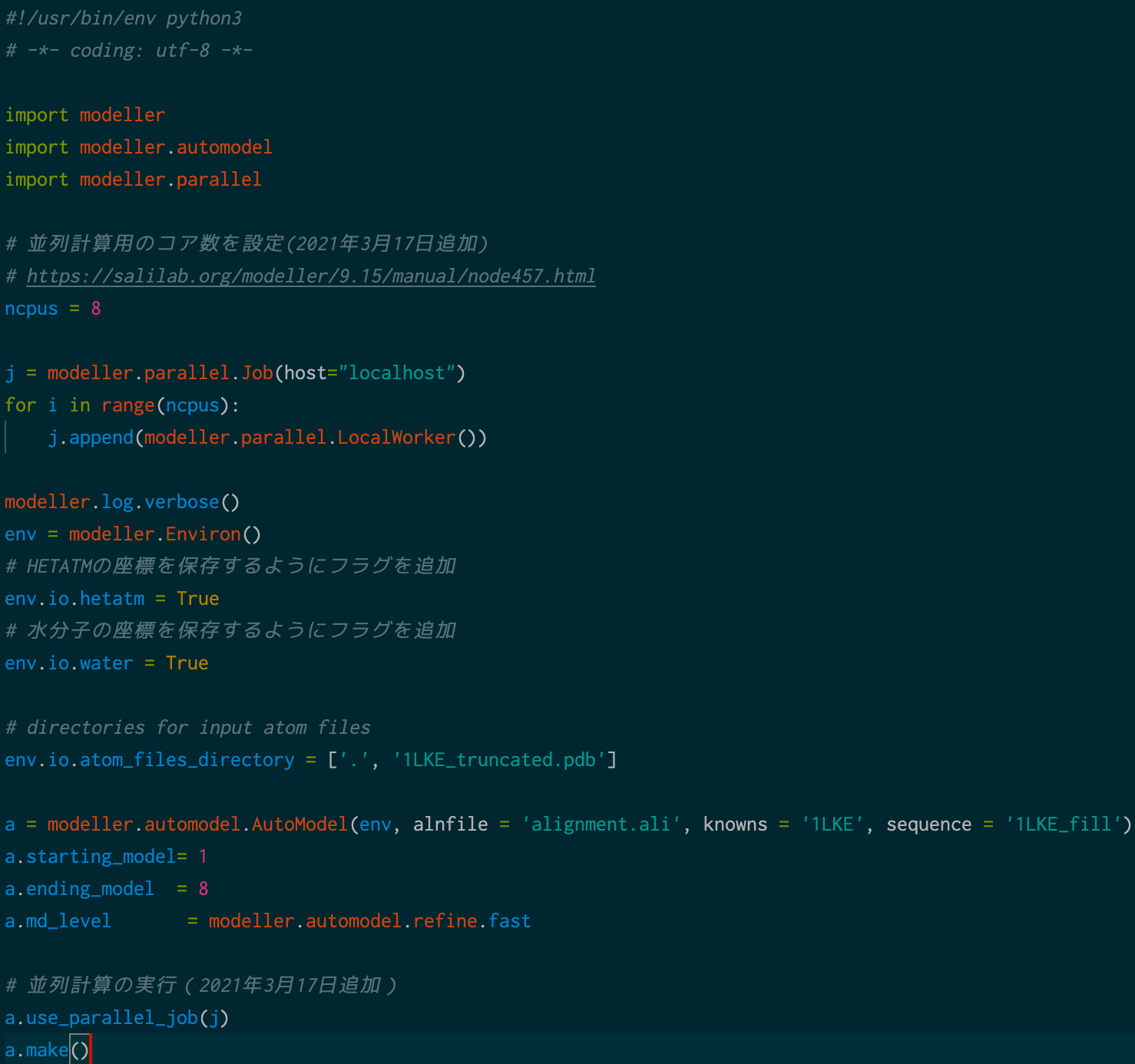
以下のalign.pyを題材にします。
# !/usr/bin/env python3
# -*- coding: utf-8 -*-
import modeller
import modeller.automodel
import modeller.parallel
# 並列計算用のコア数を設定
# https://salilab.org/modeller/9.15/manual/node457.html
ncpus = 8
j = modeller.parallel.Job(host="localhost")
for i in range(ncpus):
j.append(modeller.parallel.LocalWorker())
# logを詳細にアウトプットする
modeller.log.verbose()
# Environのインスタンスを生成。Environ()内にrand_seed=を設定することもできる
env = modeller.Environ(rand_seed=1)
# HETATMの座標を保存するようにフラグを追加
env.io.hetatm = True
# 水分子の座標を保存するようにフラグを追加
env.io.water = True
# directories for input atom files
env.io.atom_files_directory = ['.', '1LKE_truncated.pdb']
a = modeller.automodel.AutoModel(env, alnfile = 'alignment.ali', knowns = '1LKE', sequence = '1LKE_fill')
a.starting_model= 1
a.ending_model = 8
a.md_level = modeller.automodel.refine.fast
# jで設定したコアだけ並列計算させる設定
a.use_parallel_job(j)
# モデリング実行
a.make()
これをPyCall.jlを用いてJuliaに移植します。それがこちら
# 最初のみ以下の#を外して必要なパッケージをインストールする
# using Pkg
# Pkg.add("PyCall")
# Pkg.add("Conda")
using PyCall
using Conda
# Conda.update()
# Conda.add_channel("salilab")
# Conda.add("modeller")
# PYTHONバイナリへのPATHの設定
ENV["PYTHON"] = ENV["HOME"] * "/.julia/conda/3/bin/python3.8"
Pkg.build("PyCall")
# PythonからModellerをimportしたときのconst変数名
const modeller = pyimport("modeller")
const automodel = pyimport("modeller.automodel")
const parallel = pyimport("modeller.parallel")
ncpus = 8
# ここ注意点
j = pycall(parallel[:Job], PyObject, host="localhost")
for i in 1:ncpus
append!(j, [parallel[:LocalWorker]()])
end
modeller[:log].verbose()
env = modeller[:environ](rand_seed=1)
# Trueはtrueに
env.io.hetatm = true
env.io.water = true
# directories for input atom files
env.io.atom_files_directory = [".", "1LKE_truncated.pdb"]
a = automodel[:AutoModel](env, alnfile = "alignment.ali", knowns = "1LKE", sequence = "1LKE_fill")
a.starting_model = 1
a.ending_model = 8
a.md_level = automodel[:refine].fast
# jで設定したコアだけ並列計算させる設定
a.use_parallel_job(j)
# モデリング実行
a.make()
上記align.jlをJulia上で入力するとModellerを使ったモデリングが実行されます。
解説と注意点
PyCall.jlのREADMEを読むのが正攻法ですが、とりあえず私が現時点までで理解したところを書いてみます。
初回のみの操作
初回はJuliaのcondaパッケージでPython3とmodellerをインストールする操作が必要です。salilabチャンネルにあるmodellerをインストールするために
using Pkg
Pkg.add("PyCall")
Pkg.add("Conda")
using PyCall
using Conda
Conda.update()
Conda.add_channel("salilab")
Conda.add("modeller")
を実行しておきます。
# PYTHONバイナリへのPATHの設定
ENV["PYTHON"] = ENV["HOME"] * "/.julia/conda/3/bin/python3.8"
Pkg.build("PyCall")
ここはcondaで入れたPython3.8へのPATHを設定しています。もうそろそろしたらデフォルトがpython3.8からpython3.9にアップグレードされるかもしれませんので、上記では動作しないかもしれません。
Pythonライブラリのimport
Pythonライブラリのimportを行うコマンド
import A.B as C
は
const C = pyimport("A.B")
と書けます。……たぶん。
PythonにおいてA.B.C()とあったときに、仕様を熟知していないとCがクラスなのか(つまりインスタンスを生成しているのか)、それともメソッド(クラス内関数)なのか、変数なのかわかりにくいときがあります。クラスである場合にはA[:B]と書く必要があります。
これは例えばVSCode上でPython, Pylance拡張機能を使っているとわかりやすいと思います。
テーマごとの配色は異なりますが、クラスの場合と変数/メソッドの場合で色が違っています。上の図ですとクラスの場合は赤色、変数/メソッドの場合は青色になっていますね。
Juliaに持ってきたときに型が勝手に変わってしまう問題について
Juliaへの移植は上記の変換ルールを守れば、あとはJulia文法を少し学べばだいたい移植可能な気もするのですが、1つだけ今回ハマった点がありました。それは
j = modeller.parallel.job(host="localhost")
これをJuliaに移植するときです。これを上記の変換ルールで単純に書き換えてみると
j = parallel[:Job](host="localhost")
になって然るべきかと思います。これ自体は間違いではなく、実行してみると一見なんの問題もなく動作するように見えます。
julia> j = parallel[:Job](host="localhost")
Any[]
さらに次のLocalWorker()をappendするところも実行してみます。
julia> for i in 1:ncpus
append!(j, [parallel[:LocalWorker]()])
end
julia> print(j)
Any[PyObject <Worker on localhost>, PyObject <Worker on localhost>, PyObject <Worker on localhost>, ...]
julia> typeof(j)
Array{Any,1}
変数jが普通のArray型、Pythonで言うところのlist型っぽくなっています。
しかし、この状態で実行していくと最後のa.make()のところで以下のようなエラーが発生しました。
(ここまでModellerアウトプット)
Pseudo atoms in memory : 0
ERROR: PyError ($(Expr(:escape, :(ccall(#= /Users/YoshitakaM/.julia/packages/PyCall/tqyST/src/pyfncall.jl:43 =# @pysym(:PyObject_Call), PyPtr, (PyPtr, PyPtr, PyPtr), o, pyargsptr, kw))))) <class 'AttributeError'>
AttributeError("'list' object has no attribute 'queue_task'")
File "/Users/YoshitakaM/.julia/conda/3/lib/python3.8/site-packages/modeller/automodel/automodel.py", line 150, in make
self.multiple_models(atmsel)
File "/Users/YoshitakaM/.julia/conda/3/lib/python3.8/site-packages/modeller/automodel/automodel.py", line 252, in multiple_models
self.parallel_multiple_models(atmsel)
File "/Users/YoshitakaM/.julia/conda/3/lib/python3.8/site-packages/modeller/automodel/automodel.py", line 271, in parallel_multiple_models
job.queue_task(ModelTask(self, num, atmsel))
これはAttributeErrorが起きているということですが、「listオブジェクトにqueue_taskというattributeはない」とのことでした。queue_taskとは何かとModellerのソースコードを調べてみると、modeller.parallel.Job.queue_task()のメソッドであることがわかりました。つまり何が起きているかと言うと、変数jが本来modeller.parallel.Jobのオブジェクトクラスであるべきところなのにも関わらず、リスト型オブジェクトクラス扱いに自動的に変換されてしまっており、このためattribute errorを起こしているようでした。
事実、pythonの方で変数jの型を調べてみるとlist型でないことがわかります。
>>> j = modeller.parallel.Job(host="localhost")
>>> print(type(j))
<class 'modeller.parallel.job.Job'>
これを解決するには、Juliaで変数jを定義するときに以下のように書く必要があります。
j = pycall(parallel[:Job], PyObject, host="localhost")
pycallを使って明示的にこうすれば、
julia> j = pycall(parallel[:Job], PyObject, host="localhost")
PyObject <Parallel job []>
jがPythonのオブジェクト<Parallel job []>となっていました。これでModellerをJulia上でparallelを使った並列処理をさせることができました。