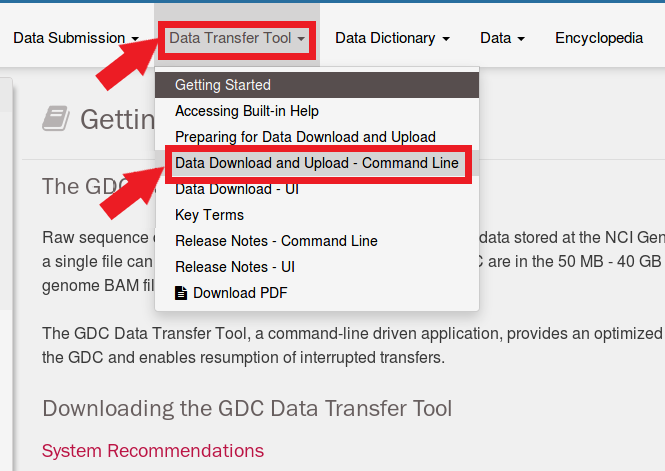
#TCGAからの原始的なデータ取得
癌研究者にとっては一つのインフラとなった感のある、米国のデータベースTCGA(The Cancer Genome Atlas)。2016年6月にサイトの構造が大幅に変わり、それまでの体裁からNIHのデータベースサイトであるGDC data portalの一部分という形に変貌した。
この変更と同時に、入手可能なデータもアップデートがなされ、RNA-Seqデータについて述べると
- マッピング法が変わった
- アノテーションがGene Symbol→Ensembl準拠となった
- 発現量を参照できる遺伝子数が増えた。
というような変化が起きている。以前のRNA-Seqデータで得られる発現情報は約20500遺伝子分であったところが、2016年以降のデータでは60500種類ちかい遺伝子の発現情報が得られることになった。そして個人的には大きな変化と感じたのが
- データ入手法が変わった
という点だ。以前はファイル入手にあたって特別なツールは必要なく、解凍ツールさえあればよかったものが、現在はNIHの配布するプログラムであるGDC-clientが必須になったといってよいだろう。私個人のWindows10環境だとエラーが出るので、VirtualBoxでLinuxOSを動かすか、MacOSまたはLinux搭載PCで行うことになる。
本稿では、GDC data portalからTCGAのメラノーマ検体群のRNA-Seqファイルの入手を例にダウンロードと発現リストの作成法を記して行きたい。 (12/23/2018注記:発現リスト作成法は別項にて解説予定です)
本稿の作業は、Ubuntu16.04LTSの環境で行っております。本稿に掲載されている画像は、特記なき場合は2018年12月に作成したものです。
###Data portalに行く
####ほしいファイルが記載されたマニフェストを作成する
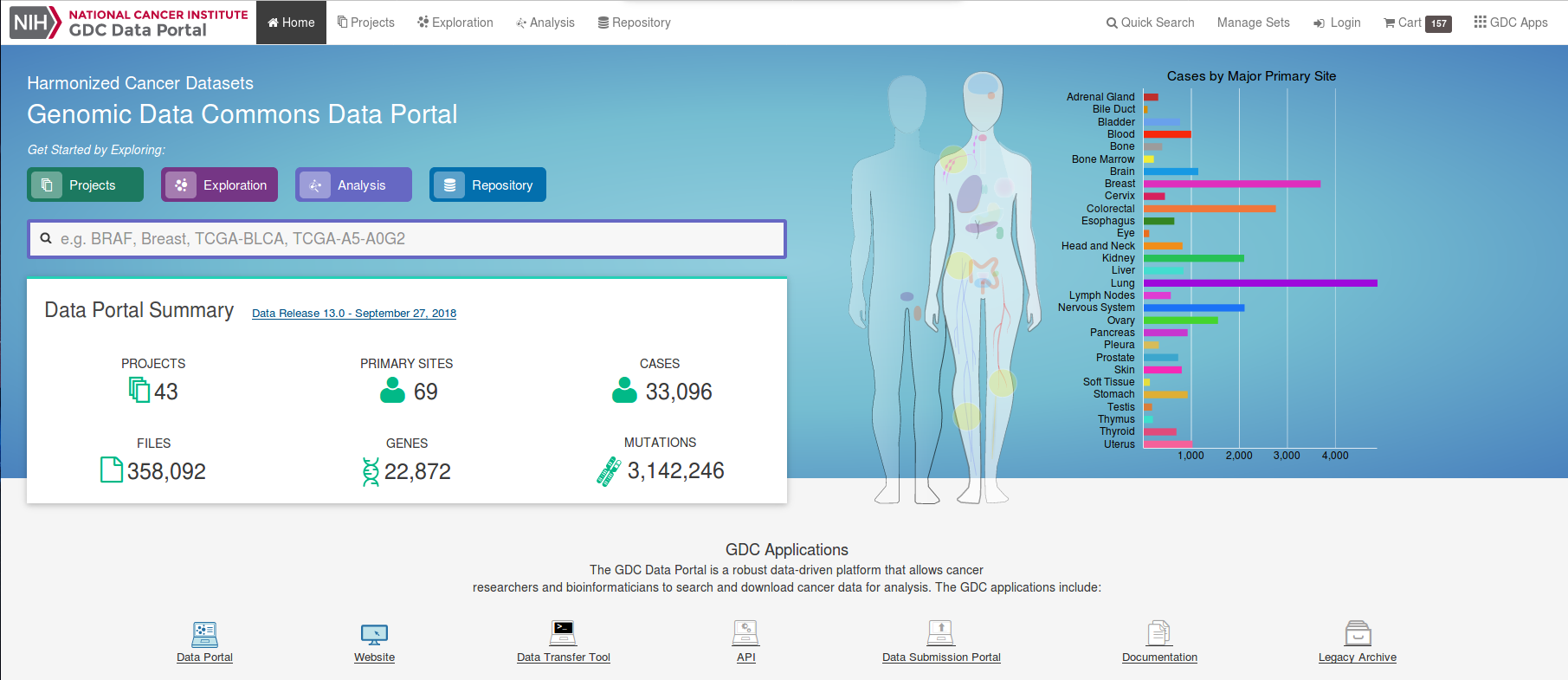
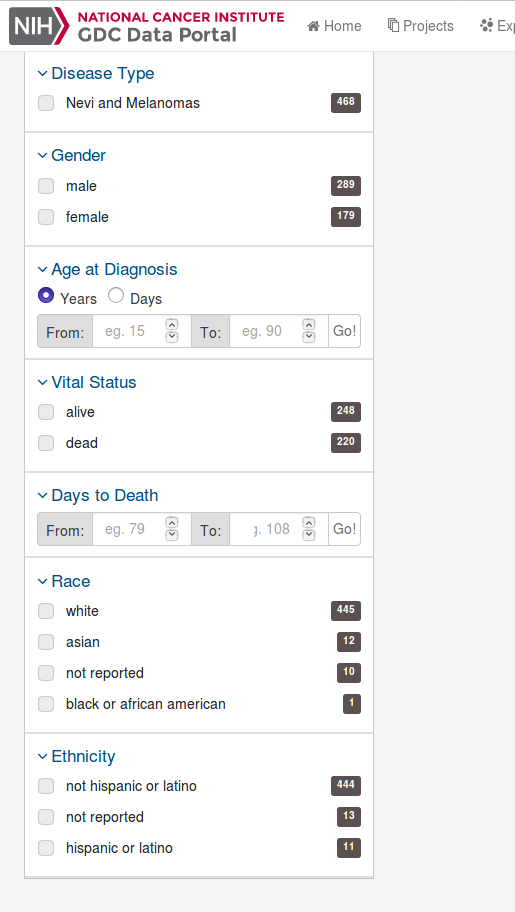
43種類のプロジェクトで、69種の腫瘍のデータが収録されていることがわかる。検体の総数は、33096人分だ。
-
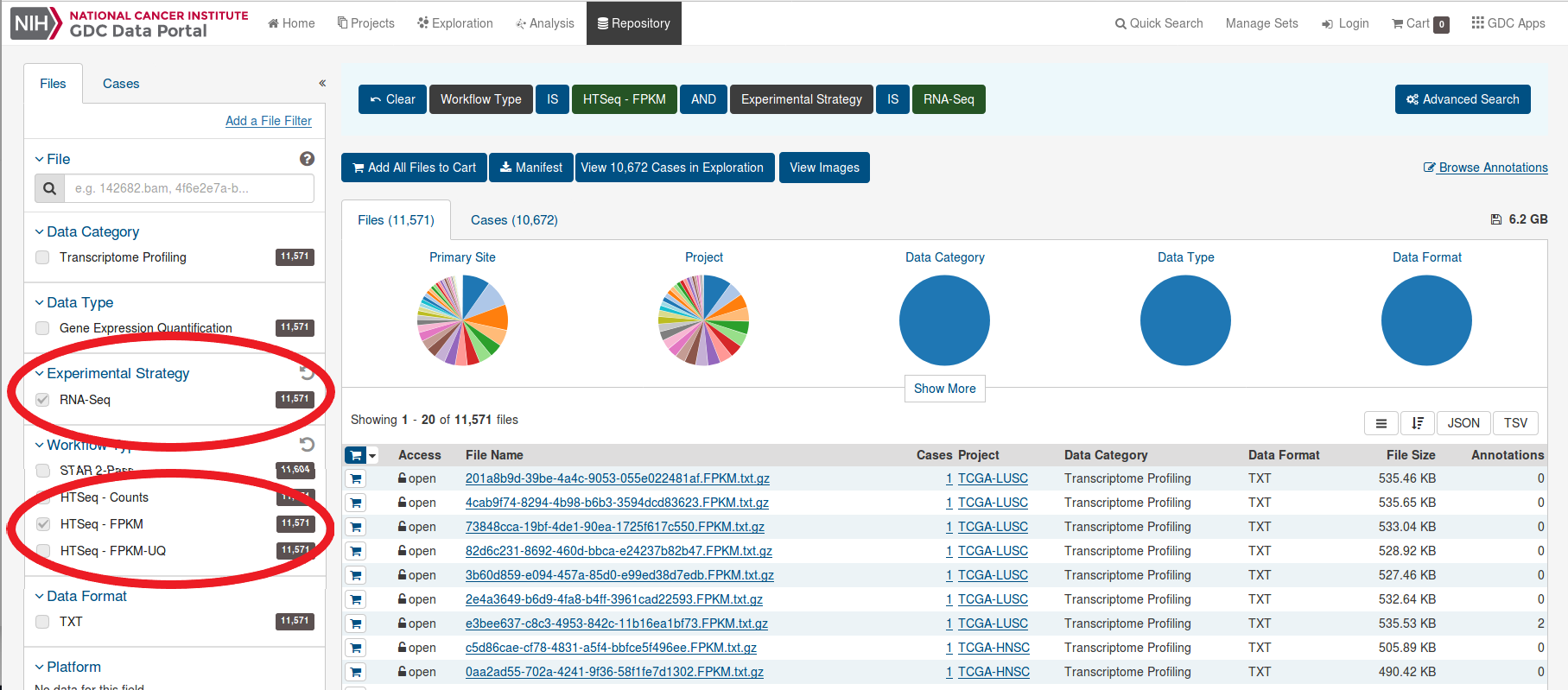
左側の「Files」タブで入手したいファイルのカテゴリを選択する。
-
右側の「Cases」タブで、入手するファイルの細目を決定する
-
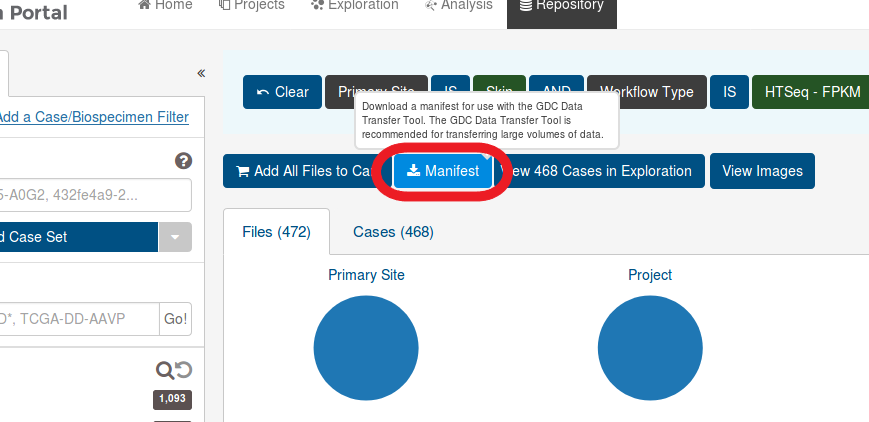
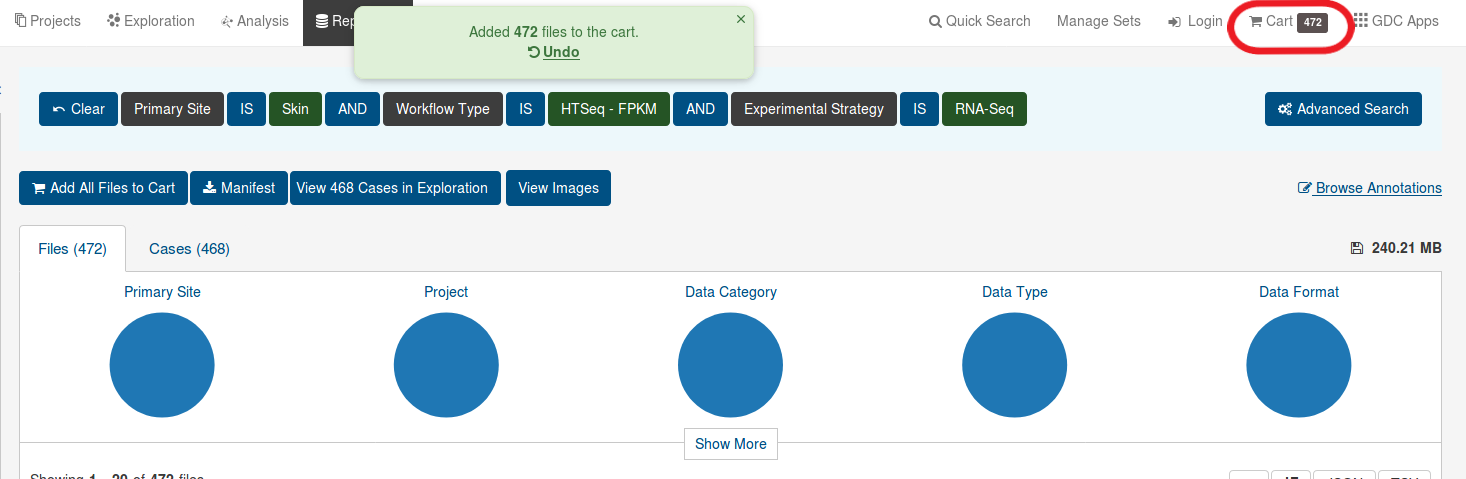
これで、発現情報を入手するための手はずが整った。ブラウザの右下には、これからダウンロードするファイルの一部が表示される。
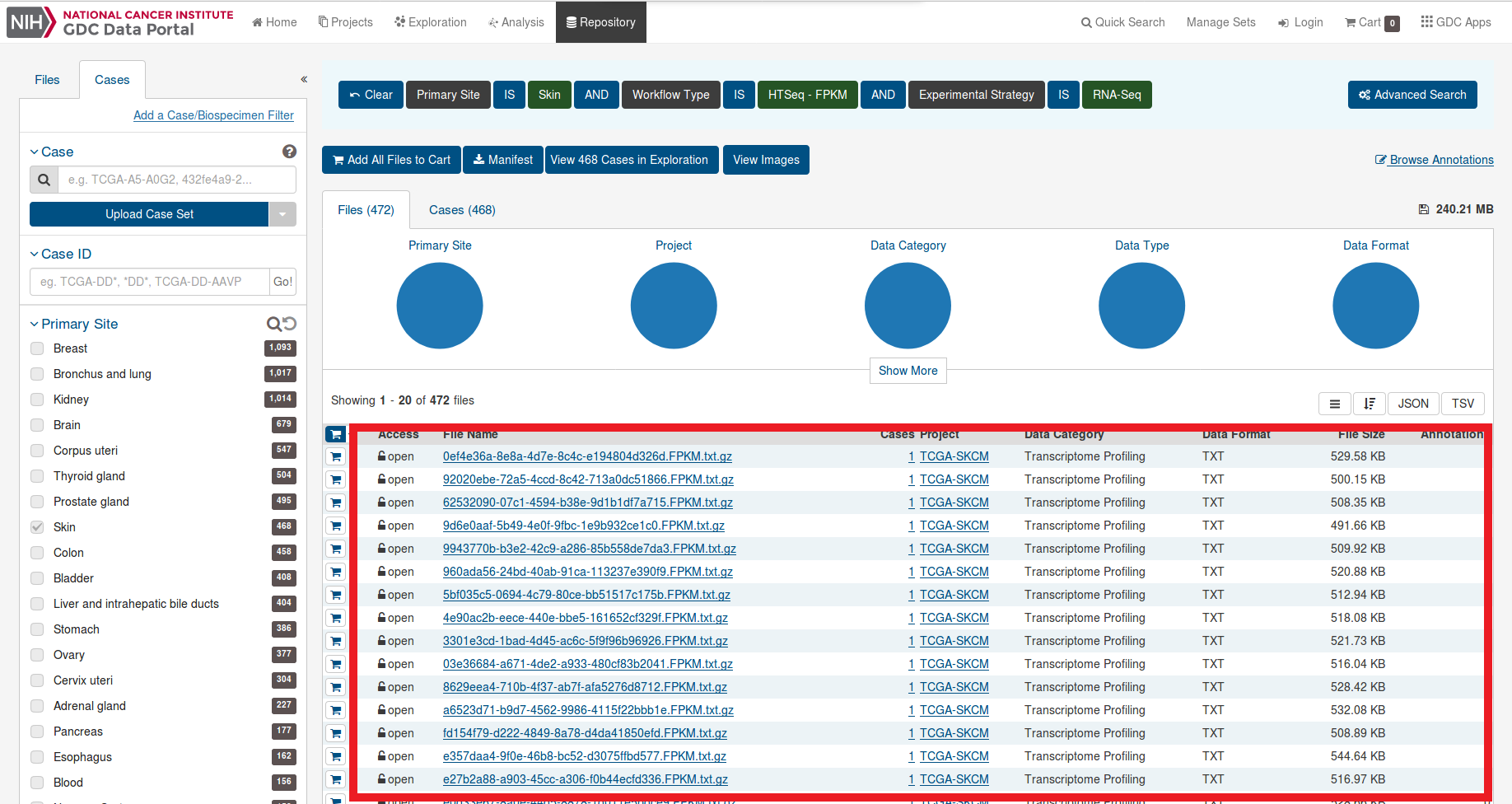
-
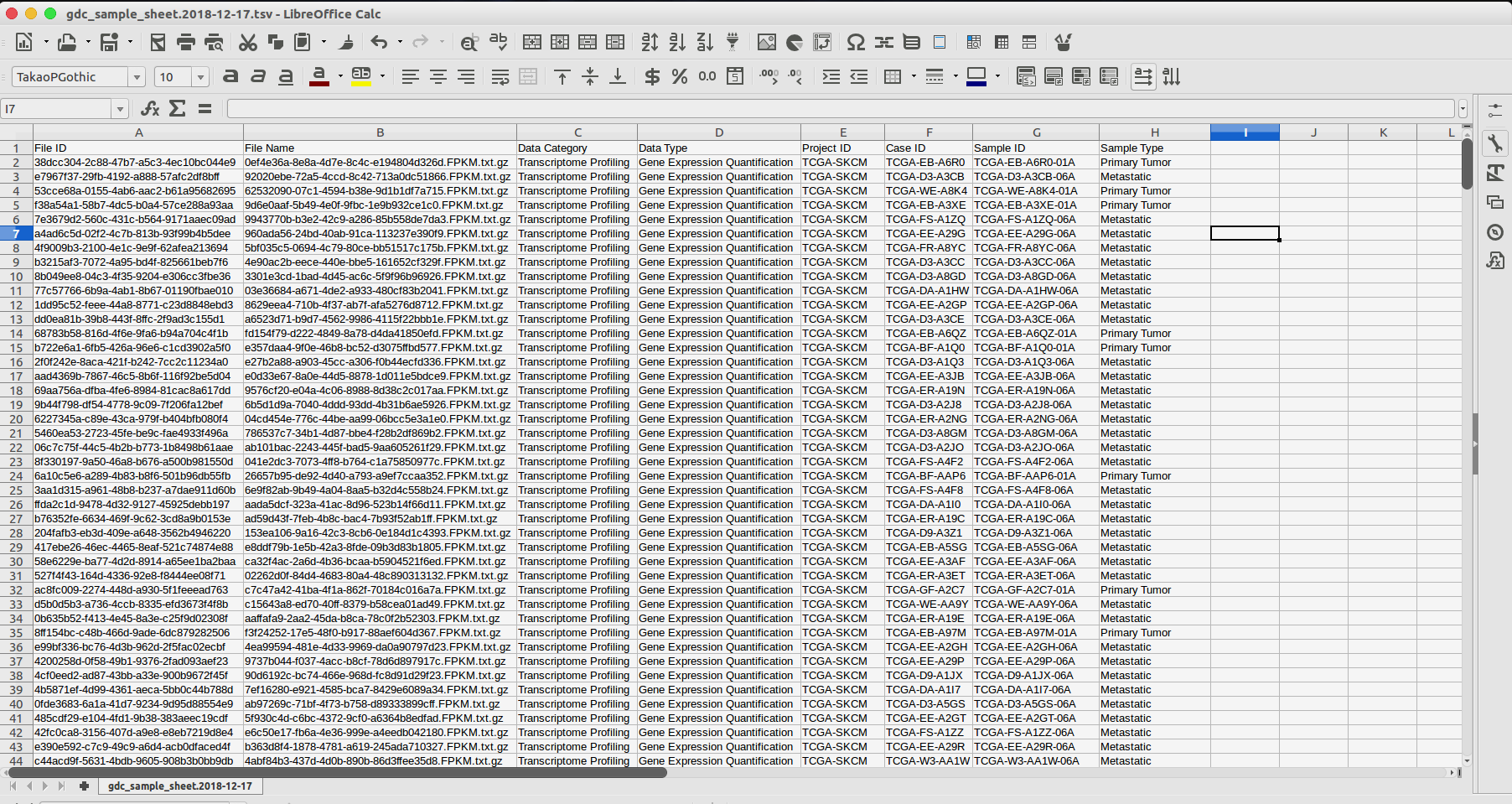
マニフェストファイルの中身はこのような感じ。
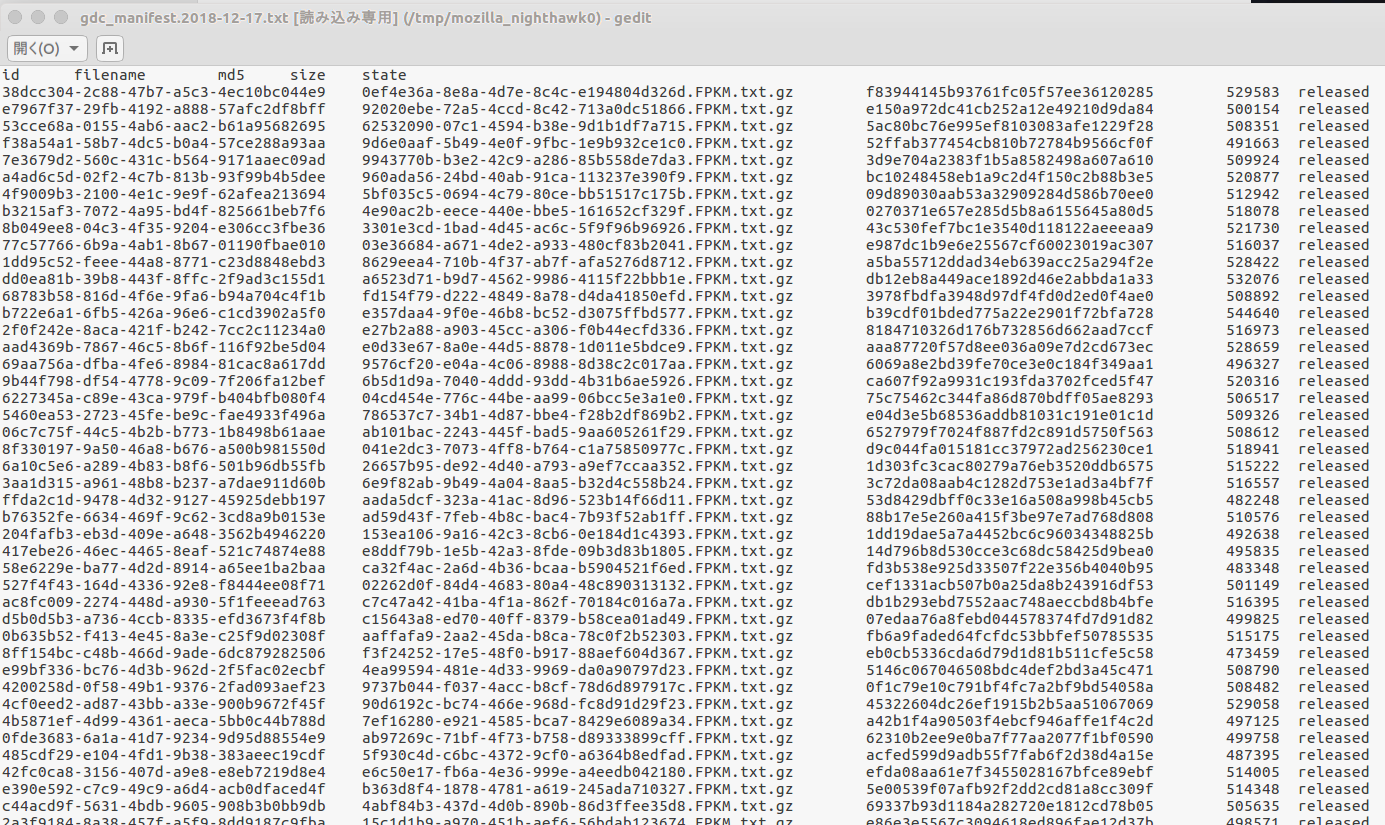
アッセイID、ファイル名、MD5、サイズ等がリストされている。- 各ファイルには、TCGAのID(TCGA-xx-xxxx)ではなく、個別のアッセイID(xxxxxxxx-xxxx-xxxx-xxxxxxxx)がファイル名として付けられていて、TCGAのIDはマニフェストファイルからは判らない。
- 単にFPKMの情報をダウンロードするだけでは、この発現情報が原発巣由来なのか、転移巣由来なのか、はたまた正常組織なのかすらも不明だ。このため、アッセイIDとTCGA IDが共に記載されているファイルが別途必要だ。
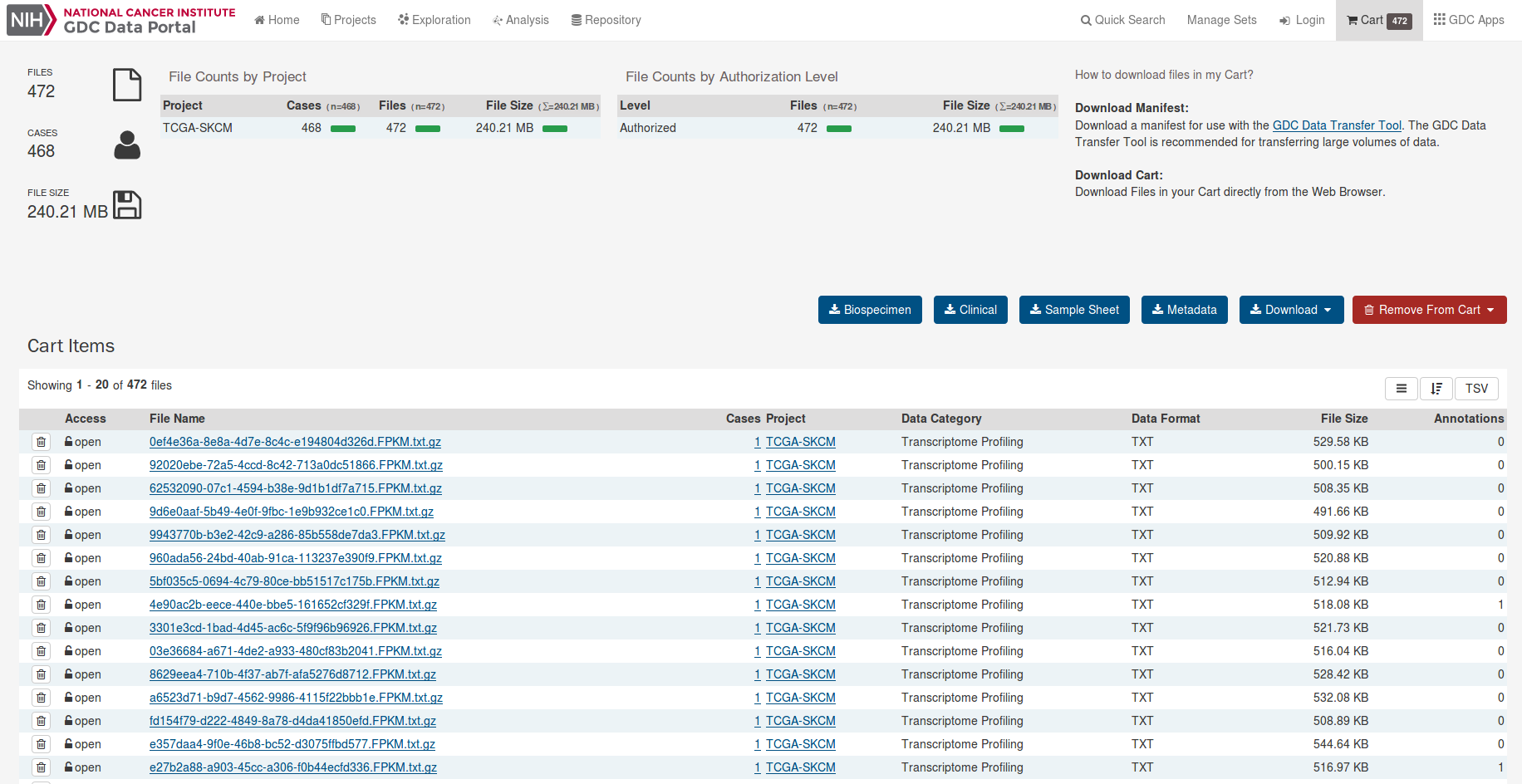
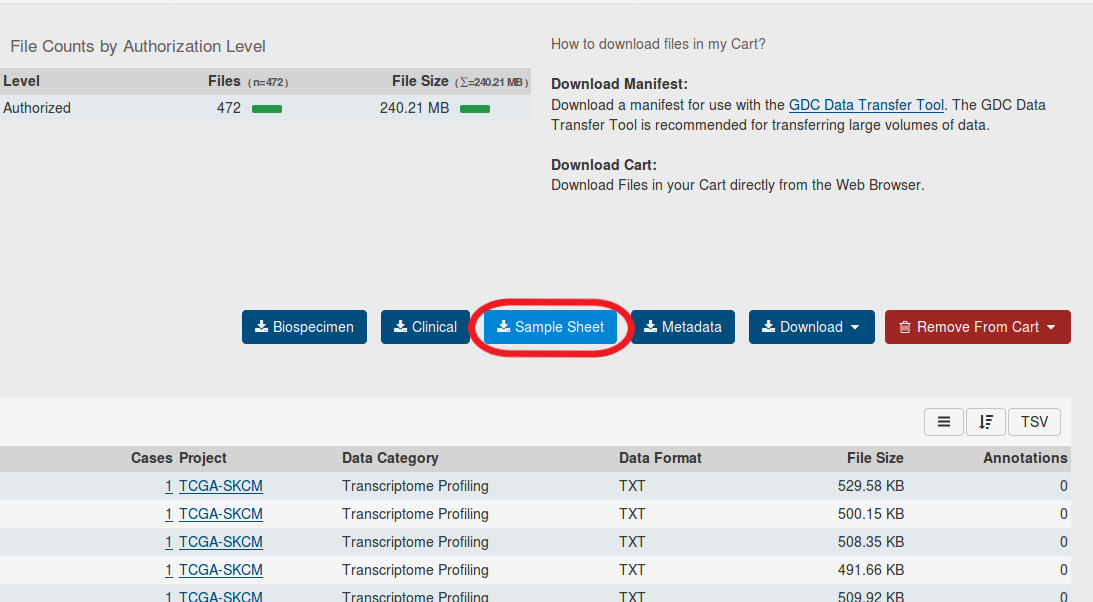
####サンプルシートの入手
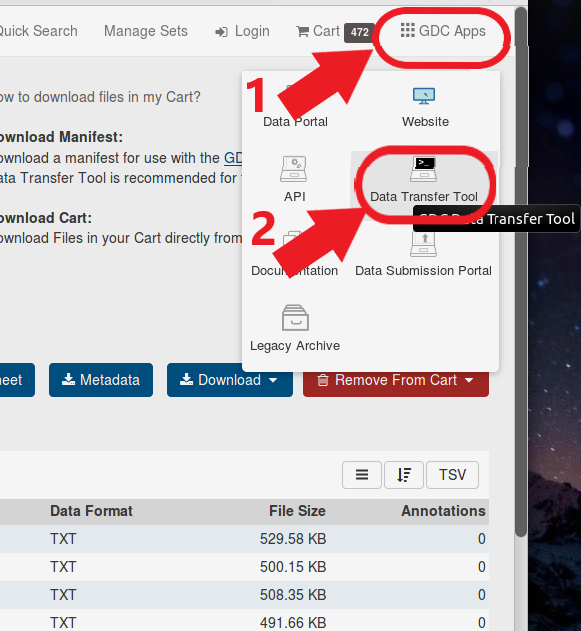
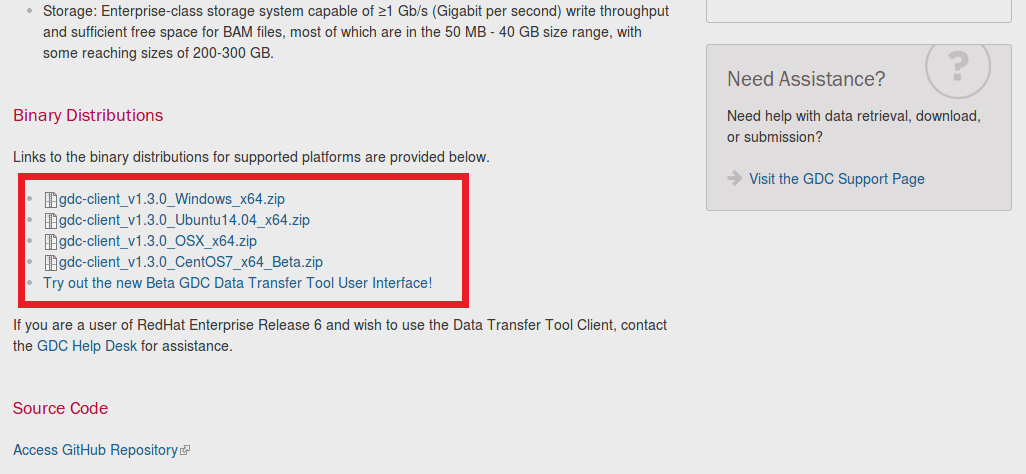
####GDC-clientの入手
####2019/12/24 追記:2019年12月現在、Data Transfer Toolはversion1.4.0が配布されています。Linux用はUbuntuのみとなり、CentOS用の配布がなくなった模様。
####2020/9/4 追記:2020年9月現在、Data Transfer Toolはversion1.6.0が配布されています。Python2のサポート終了に伴い、Python3に対応したツールとなっています。
####2023/2/22 追記:2023年2月現在、Data Transfer Toolはversion1.6.1が配布されています。
-
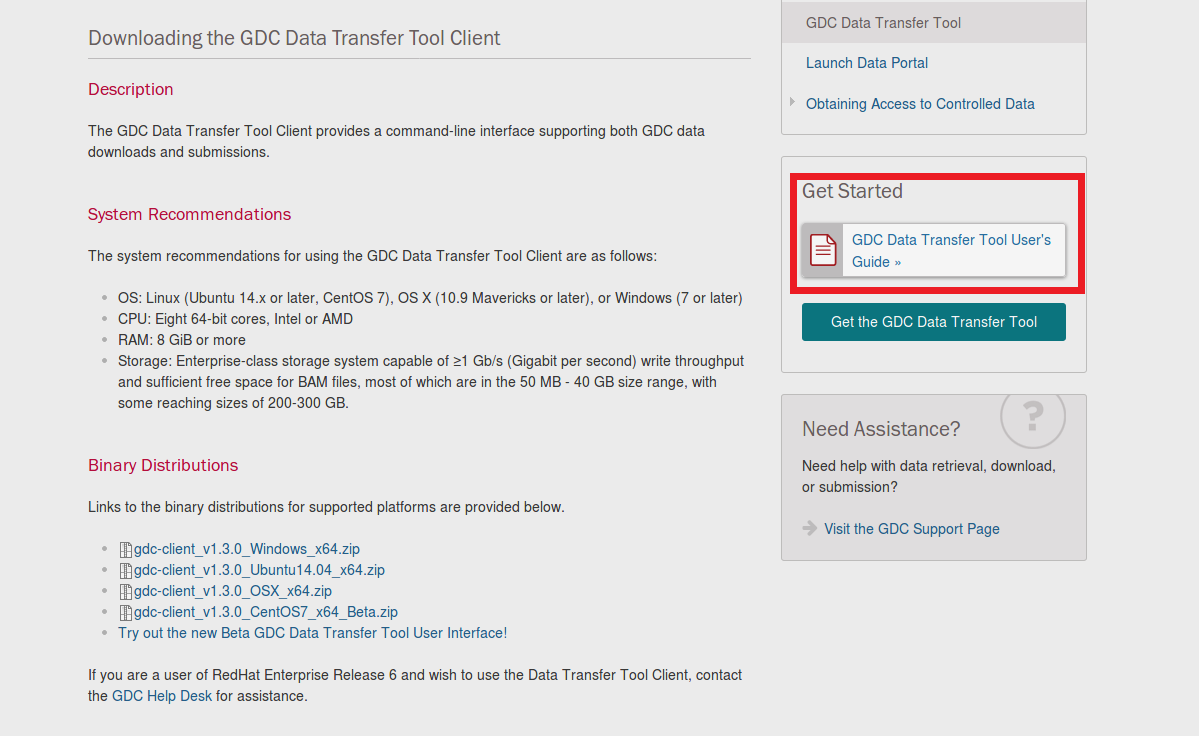
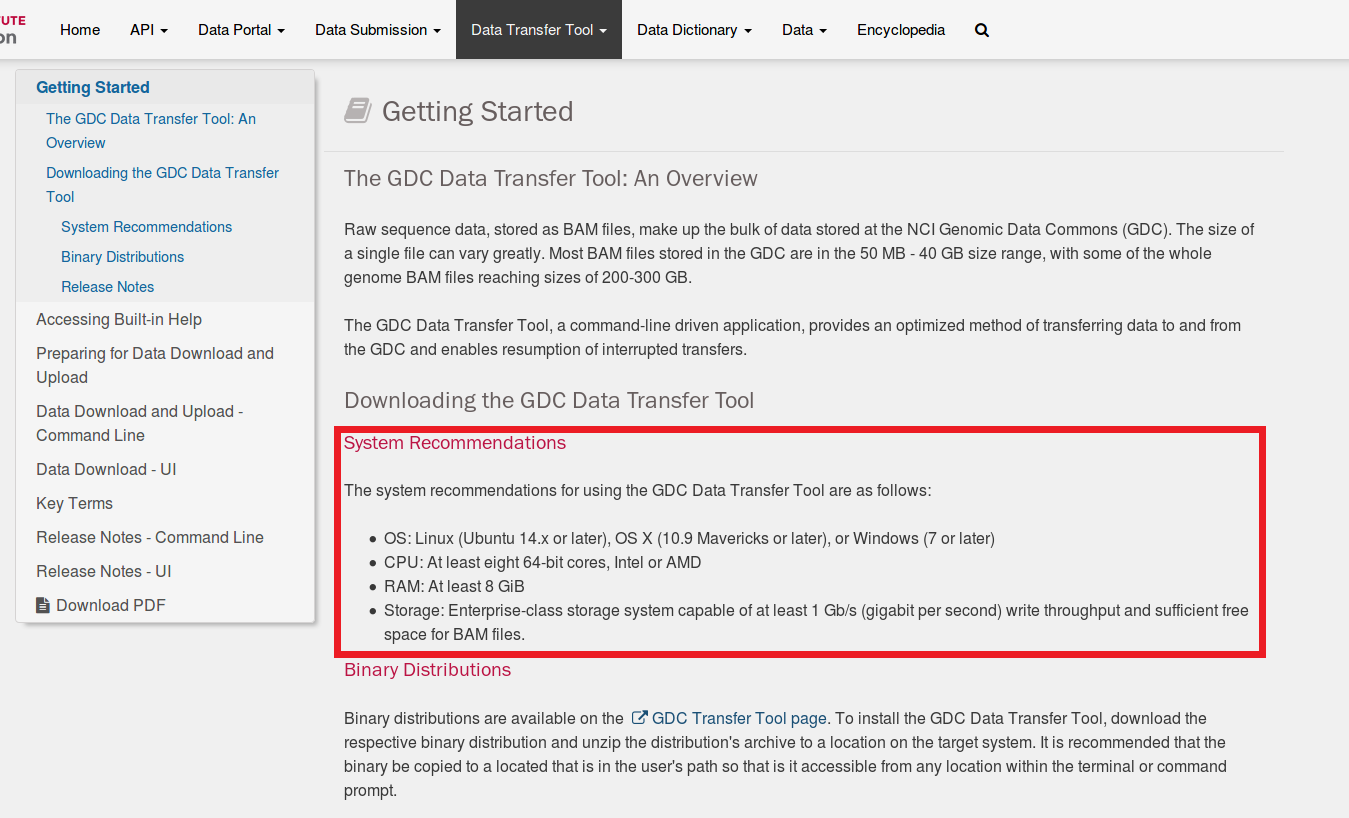
Data Transfer Toolを使うにあたって要求されるマシンスペックはご覧の通り。今回はBAMファイルを入手するわけではないので、ストレージ容量はさほど気にしない。
-
Tranfer toolの詳細な利用方法は、
-
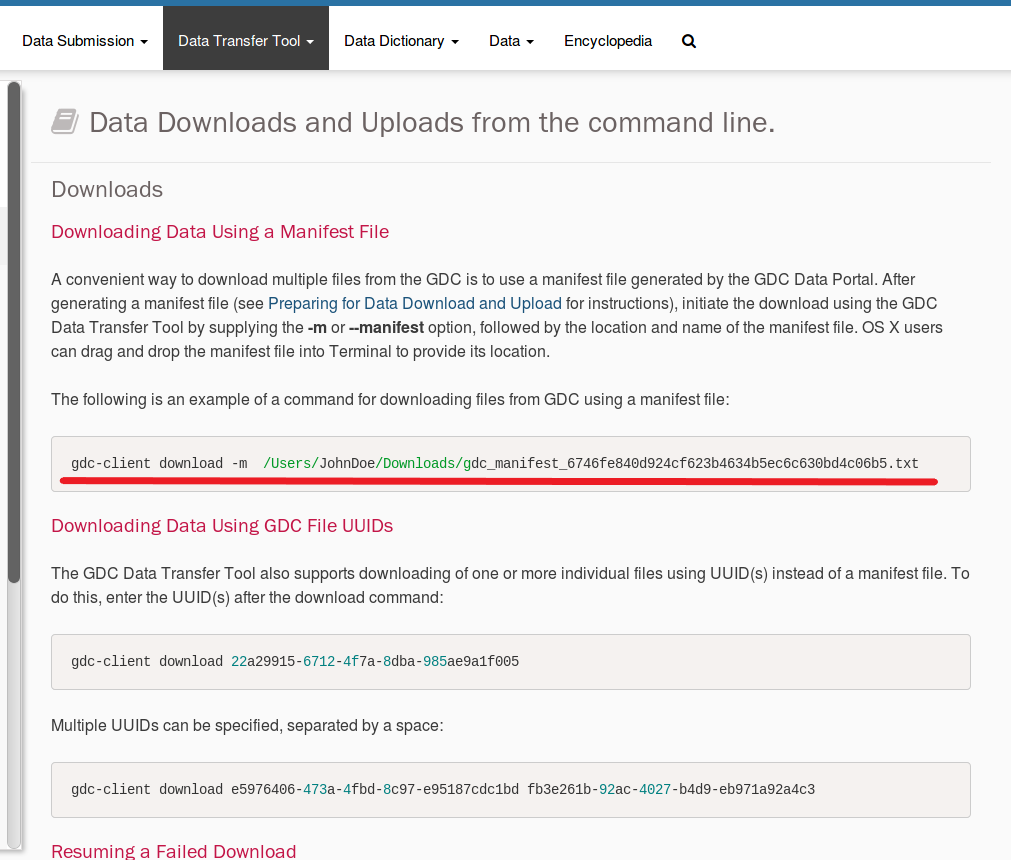
で、「Downloading Data Using a Manifest File」の項目の枠内にあるコマンドラインが、今回使うもの。
####ダウンロードの開始
道具が揃ったので、ようやくファイルのダウンロードに移れる。
- 適当にファイルをダウンロードするためのフォルダを作成。「TCGA-SKCM」とでもしておこう。
mkdir TCGA-SKCM
で、先に入手したマニフェストファイル、gdc-clientファイル、サンプルシートをこのフォルダに移動しておく。
ターミナルでこの作業フォルダの中身をみると、下記のような状態になっている。
$ls -l
-rwxr-xr-x 1 hoge hoge 24647040 8月 14 2017 gdc-client
-rw-rw-r-- 1 hoge hoge 63747 12月 3 14:09 gdc_manifest.2018-12-03.txt
-rw-rw-r-- 1 hoge hoge 91036 12月 17 19:45 gdc_sample_sheet.2018-12-17.tsv
役者はそろった。いよいよ実行だ。
./gdc-client download -m ./gdc_manifest.2018-12-03.txt
やがてダウンロードが始まり
100% [############################################################################################################################] Time: 0:00:01 253.30 kB/s
100% [############################################################################################################################] Time: 0:00:02 233.15 kB/s
100% [############################################################################################################################] Time: 0:00:02 235.66 kB/s
100% [############################################################################################################################] Time: 0:00:02 245.85 kB/s
................
100% [############################################################################################################################] Time: 0:00:02 225.63 kB/s
100% [############################################################################################################################] Time: 0:00:02 244.29 kB/s
100% [############################################################################################################################] Time: 0:00:02 210.72 kB/s
100% [############################################################################################################################] Time: 0:00:02 232.12 kB/s
Successfully downloaded: 472
目出度く472個のファイルのダウンロードが完了する。
- TCGAからDLした解析ファイルは、すべて個別のフォルダに入った状態で解凍される。今回は、472検体分のファイルを落としたので、フォルダが472個出来ていればひとまず作業は成功したことになります。