はじめに
システムの稼働状況とかミドルウェアの統計情報とかを手っ取り早く可視化するためにRを使ってみようということで書き始めた連載企画です。今回はプログラミング編です。
インフラ屋さんがレポートをサクッとRで作るためにはこの辺知っておくといいよ、という辺りをまとめてます。
(最終的には、csvファイルとかRDBとかの情報を引っ張ってきて、表/グラフで可視化し、きれいなレポートを作る!というのが目標です。)
関連記事
インフラ屋さんのためのR言語: 環境構築編
オフラインでのR環境構築 on RHEL
z/OSにRを導入してみた
インフラ屋さんのためのR言語: プログラミング編 <= 当記事
R Markdownによるレポート生成
R MarkdownのHTMLレポートをブラッシュアップ
R - ShinyによるWebアプリケーション作成: 基礎編
R - ShinyによるWebアプリケーション作成: shinydashboard編
R - ShinyによるWebアプリケーション作成Tips: shinydashboardでの画面遷移制御
R - ShinyによるWebアプリケーション作成Tips: UIオブジェクトの動的制御
R - Shinyアプリ/管理サーバー テンプレート
R - ShinyアプリでJリーグの勝点推移グラフを作成してみた
R基本操作
まずは軽く入門的にベースになる部分を紹介します。
ベクトル
統計分析とか慣れてないので最初戸惑うのが「ベクトル」(vector)としての操作。ベクトルというのはRで扱えるデータ型の1つで、「同じ型の要素を集めたもの」です。
例えば、(1, 2, 3, 4, 5)という数値の集合とか、("AAA","BBB","CCC","DDD")といった文字列の集合などです。異なる型を扱えるListというのもありますが、まぁこういう風に可変の配列みたいなものをベクトルとして扱うのが非常にやりやすいです。
Excelの"列"単位でデータを扱うイメージです。
c()という関数?演算子?でベクトルが作れます。
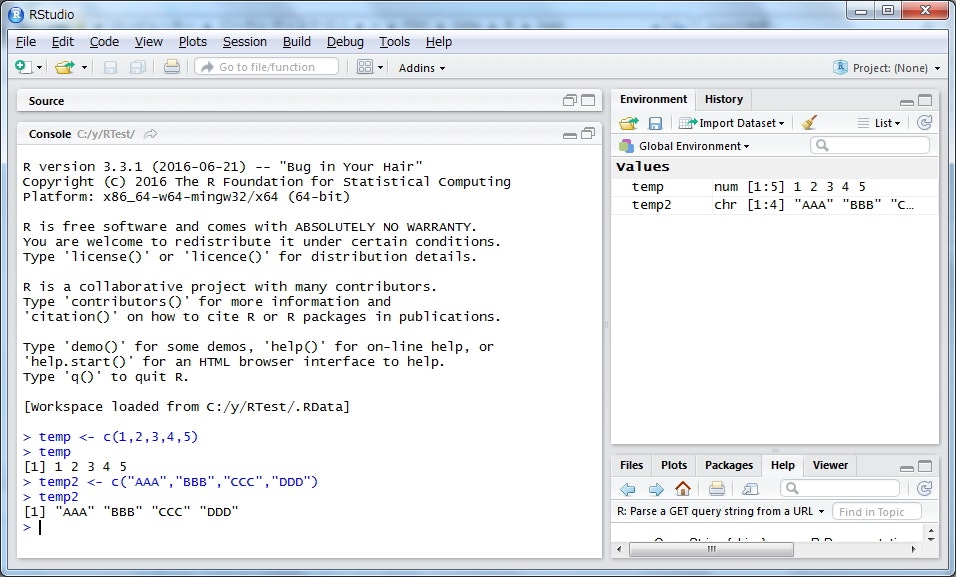
コンソールで操作しているイメージはこちら。
> temp <- c(1,2,3,4,5)
> temp
[1] 1 2 3 4 5
> temp2 <- c("AAA","BBB","CCC","DDD")
> temp2
[1] "AAA" "BBB" "CCC" "DDD"
ちなみに変数への代入は「=」も使えますが慣例的には「<-」記号を使うことが多いようです。
RStudioの画面イメージはこちら。
ベクトルに対する演算がすごく楽なのが特徴です。例えば、上のtempという変数に対して演算を行うと、各要素それぞれに対する演算結果が得られます。
> temp * 2
[1] 2 4 6 8 10
data.frame(データフレーム)
data.frame(データフレーム)というのはEXCELの"表"のようなイメージでデータを扱うことができる型です。行と列を持ちます。
> Name <- c("Taro", "Jiro", "Saburo") #Name列の定義
> Age <- c(35, 32, 21) #Age列の定義
> Address <- c("Tokyo","Chiba","Kanagawa") #Address列の定義
> NameList <- data.frame(Name, Age, Address) #data.frame作成
> NameList #中身確認
Name Age Address
1 Taro 35 Tokyo
2 Jiro 32 Chiba
3 Saburo 21 Kanagawa
Name, Age, Addressというベクトル型の変数を用意し、それぞれ値を設定しています。Excelの「列」を作っているイメージです。
それらを合わせて、NameListというデータフレームを作成しています。すなわち「表」のイメージのデータとなります。
このデータフレームの操作が肝です。この操作は後ほど詳しく整理します。
文字列操作
文字列表記
ダブルクォーテーション(")orシングルクォーテーション(')で括ります。
ダブルクォーテーションorシングルクォーテーションのどちらかを含む文字列を扱いたい場合は使わない方の記号を使って括るか、バックスラッシュでエスケープする必要があります。
> strTest <- "abcabc"
> strTest
[1] "abcabc"
> strTest <- 'abc"abc"abc'
> strTest
[1] "abc\"abc\"abc"
> strTest <- "abc'abc'abc"
> strTest
[1] "abc'abc'abc"
置換
sub関数/gsub関数で文字列置換が行えます。
以下、strTestに含まれる文字列のうち、"abc"=>"XXX"に置換する例です。
sub関数は最初に条件に合致するもののみ置換、gsub関数は全ての条件に合致するものを置換する関数です。
> strTest <- "abcabc"
> sub("abc","XXX", strTest)
[1] "XXXabc"
> vectorTest <- c("abcabc", "aaabccc", "abcccabc")
> sub("abc","xxx",vectorTest)
[1] "xxxabc" "aaxxxcc" "xxxccabc"
連結
paste関数/paste0関数で文字列を連携できます。paste関数はsepオプションで区切り文字を指定できます(デフォルトはブランク区切り)。
> strTest1 <- "aaa"
> strTest2 <- "bbb"
> paste(strTest1, strTest2, "ccc")
[1] "aaa bbb ccc"
> paste(strTest1, strTest2, "ccc", sep=",")
[1] "aaa,bbb,ccc"
> paste0(strTest1, strTest2, "ccc")
[1] "aaabbbccc"
分割
strsplit関数で、指定した区切り文字で文字列を分割することができます。区切り文字は正規表現が使えます。結果はList型のオブジェクトとして返されます。
> strTest <- "aaa=111,bbb=222,ccc=333"
> strsplit(strTest, ",")
[[1]]
[1] "aaa=111" "bbb=222" "ccc=333"
> strsplit(strTest, ",|=")
[[1]]
[1] "aaa" "111" "bbb" "222" "ccc" "333"
検知
str_detect関数で、特定の文字列が含まれているかどうかを判断することができます。検索対象の文字列指定には正規表現が使用できます。結果は論理値型(TRUE or FALSE)で返されます。str_detect関数はベースのパッケージではなくstringrというパッケージで提供される関数ですので、無ければパッケージをInstallした後、library(stringr)でロードしてから使ってください。
> library(stringr)
> strTest1 <- "abcabc"
> strTest2 <- "XXXXXX"
> str_detect(strTest1, "abc")
[1] TRUE
> str_detect(strTest2, "abc")
[1] FALSE
フロー制御
if文による条件分岐
まぁこんな感じです。
if (rc <= 0) {
strResult <- "OK"
} else if( (0 < rc) & (rc <= 4)) {
strResult <- "application error"
} else if (rc > 4) {
strResult <- "system error"
}
論理積/論理和は、「&」,「&&」/「|」,「||」 というようにそれぞれ2パターンあります。
ベクトルに対して論理演算する場合、「&」/「|」はベクトルのそれぞれの要素に対して演算されますが、「&&」/「||」はベクトルの最初の要素のみ演算するらしいです。意識して使うシチュエーションにであったこと無いけど気になったので一応参考までに...
> b1 <- c(TRUE,FALSE,FALSE)
> b2 <- c(TRUE,TRUE,FALSE)
> b1 & b2
[1] TRUE FALSE FALSE
> b1 && b2
[1] TRUE
forループ
for文の例です。
iの値を1~10までカウントアップして繰り返します。ここでは1~10まで単純に足し算してます。
x <- 0
for (i in 1:10){
x <- x + i
}
Rの繰り返し処理はパフォーマンスがよろしくないようなので、ベクトル演算できるならその方がよいようです(列の合計を計算させるとか)。なのでfor文で上のように単純に計算させるということはあまりしないと思います。あくまで単純な例として使ってます。
whileによるループも使えますが、同様に遅いらしいので利用は必要最小限にするのがよいでしょう。
関数定義
共通ロジックは関数として定義した方が何かと便利です。
以下、単純に2つの引数を足し算して結果を返す関数の例です。
funcAdd <- function(x,y){
result <- x + y
return(result)
}
> funcAdd(1,2)
[1] 3
> funcAdd(10,30)
[1] 40
エラーハンドリング
tryCatchで例外をハンドリングすることができます。
funcTest01 <- function(x){
if (x == 0) {
return("OK")
} else if(x > 0) {
strMessage <- paste0("Warning! x=", x)
warning(strMessage) # issue warning
} else {
strMessage <- paste0("Error! x=", x)
stop(strMessage) # issue error
}
}
funcTest02 <- function(y) {
strResult <- ""
strMessage <- ""
tryCatch({
strMessage <- funcTest01(y)
}, warning = function(w){
strMessage <<- w$message
}, error = function(e) {
strMessage <<- e$message
}, finally = {
strResult <- paste(Sys.time(), strMessage)
})
return(strResult)
}
funcTest01は、意図的にwarning, errorを発行させるための関数です。
funcTest02は、funcTest01を実行し、そこで発生したwarning,errorをハンドリングするサンプルコードです。
> funcTest02(-1)
[1] "2016-09-27 21:39:18 Error! x=-1"
> funcTest02(2)
[1] "2016-09-27 21:39:22 Warning! x=2"
> funcTest02(0)
[1] "2016-09-27 21:39:29 OK"
補足:
funcTest02内で、warning, errorのハンドリングを行っている箇所で、変数への代入文に <-ではなく<<-を使っている箇所があります。これは変数のスコープに関連します。前者はローカル変数への代入、後者はグローバル変数への代入を意味します。warning, errorハンドリングを行う部分は関数になっているので変数のスコープが他とは異なります。そこではグローバル変数の値を変更したいので、<<-を使っています。
その他気になる表記
関数のドット表記について
data.frameなど、関数名の間にドットが含まれているものがあります。Javaとかに慣れていると、パッケージ名のように何か階層になってるのかな?とか、dataクラスのframe変数みたいな意味かな?と思ってしまうのですが、Rの場合あまりそういう深い構造的な意味は無いようです。
as.integer,as.character,etcとか、is.null,is.numeric,etcなど、似たような関数はドットで区切られて同じ接頭語がついていたりするものもあるんですけどね。
関数名の一部としてたまたまドット記号が使われているという捕らえ方でよさそうです。
::(コロンx2)
base::mergeといったように、::が使用されるケースがあります。これは、<パッケージ名>::<関数名> というように使用します。Rでは様々なパッケージが提供されていますが、同一の関数名が別のパッケージで提供されることがあります。そのような場合にどちらのパッケージの関数を使用するか明示的に指定するのにこのような表記を使うようです。
同一関数を含む複数のパッケージをロードしている状況で、パッケージ名指定しない場合にどういうルールでどちらが使われるのかはよく分かりません。分かりやすさのためにも名前がかぶるものは明示指定しておくのがよいでしょう。
ちなみに<パッケージ名>::<関数名>という記述をすると、事前にそのパッケージをロードしていなくても関数実行できるようです。
ファイル入出力
さて、ここからがやりたいことの本題に深く関わってくる所です。
(csv形式のファイルを読んで、グラフなどで可視化する、ということを想定しています。)
ファイル読み込み
大量データを扱う場合ファイル読み込みの処理時間がバカにならないことが多いです。
read.csvやread.tableというのが標準ライブラリに含まれていますが、この辺りはパフォーマンスに優れたものがいくつか別のパッケージとして提供されているものがあるのでなるべくそれらを使ったほうがよさそうです。ざっと世の中の評価を見てみると、「data.table」というパッケージに含まれるfread()という関数を使うのが良さそうなので、ここではそれを使うことにします。
> system("cat NameList.csv") #ファイルの中身確認
名前,年齢,住所
太郎,35,東京
次郎,32,千葉
三郎,21,神奈川
> library(data.table) #freadを使用するためdata.tableパッケージのロード
data.table 1.9.6 For help type ?data.table or https://github.com/Rdatatable/data.table/wiki
The fastest way to learn (by data.table authors): https://www.datacamp.com/courses/data-analysis-the-data-table-way
> dfNameList <- fread("NameList.csv",data.table=FALSE) #csvファイル読み込み
> dfNameList #読み込んだ中身の確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> str(dfNameList) #作成されたオブジェクトの構造確認
'data.frame': 3 obs. of 3 variables:
$ 名前: chr "太郎" "次郎" "三郎"
$ 年齢: int 35 32 21
$ 住所: chr "東京" "千葉" "神奈川"
fread()関数を使用することで、csvファイルをdata.frame型のオブジェクトとして取り込むことができます。あとはこれをR上でいろいろ加工することになります。
※補足:
system()関数ではシステムコマンドを実行することができます。上の例ではcatでファイルの中身を確認しています。
str()関数ではオブジェクトの構造を確認することができます。"表"の形式の構造を扱うために、data.frameという型がありますが、それをさらに拡張したものとしてdata.tableという型が提供されています。fread()関数はそのdata.table用に提供されているものなので、デフォルトでは作成されたオブジェクトはdata.table として作成されます。data.tableは高機能ではありますが、扱う作法が若干data.frameとは異なるようです。一般的にはdata.frameの方が普及しておりノウハウも容易に手に入るので、ここではdata.frameとしてデータを扱うことにします(data.table=FALSEを指定することで、data.frame型のオブジェクトが作成されます)。
fread()関数はオプションで各種指定ができます(区切り文字の指定、1行目をヘッダーではなくデータ行として扱う、読み込む行数指定、など).
> system("cat NameList2.csv")
名前:年齢:住所
太郎:35:東京
次郎:32:千葉
三郎:21:神奈川
> dfNameList2 <- fread("NameList2.csv", data.table=FALSE, sep=":", header=FALSE, nrows=3)
> dfNameList2
V1 V2 V3
1 名前 年齢 住所
2 太郎 35 東京
> str(dfNameList2)
'data.frame': 3 obs. of 3 variables:
$ V1: chr "名前" "太郎" "次郎"
$ V2: chr "年齢" "35" "32"
$ V3: chr "住所" "東京" "千葉"
ファイル書き出し
まぁファイルとして書き出すのは集計結果(合計とか平均とか)くらいの想定なので、標準のwrite.tableやwrite.csvを使えばよいでしょう。
> write.table(dfNameList, "NameListOut.txt") # ブランク区切りでのデータ書き出し
> system("cat NameListOut.txt")
"名前" "年齢" "住所"
"1" "太郎" 35 "東京"
"2" "次郎" 32 "千葉"
"3" "三郎" 21 "神奈川"
> write.table(dfNameList, "NameListOut2.csv", row.names=FALSE, col.names=FALSE, sep=",") # カンマ区切りでのデータ書き出し(カラム名、行名なし)
> system("cat NameListOut2.csv")
"太郎",35,"東京"
"次郎",32,"千葉"
"三郎",21,"神奈川"
data.frame操作
一旦ファイルからdata.frameにデータを読み込んだら、これをこねくり回して必要な情報を集計したりしたいですよね。
あるいは、きれいなグラフを書きたいと思った場合、グラフを書くためのパッケージというのが提供されていますが、利用するパッケージやグラフの種類によって、どういう形式でデータを与えなければいけないかが変わってきます。
そのため、data.frameの情報を目的に合わせて色々と操作する必要があります。出来ることは山のようにありますが、ここではまずはこの辺抑えておくといいですよ、というのを取り上げます。細かいところは必要に迫られた時に調べればよいでしょう。
行番号、列番号の指定
表の行番号、列番号を指定して必要な部分だけ抜き出すことができます。
> dfNameList # 内容確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList[1,3] # 1行目、3列目のデータ抽出
[1] "東京"
> dfNameList[1,] # 1行目の全データ抽出
名前 年齢 住所
1 太郎 35 東京
> dfNameList[,3] # 3列目の全データ抽出
[1] "東京" "千葉" "神奈川"
> dfNameList[1:2,] # 1~2行目の全データ抽出
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
> dfNameList[,2:3] # 2~3列目の全データ抽出
年齢 住所
1 35 東京
2 32 千葉
3 21 神奈川
[行,列]で指定した範囲を抽出可能です。行/列を省略するとそれぞれ全行/全列を扱うということになります。
列名を指定した操作
列番号ではなく列名を使用してデータを扱うことができます。
> dfNameList$名前 # 名前列の抽出
[1] "太郎" "次郎" "三郎"
> dfNameList$住所 # 住所列の抽出
[1] "東京" "千葉" "神奈川"
> dfNameList$住所[2] # 住所列のうち2つめの要素抽出
[1] "千葉"
> dfNameList[,c("住所","名前")] #住所列および名前列の抽出
住所 名前
1 東京 太郎
2 千葉 次郎
3 神奈川 三郎
条件に合うデータの抽出
条件を指定して、その条件に合う行のみ抽出するということができます。Excelで特定の表にフィルターをかけるイメージですかね。
> dfNameList # 内容確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList[dfNameList$年齢>=30, ] # 年齢が30以上の行を抽出
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
> dfNameList[dfNameList$年齢>=30, c("名前","年齢")] # 年齢が30以上の行で、名前列と年齢列のみ抽出
名前 年齢
1 太郎 35
2 次郎 32
> dfNameList[(dfNameList$年齢>=30 & dfNameList$住所=="東京"),] # 年齢が30以上、かつ、住所が東京の行を抽出
名前 年齢 住所
1 太郎 35 東京
[行条件,列条件]を指定して、合致するものが抽出されるというイメージです。
もう1つ、subset()という関数を使った方法もあります。こちらの方が分かりやすいかも。
> dfNameList # 内容確認
名前 年齢 住所
1: 太郎 35 東京
2: 次郎 32 千葉
3: 三郎 21 神奈川
> subset(dfNameList, (dfNameList$年齢>=30 & dfNameList$住所=="東京")) # 年齢が30以上、かつ、住所が東京の行を抽出
名前 年齢 住所
1: 太郎 35 東京
並べ替え/ソート
行のソート
data.frame()のソートは最初ちょっと分かりにくいです。
> dfNameList
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> order(dfNameList$年齢) #order関数で年齢でソート(昇順)した順番を確認すると...
[1] 3 2 1
> dfNameList <- dfNameList[order(dfNameList$年齢),] #order関数の順番で行をアクセスし直し
> dfNameList # 結果確認
名前 年齢 住所
3 三郎 21 神奈川
2 次郎 32 千葉
1 太郎 35 東京
> rownames(dfNameList) <- NULL # 行番号をリセット
> dfNameList
名前 年齢 住所
1 三郎 21 神奈川
2 次郎 32 千葉
3 太郎 35 東京
order()関数を使うと、特定のベクトルの値をソートした時の並び順を返してくれます。上の例だと、年齢列(dfNameList$年齢で取得できるベクトル)の並びは(35,32,21) となっていますので、order(dfNameList$年齢)で返される値は(3,2,1)となります。昇順にソートしたときの順番が返されます。つまり、この行番号順に並べるとソートされるということになります。
並べ替えても行番号(行名)は保持されるので、rownames(dfNamesList)<-NULLで行番号をリセットして完了です。
行のソートその2
dplyrパッケージのarrange()という関数を使うと、もっとスマートにソートできることが分かったので追記します。
> library(dplyr) # dplyrパッケージのロード
> dfPoint # data.frame内容確認
name age point
1 太郎 35 1
2 次郎 32 5
3 三郎 32 10
> arrange(dfPoint, age) # ageでソート(昇順)
name age point
1 次郎 32 5
2 三郎 32 10
3 太郎 35 1
> arrange(dfPoint, age, desc(point)) # ageとpoint複数でソート (pointは降順でソート)
name age point
1 三郎 32 10
2 次郎 32 5
3 太郎 35 1
arrange()で列名を指定すればその列でソートされます。複数列を指定することもできます。降順にしたい場合は列名をdesc()でくくります。
arrange()を使うと行番号は保持されません(勝手にリセットされる)。
こっちのほうが断然分かりやすいですね!
列のソート
data.frameにまとめたデータを"表"としてそのままレポート出力するような場合、列をどのような順番で出力するか、ということを調整したい場合があります。
行はデータ量によって可変なので行名は単なる行番号でしかない場合が多いですが、列名は項目ごとに意味のある名前がついていることが多いです(上の例で示している名前のリストのように...)。
こういう順番で並べたいというのは列名を指定してその順番に並べ替えることができます。
> dfNameList # data.frameの中身の確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList <- dfNameList[,c("住所","名前","年齢")] # 並びを変更して自分自身を更新
> dfNameList # 再度中身を確認
住所 名前 年齢
1 東京 太郎 35
2 千葉 次郎 32
3 神奈川 三郎 21
例えば列名の名前順に並べたいというようなことがあれば、列のソートと同じ要領で、項目名でソートしてその順番に並べるということも可能です。
> dfNameList # 中身確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> colnames(dfNameList) <- c("Name", "Age", "Address") # 分かりやすさのため列名をアルファベットに変更
> dfNameList # 中身確認
Name Age Address
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> order(colnames(dfNameList)) # order関数で列名の順番を確認すると...
[1] 3 2 1
> dfNameList <- dfNameList[,order(colnames(dfNameList))] # order関数の順番で列を取得し直し
> dfNameList # 結果確認
Address Age Name
1 東京 35 太郎
2 千葉 32 次郎
3 神奈川 21 三郎
※補足:
上の例のように、colnames()関数で、列名を参照したり変更したりすることができます。
マージ
行の追加
同じ列を持つ表(data.frame)の行をマージします。
> dfNameList # 内容確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList2 # 内容確認
名前 年齢 住所
1 花子 25 埼玉
2 雪子 22 栃木
> dfNameList3 <- rbind(dfNameList,dfNameList2) # 行をマージ
> dfNameList3 # 結果確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
4 花子 25 埼玉
5 雪子 22 栃木
rbind()でマージする各表は、同一の列数、列名を持つ必要があります。
列の追加
> dfNameList # 内容確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList4 # 内容確認
所属 組織コード
1 営業 A01
2 総務 A02
3 経理 A03
> dfNameList5 <- cbind(dfNameList, dfNameList4) # 列をマージ
> dfNameList5 # 結果確認
名前 年齢 住所 所属 組織コード
1 太郎 35 東京 営業 A01
2 次郎 32 千葉 総務 A02
3 三郎 21 神奈川 経理 A03
cbind()関数でマージする各表は、同一の行数を持つ必要があります。
行名は違っていてもOKでした(あまり付けることは無いと思いますが)。
手っ取り早く1列データを追加するには以下のような方法もあります。
> dfNameList5 # 内容確認
名前 年齢 住所 所属 組織コード
1 太郎 35 東京 営業 A01
2 次郎 32 千葉 総務 A02
3 三郎 21 神奈川 経理 A03
> dfNameList5$年収 <- c("800","500","600") # "年収"列追加
> dfNameList5$性別 <- "男性" # "性別"列追加
> dfNameList5 # 内容確認
名前 年齢 住所 所属 組織コード 年収 性別
1 太郎 35 東京 営業 A01 800 男性
2 次郎 32 千葉 総務 A02 500 男性
3 三郎 21 神奈川 経理 A03 600 男性
異なる表のマージ
> dfNameListA # 内容確認
名前 年齢
1 太郎 35
2 次郎 32
4 花子 25
> dfNameListB # 内容確認
名前 住所
1 太郎 東京
2 次郎 千葉
3 三郎 神奈川
5 雪子 栃木
> dfNameListC <- merge(dfNameListA, dfNameListB) # マージ(オプション無し)
> dfNameListC # 同一列名(ここでは"名前")の値がマッチするものについてマージされる
名前 年齢 住所
1 次郎 32 千葉
2 太郎 35 東京
> dfNameListD <- merge(dfNameListA, dfNameListB, all=TRUE) # マージ(all=TRUEオプション付)
> dfNameListD # 値がマッチしないものも全てマージされる
名前 年齢 住所
1 花子 25 <NA>
2 三郎 NA 神奈川
3 次郎 32 千葉
4 雪子 NA 栃木
5 太郎 35 東京
異なる列名をマッチングさせてマージさせることもできます。
> dfNameListA # 内容確認
名前 年齢
1 太郎 35
2 次郎 32
4 花子 25
> dfNameListB # 内容確認
Name Address
1 太郎 東京
2 次郎 千葉
3 三郎 神奈川
5 雪子 栃木
> dfNameListX <- merge(dfNameListA, dfNameListB) # マージ
> dfNameListX # 重複する列が無いので全組み合わせの表が作成される
名前 年齢 Name Address
1 太郎 35 太郎 東京
2 次郎 32 太郎 東京
3 花子 25 太郎 東京
4 太郎 35 次郎 千葉
5 次郎 32 次郎 千葉
6 花子 25 次郎 千葉
7 太郎 35 三郎 神奈川
8 次郎 32 三郎 神奈川
9 花子 25 三郎 神奈川
10 太郎 35 雪子 栃木
11 次郎 32 雪子 栃木
12 花子 25 雪子 栃木
> dfNameListY <- merge(dfNameListA, dfNameListB, by.x="名前", by.y="Name") # "名前"列と"Name"列をマッチングさせてマージ
> dfNameListY # "名前"列と"Name"列の値が同一のものについてマージされる
名前 年齢 Address
1 次郎 32 千葉
> dfNameListZ <- merge(dfNameListA, dfNameListB, by.x="名前", by.y="Name",all=TRUE) # "名前"列と"Name"列をマッチングさせてマージ (all=TRUEオプション付)
> dfNameListZ # "名前"列と"Name"列の値が異なるものも全てマージ
名前 年齢 Address
1 花子 25 <NA>
2 三郎 NA 神奈川
3 次郎 32 千葉
4 雪子 NA 栃木
5 太郎 35 東京
横持ちから縦持ちへの変換(melt)
data.frameで持っているデータを可視化するために、単なる表形式ではなく様々なグラフを駆使したいのですが、その時使用するグラフの種類や使用するパッケージによって要求される表の形式が異なります。そのため、表の構造を変更しなければならない場合があります。
そういう時に使用される関数の1つとして、melt()という横持ちの表を縦持ちに変換する関数があります。
melt()はreshape2というパッケージで提供される関数なので、library(reshape2)を実行してパッケージをロードしてから利用する必要があります。
> dfWideFormat # 内容確認(wide formatのデータ)
名前 年齢 住所 所属 組織コード 年収 性別
1 太郎 35 東京 営業 A01 800 男性
2 次郎 32 千葉 総務 A02 500 男性
3 三郎 21 神奈川 経理 A03 600 男性
> dfLongFormat <- melt(dfWideFormat, id.vars=c("名前","年齢","組織コード"), variable.name="属性", value.name="値") # wide=>long変換
> dfLongFormat # 内容確認(long formatのデータ)
名前 年齢 組織コード 属性 値
1 太郎 35 A01 住所 東京
2 次郎 32 A02 住所 千葉
3 三郎 21 A03 住所 神奈川
4 太郎 35 A01 所属 営業
5 次郎 32 A02 所属 総務
6 三郎 21 A03 所属 経理
7 太郎 35 A01 年収 800
8 次郎 32 A02 年収 500
9 三郎 21 A03 年収 600
10 太郎 35 A01 性別 男性
11 次郎 32 A02 性別 男性
12 三郎 21 A03 性別 男性
横持ち、縦持ちについて少し補足すると、以下のイメージです。
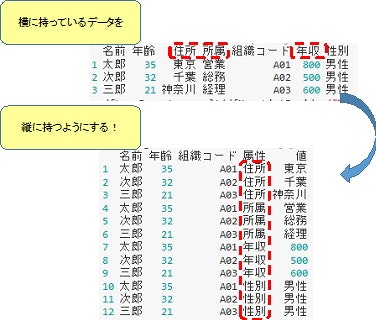
melt関数のid=...で指定した列以外(上の例だと住所、所属、年収)の列名が、"属性"列のデータとして使用されます。各列の値として入っていたデータは、"値"列のデータとなります。
縦持ちへから横持ちの変換(dcast)
meltの逆のイメージで、縦持ちから横持ちの表に変換するdcast()という関数があります。dcast()もmelt()と同様reshape2というパッケージで提供される関数です。
上で縦持ちにしたものを横持ちの表に戻してみましょう。
> dfLongFormat # 内容確認(wide formatのデータ)
名前 年齢 組織コード 属性 値
1 太郎 35 A01 住所 東京
2 次郎 32 A02 住所 千葉
3 三郎 21 A03 住所 神奈川
4 太郎 35 A01 所属 営業
5 次郎 32 A02 所属 総務
6 三郎 21 A03 所属 経理
7 太郎 35 A01 年収 800
8 次郎 32 A02 年収 500
9 三郎 21 A03 年収 600
10 太郎 35 A01 性別 男性
11 次郎 32 A02 性別 男性
12 三郎 21 A03 性別 男性
> dfWideFormat2 <- dcast(dfLongFormat, 名前+年齢+組織コード ~ 属性, value.var="値") # long=>wide変換
> dfWideFormat2 # 内容確認(wide formatのデータ)
名前 年齢 組織コード 住所 所属 年収 性別
1 三郎 21 A03 神奈川 経理 600 男性
2 次郎 32 A02 千葉 総務 500 男性
3 太郎 35 A01 東京 営業 800 男性
行や列の順番は違っていますが、元の表と同じ構造/内容に戻っています。
"名前+年齢+組織コード" の部分がmeltした時のidに相当する部分です。
"~"の右側で指定している列が、横長の列として扱いたいものです。
"value.var"で指定している列が、横長の列にしたときの値になる部分です。
dcastは、他にも集計などで使われるものなので、使い方にいろいろバリエーションがあります。
Helpに載っている例を上げると...
(reshape2に含まれるairqualityというdata.frameの例です)
> names(airquality) <- tolower(names(airquality)) # 列名を小文字に変換
> head(airquality) # airqualityの内容確認(先頭部分のみ)
ozone solar.r wind temp month day
1 41 190 7.4 67 5 1
2 36 118 8.0 72 5 2
3 12 149 12.6 74 5 3
4 18 313 11.5 62 5 4
5 NA NA 14.3 56 5 5
6 28 NA 14.9 66 5 6
> aqm <- melt(airquality, id.vars=c("month", "day"), na.rm=TRUE) # 縦持ちに変換
> head(aqm) # 内容確認(先頭部分のみ)
month day variable value
1 5 1 ozone 41
2 5 2 ozone 36
3 5 3 ozone 12
4 5 4 ozone 18
6 5 6 ozone 28
7 5 7 ozone 23
> dcast(aqm, month ~ variable, mean, value.var="value") # 横持ち変換 & 集計
month ozone solar.r wind temp
1 5 23.61538 181.2963 11.622581 65.54839
2 6 29.44444 190.1667 10.266667 79.10000
3 7 59.11538 216.4839 8.941935 83.90323
4 8 59.96154 171.8571 8.793548 83.96774
5 9 31.44828 167.4333 10.180000 76.90000
上の例だと、melt()の際にはidとしてmonth, dayの二つを指定しています。一方で、dcastで横持ちに戻す際に、idとして指定しているのはmonthのみです。つまり、month毎の集計を行っているという意味になります。また、ここでは集計する際に使われる関数として「mean」を指定していますので、各月の平均値が集計されるということになります。他にはsum(合計)やlength(該当するレコード件数)などが利用できます(指定しない場合はデフォルトでlengthが使用されるます)。
その他よく使う関数
列名を扱う: colnames
colnames()関数で列名を参照したり変更したりすることができます。
> dfNameList # data.frameの内容確認
名前 年齢 住所
1: 太郎 35 東京
2: 次郎 32 千葉
3: 三郎 21 神奈川
> colnames(dfNameList) # 列名を出力
[1] "名前" "年齢" "住所"
> colnames(dfNameList) <- c("Name", "Age", "Address") # 別の値を代入することで列名変更
> dfNameList # 結果確認
Name Age Address
1: 太郎 35 東京
2: 次郎 32 千葉
3: 三郎 21 神奈川
> colnames(dfNameList) <- NULL # NULLを代入することで列名を削除することも可能
> dfNameList # 結果確認
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
列名の変更: rename
dplyrパッケージのrename()関数を使うと、列ごとに列名を置き換えることができます。
> dfPoint # data.frame内容確認
name age point
1 太郎 35 1
2 次郎 32 5
3 三郎 32 10
> rename(dfPoint, 名前=name, ポイント=point) # name, point列の名前を変更
名前 age ポイント
1 太郎 35 1
2 次郎 32 5
3 三郎 32 10
行名を扱う: rownames
rownames()を使用すると、colnames()と同じように行名を操作できます。が、行は可変で扱うことが多いので、あんまり行名を個別につけることは無いでしょう。ソートの節の例でも示したように、並べ替えをした後の列番号をリセットするという時によく使います。
> dfNameList # 内容確認
名前 年齢 住所
1 太郎 35 東京
2 次郎 32 千葉
3 三郎 21 神奈川
> dfNameList <- dfNameList[order(dfNameList$年齢),] # 年齢でソート
> dfNameList # 結果確認
名前 年齢 住所
3 三郎 21 神奈川
2 次郎 32 千葉
1 太郎 35 東京
> rownames(dfNameList) <- NULL # 列番号リセット
> dfNameList # 結果確認
名前 年齢 住所
1 三郎 21 神奈川
2 次郎 32 千葉
3 太郎 35 東京
列数を取得する: ncol
ncol()で列数が取得できます。そのまんまです。
> dfNameList5 # 内容確認
名前 年齢 住所 所属 組織コード
1 三郎 21 神奈川 営業 A01
2 次郎 32 千葉 総務 A02
3 太郎 35 東京 経理 A03
> ncol(dfNameList5) # 列数取得
[1] 5
行数を取得する: nrow
nrow()で列数が取得できます。そのまんまです。
> dfNameList5 # 内容確認
名前 年齢 住所 所属 組織コード
1 三郎 21 神奈川 営業 A01
2 次郎 32 千葉 総務 A02
3 太郎 35 東京 経理 A03
> nrow(dfNameList5)
[1] 3
csvファイルやDBからデータを引っ張ってきて何か処理をさせる場合に、データがある場合と無い場合で処理を分岐させたい場合があります(例えば、データが無い場合はエラーになるので、データがある場合だけなんらかの処理をしたい時など)。その場合、以下のような感じで使うことがあります。
if (nrow(dfXXX)>0) {
# データフレームの処理
}
外部データ取り込み
CSVファイルからデータを読み込むだけでなく、RDBアクセスやJSON/RESTなどを行ってデータを取得したいケースもあると思います。ここではJDBCアクセスとREST(JSON)アクセスの例をあげます。
データベースアクセス(JDBC)
JDBCでRDBのデータを取得する方法を挙げておきます。(ODBCも接続の仕方が異なるだけでほぼ考え方は流用できます。)
RJDBCというパッケージを使用します。(合わせてDBI, rJavaというパッケージも必要になります。)
JDBCなので当然Javaの環境が必要になります。また、環境変数としてJAVA_HOME, PATHも設定しておく必要があります。
PATH環境変数では、JREのbinだけでなく、xxx\jre\bin\defaultディレクトリも追加しないとdllが見つからないというエラーが出ました。(IBM Java V1.7使用)
DB2提供のType4 JDBC Driverを使用する例です。
> jcc <- JDBC("com.ibm.db2.jcc.DB2Driver","c:/xxx/db2jcc4.jar")
> conn <- dbConnect(jcc,"jdbc:db2://hostname:50000/SAMPLE",user="user01",password="xxx") # DBへの接続確立
> rs <- dbSendQuery(conn, "select * from employee") # SQL発行
> df <- fetch(rs, -1) # フェッチしてデータ取得
> dbDisconnect(conn) # DBから切断
[1] TRUE
> head(df) # 取得されたデータ・フレーム確認
EMPNO FIRSTNME MIDINIT LASTNAME WORKDEPT PHONENO HIREDATE JOB EDLEVEL SEX BIRTHDATE SALARY BONUS COMM
1 000010 CHRISTINE I HAAS A00 3978 1995-01-01 PRES 18 F 1963-08-24 152750 1000 4220
2 000020 MICHAEL L THOMPSON B01 3476 2003-10-10 MANAGER 18 M 1978-02-02 94250 800 3300
3 000030 SALLY A KWAN C01 4738 2005-04-05 MANAGER 20 F 1971-05-11 98250 800 3060
4 000050 JOHN B GEYER E01 6789 1979-08-17 MANAGER 16 M 1955-09-15 80175 800 3214
5 000060 IRVING F STERN D11 6423 2003-09-14 MANAGER 16 M 1975-07-07 72250 500 2580
6 000070 EVA D PULASKI D21 7831 2005-09-30 MANAGER 16 F 2003-05-26 96170 700 2893
※補足
df <- fetch(rs, -1)という箇所でデータをフェッチしてデータフレーム(df)に格納していますが、第二引数では取得するレコード数を指定できます。上の例では-1を指定しているので、全レコードを取得するということになります。
以下はSQL Serverアクセスの例です。JDBCのクラスやjarの指定が異なるだけで基本一緒です。JDBCなので当たり前ですが。
jcc <- JDBC("com.microsoft.sqlserver.jdbc.SQLServerDriver","C:/xxx/SQLServer/sqljdbc_4.0/jpn/sqljdbc4.jar")
conn <- dbConnect(jcc,"jdbc:sqlserver://hostname:1433;databaseName=test01",user="sa",password="xxx")
rs <- dbSendQuery(conn, "select * from dbo.table01")
df <- fetch(rs, -1)
dbDisconnect(conn)
JSON/REST
REST APIでアクセスできるサービスからJSONデータを取ってくるような場合を想定します。
例えば、Qiitaの記事についての情報はAPIが公開されているのでRESTで情報を引っ張ってくることが出来ます。
QiitaのAPIについてはこちら。
Qiita API v1 document
このQiitaのAPIを使用して記事の情報を取得してみましょう。
ここではjsonliteというパッケージのfromJSONという関数を使います。
> library(jsonlite) # パッケージのロード
> Name <- "tomotagwork"
> QiitaURI <- paste0("https://qiita.com/api/v1/users/", Name, "/items/") # URIの組み立て
> dfQiitaItemData <- fromJSON(QiitaURI) # 返されたJSONデータをdata.frameとして取得
上の例は、私のQiita ID(tomotagwork)についての記事の情報を取得している例です。
こんな感じのdata.frameの情報が取得されます。1行に1記事の情報が格納されるイメージです(RStudioでdata.frameの中身を表示させた例)。
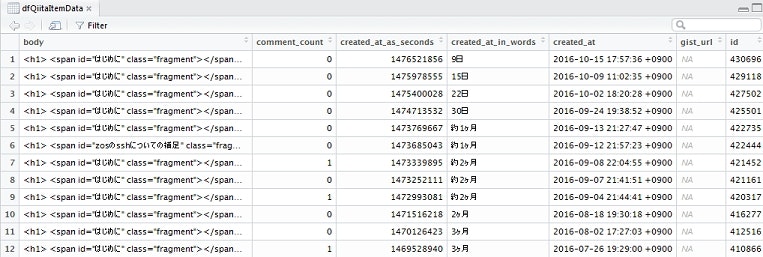
JSONデータが階層になっている部分は、さらに別の型のデータとして情報が格納されることになります。例えば、上の例でいうと、記事毎のタグ情報が含まれるtag列(dfQiitaItemData$tags)のデータは、最大5つのタグ情報を含むList型のデータが格納されることになります。
可視化(グラフ)
最終的には集計した結果を表とかグラフとか分かりやすい形で表現したいです。ただ、これは最終的なレポート出力形式によって使用する機能が違ってくるので、ここではさらりといきます。(後続の記事で書く予定)
よく見るのが、ggplot2というパッケージを使ったグラフ描画です。ヒストグラムとか散布図とか箱ひげ図とか、統計処理でよく使われるグラフが、結構きれいに、割と簡単に描けます。ここではあまり統計的な分析を主眼としていないし、この辺はググると色々詳しい情報はたくさんあるし、で、そちらをご参照ください。
例えば、この辺はggplotの考え方が分かりやすいかと。
Rのグラフィック作成パッケージ“ggplot2”について
こういうのもありますね。
ggplot2に関する資料まとめ
ポイントは、利用するパッケージ/機能によって、どういう形でデータを与えなければいけないかが変わってくるという点です。
利用しようとしているパッケージ/機能が求めているデータ形式に合わせてデータを整形し、グラフ化する、という流れになります。そのために先に示したようなdata.frameの操作なんかが必要になってきます。
日付/時刻の操作
日付/時刻取得
Sys.Date()、Sys.time() という関数でそれぞれその時点の日付、日時を取得できます。
> Sys.Date()
[1] "2016-10-24"
> Sys.time()
[1] "2016-10-24 17:11:35 JST"
文字列の日付/時刻型への変換
csvファイルなどに書き出されたパフォーマンス情報を取得するような場合、文字列としての日付/時刻を含む時系列の情報を取り扱うような場合があると思います。
グラフ表記やソートなどを行う場合、文字列ではなく日付/時刻型のデータとしてそれらを扱う方が便利なことが多いので、文字列=>日時型への変換方法について補足します。
フォーマット変換には、Rのベースに含まれるas.POSIXct()という関数が使えます。
> strDateTime <- "01/31/2016 12:00:00.123456" # 日時を文字列としてセット
> strDateTime # 内容確認
[1] "01/31/2016 12:00:00.123456"
> options(digits.secs=6) # 秒の小数点以下の桁数設定
> posixDateTime1 <- as.POSIXct(strDateTime, format="%m/%d/%Y %H:%M:%OS", tz="Asia/Tokyo") # 型変換
> posixDateTime1 # 変換結果確認
[1] "2016-01-31 12:00:00.123456 JST"
ですが、これはパフォーマンスがあまりよくないので、lubridateというパッケージに含まれるparse_date_time2()という関数を使うのがおススメです。同じような感じで変換できます。
> library(lubridate)
> strDateTime <- "01/31/2016 12:00:00.123456"
> options(digits.secs=6)
> posixDateTime2 <- parse_date_time2(strDateTime, "%m/%d/%Y %H:%M:%OS", tz="Asia/Tokyo")
> posixDateTime2
[1] "2016-01-31 12:00:00.123456 JST"
単に1つの変数の変換だとパフォーマンスの違いは分からないと思いますが、例えば100万行くらいあるトランザクションデータのタイムスタンプを変換しようとした場合には結構な違いになります。
参考: 実際に変換のパフォーマンス比較している記事
R で文字列を POSIX time に変換するには lubridate::parse_date_time2() がちょっぱや #rstatsj
R で文字列を POSIX time に変換するには lubridate::parse_date_time2() がやっぱりちょっぱや