ウワサの赤い本、機械学習プロフェッショナルシリーズ「異常検知と変化検知」(http://ide-research.net/book/support.html#kodansha) 第1章のグラフをPythonで書いてみる、という記事です。
こんな感じで、アニメーションで標本精度とROC曲線を描いたりしています。
コードの全体はこちら
解説は簡易的なものしかここでは記載していませんので、詳細を知りたい方は是非本をご購入ください!
図1.1 時系列データの様々な異常の例
全く同じデータではなく、似たようなデータをPythonで生成、もしくはデータを探してplotしました。
(特に心電図データ1は探すのが一苦労でした・・・ ![]() )
)
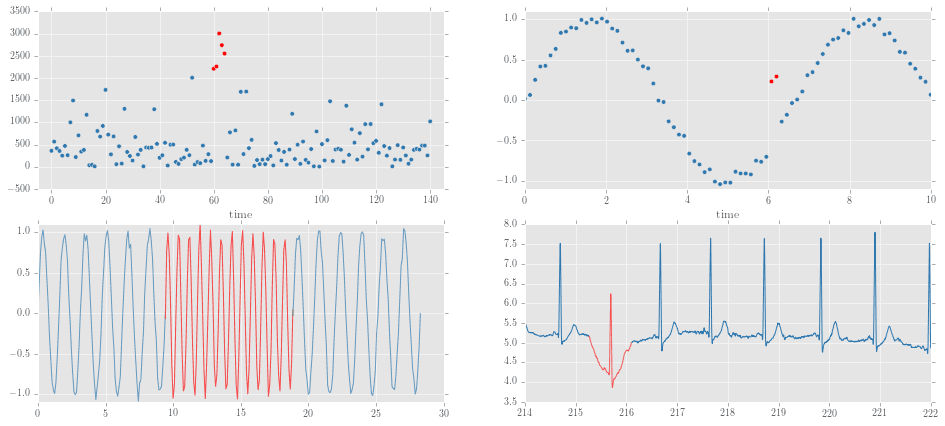
これを描画するコード全体はこちら
図1.2 ラベル付きデータについての異常判定の説明
図1.2から異常度、指示関数をつなげて描画して関連性を可視化してみました。
- 2つの正規分布に従う乱数からデータ生成し、青を正常データ、赤を異常データとしてplot
- 1.のヒストグラムから平均、標準偏差を算出し、正規分布の密度関数を描画。(分布からカーネル密度推定したものも点線で描画)
- $\ln p({\bf x}'|y=0,\mathcal{D})$、$-\ln p({\bf x}'|y=1,\mathcal{D})$を別々に描画。
- 異常度$a(x') \ln{ p({\bf x}'|y=1,\mathcal{D})\over p({\bf x}'|y=0,\mathcal{D})} $を描画
- 指示関数 $I[a(x) \ge \tau]$ を描画
さらに、分岐点閾値を密度関数の値が同じになる点として計算し、その場合の$\tau$を4つ目のグラフの横棒で表しています。
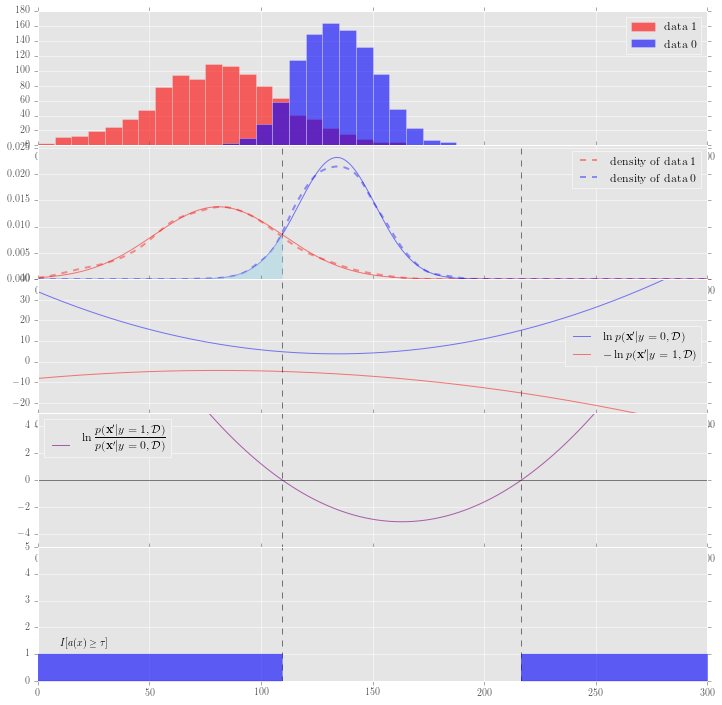
これを描画するコード全体はこちら
Pythonコード
# 乱数生成
rd.seed()
n = 1000
d_0 = rd.normal(135, 18, n) # 正常データ
d_1 = rd.normal(80, 30, n) # 異常データ
# 平均、標準偏差の算出
m_0 = np.mean(d_0)
sd_0 = np.sqrt(np.var(d_0))
m_1 = np.mean(d_1)
sd_1 = np.sqrt(np.var(d_1))
# X軸データ生成
xx = np.linspace(0,300, 5000)
# 正規分布の密度関数
density_0 = st.norm.pdf(xx, m_0, sd_0)
density_1 = st.norm.pdf(xx, m_1, sd_1)
# 異常度の計算
abnormaly_score = np.log(density_1) - np.log(density_0)
def balance_position(x_min, x_max, f1, f2, EPS=0.00001):
if abs(f1(x_max) - f2(x_max)) > EPS:
center = (x_min + x_max)/2.
if np.sign(f1(x_max) - f2(x_max)) * np.sign(f1(center) - f2(center)) < 0:
x_min = center
else:
x_max = center
x_max = balance_position(x_min, x_max, f1, f2)
else:
return x_max
return x_max
mark = balance_position(0, 200, lambda x:st.norm.pdf(x, m_0, sd_0), lambda x: st.norm.pdf(x, m_1, sd_1))
print "mark:", mark
tau_pos = np.argsort(np.abs(xx - mark))[0]
print "tau pos:", tau_pos
tau = abnormaly_score[tau_pos]
print "tau:", tau
tau2_pos = np.max(np.argsort(np.abs(abnormaly_score - tau))[0:2])
print "tau2_pos:",tau2_pos
tau2 = abnormaly_score[tau2_pos]
print "tau2:",tau2
mark2 = xx[tau2_pos]
print "mark2:",mark2
# ---------------- 描画処理 -----------------#
n_row = 5 # グラフの行数
plt.subplots(n_row, 1, sharex=True,figsize=(12,12))
gs = gridspec.GridSpec(n_row, 1, height_ratios=[3,3,3,3,3])
axs = [plt.subplot(gs[i]) for i in range(n_row) ]
# 1つ目のエリア描画
axs[0].hist(d_1, bins=40, color="r", alpha=0.6, range=(0,300), label="data 1")
axs[0].hist(d_0, bins=40, color="b", alpha=0.6, range=(0,300), label="data 0")
axs[0].legend(loc='best')
# 2つ目のエリア描画
axs[1].plot(xx, get_density(d_1, xx), "r--", alpha=.4 , lw=2, label=r"density of data 1")
axs[1].plot(xx, get_density(d_0, xx), "b--", alpha=.4 , lw=2, label=r"density of data 0")
axs[1].legend(loc='best')
axs[1].plot([0,300],[0,0],"k")
axs[1].plot(xx, density_1, c="r", alpha=.5 )
axs[1].plot(xx, density_0, c="b", alpha=.5 )
axs[1].fill_between(xx[0:tau_pos], density_0[0:tau_pos], color="lightblue", zorder = 500, alpha=.6)
axs[1].set_ylim(0,np.max(density_0)*1.1)
axs[1].plot([mark ,mark],[-100,200], "k--", lw=.5)
axs[1].plot([mark2 ,mark2],[-100,200], "k--", lw=.5)
# 3つ目のエリア描画
axs[2].plot(xx, -np.log(density_0), c="b", alpha=.5, label=r"$\ln p({\bf x}'|y=0,\mathcal{D})$")
axs[2].plot(xx, np.log(density_1), c="r", alpha=.5, label=r"$-\ln p({\bf x}'|y=1,\mathcal{D})$")
axs[2].plot([mark ,mark],[-110,200], "k--", lw=.5)
axs[2].plot([mark2 ,mark2],[-110,200], "k--", lw=.5)
axs[2].set_ylim(-25,40)
axs[2].legend(loc='best')
# 4つ目のエリア描画
axs[3].plot(xx, abnormaly_score, c="purple", alpha=.6, label=r"$$ \ln{ p({\bf x}'|y=1,\mathcal{D})\over p({\bf x}'|y=0,\mathcal{D})} $$")
axs[3].set_ylim(-5,5)
axs[3].plot([mark ,mark],[-100,200], "k--", lw=.5)
axs[3].plot([mark2 ,mark2],[-100,200], "k--", lw=.5)
axs[3].plot([0 ,300],[tau, tau], "k", lw=.5)
axs[3].legend(loc='best')
# 5つ目のエリア描画
axs[4].fill_between(xx[0:tau_pos], np.ones_like(xx[0:tau_pos]), color="blue", zorder = 500, alpha=.6)
axs[4].fill_between(xx[tau2_pos:], np.ones_like(xx[tau2_pos:]), color="blue", zorder = 500, alpha=.6)
axs[4].plot([mark, mark],[-110,200], "k--", lw=.5)
axs[4].plot([mark2, mark2],[-110,200], "k--", lw=.5)
axs[4].text(10, 1.3, r"$I[a(x) \ge \tau]$")
axs[4].set_ylim(0,5)
# すべてのエリアでxの範囲を固定
for ax in axs:
ax.set_xlim(0,300)
plt.subplots_adjust(hspace=0)
図1.3 ラベルなしデータについての異常度の定義
図1.2と同様、ヒストグラム、カーネル密度、平均・分散から推定した正規分布の密度関数を描き、「情報量」のプロットをしています。異常度は単純にデータの平均から離れるほど高くなる形となっています。
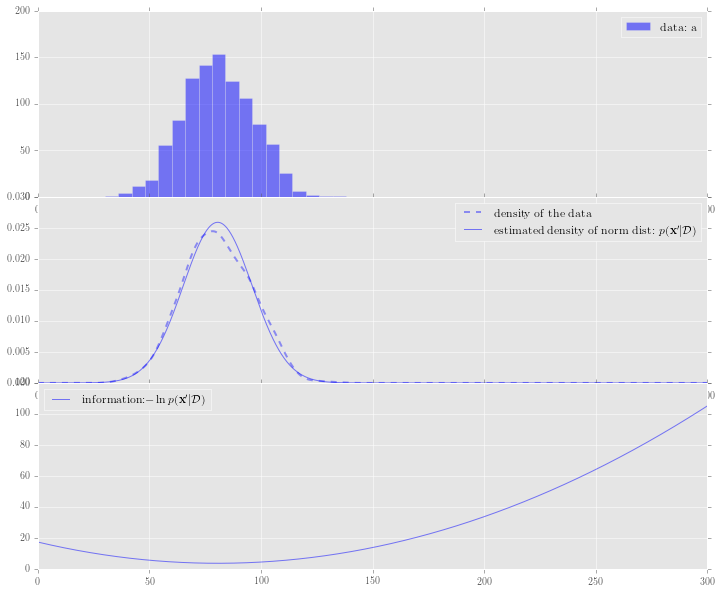
これを描画するコード全体はこちら
Pythonコード
# ラベルなしデータの場合
# 乱数生成
n = 1000
data = rd.normal(80, 15, n)
# 平均、標準偏差の算出
m = np.mean(data)
sd = np.sqrt(np.var(data))
# X軸データ生成
xx = np.linspace(0,300, 5000)
# 正規分布の密度関数
density = st.norm.pdf(xx, m, sd)
# ---------------- 描画処理 -----------------#
n_row = 3 # グラフの行数
xx = np.linspace(0,300, 5000) # X軸データ生成
plt.subplots(n_row, 1, sharex=True,figsize=(12,10))
gs = gridspec.GridSpec(n_row, 1, height_ratios=[3,3,3])
axs = [plt.subplot(gs[i]) for i in range(n_row) ]
# 1つ目のエリア描画
axs[0].hist(data, bins=50, range=(0,300), label="data: a", color="b", alpha=0.5)
axs[0].set_ylim(0,200)
axs[0].legend(loc='best')
# 2つ目のエリア描画
axs[1].plot(xx, get_density(data, xx), lw=2, linestyle="--", label="density of the data", color="b", alpha=0.4)
axs[1].plot(xx, density, lw=1, label=r"estimated density of norm dist: $p({\bf x}'|\mathcal{D})$", color="b", alpha=0.5)
axs[1].legend(loc='best')
# 3つ目のエリア描画
axs[2].plot(xx, -np.log(density), lw=1, label=r"information:$-\ln p({\bf x}'|\mathcal{D})$", color="b", alpha=0.5)
axs[2].legend(loc='best')
# すべてのエリアでxの範囲を固定
for ax in axs:
ax.set_xlim(0,300)
plt.subplots_adjust( hspace=0)
図1.4, 図1.5 正常標本精度、異常標本精度とROC曲線
正常標本精度、異常標本精度とROC曲線の関係をアニメーションにしてみました。
また、ROC曲線の詳細については以前に「統計学】ROC曲線とは何か、アニメーションで理解する。
」という記事も書いていますのでよければご参考ください。
この図では標本精度が下記で表せていたとして描いています。
r_0 = \log(1 + x) \\
r_1 = \log(e -x)
2段目のグラフはF値を表現しており定義は下記です。
f \equiv { 2r_0 r_1 \over r_0 + r_1 }
ROC曲線は $(X, Y) = (1 - r_0(\tau),\ r_1(\tau))$なので、赤い線をなぞる点のy座標値は$1-y$を使います。座標をアニメーション上に表示していますが、二つあるうちの右側の数字がこれにあたります。
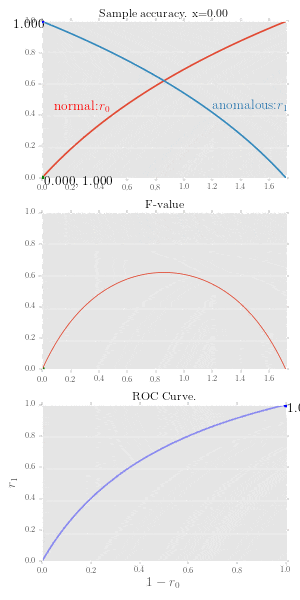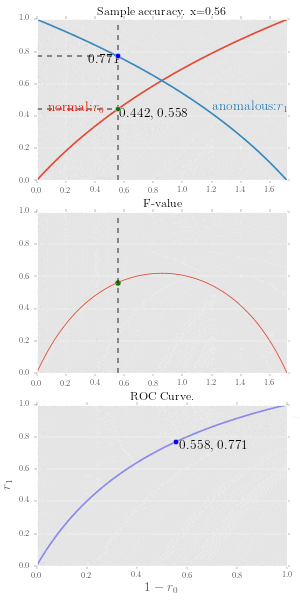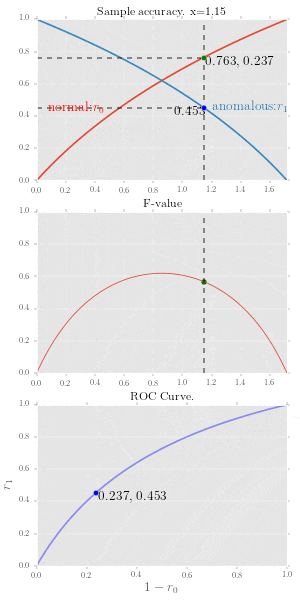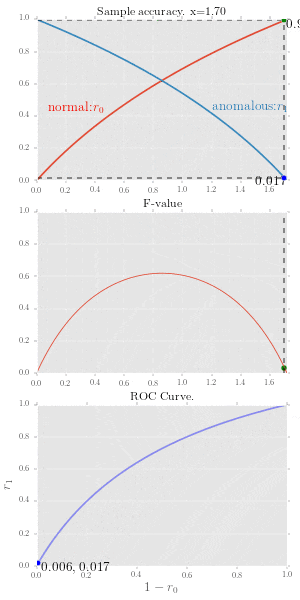
これを描画するコード全体はこちら
Pythonコード
def animate(nframe):
global num_frame
sys.stdout.write(str(int(float(nframe)/num_frame*100)) + "%, ")
plt.clf()
# xの最小値、最大値
xmin = 0
xmax = np.e -1
# xの分割数
sx = num_frame * 2
# 現在位置
pos = nframe * 2
# x軸生成
xx = np.linspace(xmin, xmax, sx)
# 標本精度
cx1 = np.log(1+xx)
cx2 = np.log(np.e -xx)
# 1つ目のグラフ描画 -----------------------
plt.subplot(311)
plt.title("Sample accuracy. x={0:.2f}".format(xx[pos]))
plt.xlim(xmin, xmax)
plt.ylim(0,1)
# 曲線の描画
plt.plot(xx,cx1,linewidth=2)
plt.plot(xx,cx2,linewidth=2)
# 点と座標値の描画
plt.scatter(xx[pos],cx1[pos], c="g", s=40, zorder=100)
plt.text(xx[pos]+.01,cx1[pos]-.05,"{0:.3f}, {1:.3f}".format(cx1[pos], 1-cx1[pos]), size=16)
plt.scatter(xx[pos],cx2[pos], c="b", s=40, zorder=100)
plt.text(xx[pos]-.20,cx2[pos]-.05,"{0:.3f}".format(cx2[pos]), size=16)
# 点線の描画
plt.plot([xx[pos], xx[pos]], [0,1], "k--", alpha=.5, lw=2 )
plt.plot([0, xx[pos]], [cx1[pos],cx1[pos]], "k--", alpha=.5, lw=2 )
plt.plot([0, xx[pos]], [cx2[pos],cx2[pos]], "k--", alpha=.5, lw=2 )
plt.text(.08, .42, r"normal:$r_0$", color="r", size=16)
plt.text(1.2, .42, r"anomalous:$r_1$", color="#2F79B0", size=16)
# 2つ目のグラフ描画 -----------------------
plt.subplot(312)
plt.title("F-value")
plt.xlim(xmin, xmax)
plt.ylim(0,1)
F = 2*cx1*cx2/(cx1+cx2)
F_pos = 2*cx1[pos]*cx2[pos]/(cx1[pos]+cx2[pos])
plt.scatter(xx[pos], F_pos, c="g", alpha=1, s=50)
plt.plot(xx, F)
plt.plot([xx[pos], xx[pos]], [0,1], "k--", alpha=.5, lw=2 )
plt.tight_layout()
# 3つ目のグラフ描画 -----------------------
plt.subplot(313)
plt.title("ROC Curve.")
plt.xlim(0,1)
plt.ylim(0,1)
# 曲線と点の描画
plt.plot(1-cx1, cx2, linewidth=2, color="b", alpha=.4)
plt.scatter(1-cx1[pos],cx2[pos], c="b", s=40, zorder=100)
plt.text(1-cx1[pos]+.01,cx2[pos]-.05, "{0:.3f}, {1:.3f}".format(1-cx1[pos], cx2[pos]), size=16)
plt.xlabel(r"$1-r_0$", size=16)
plt.ylabel(r"$r_1$", size=16)
plt.tight_layout()
num_frame = 50
fig = plt.figure(figsize=(5,10))
anim = ani.FuncAnimation(fig, animate, frames=num_frame, blit=True)
anim.save('ROC_curve2.gif', writer='imagemagick', fps=3, dpi=60)
参考図書
「異常検知と変化検知」 井手剛、杉山将著 (機械学習プロフェッショナルシリーズ)
http://ide-research.net/book/support.html
-
異常にした赤い部分は加工したものなので、正しい心電図データではないことにご注意ください。データの取得先URLについては、GitHubの方に記載してあります。 ↩