遺伝的アルゴリズムの勉強の手始めにN-Queen問題を解いてみる(解の一つを求める)ことにしました。プログラム言語は何でもよかったのですが、手軽に書けるのでpythonを使うことにします。
N-Queen問題
NxNの盤面上にN個のチェスのQueenを互いに取り合わないように置くパターンを求める問題。
Queenはタテ、ヨコ、ナナメに制限なく動けます。
本当は解のすべてを見つけられれば良いのですが、今回は遺伝的アルゴリズムを用いて解のうちの一つを求めてみます。
問題を簡単にするため、はじめにN=8の場合を考え、その後任意のNに対応させてみます。
遺伝的アルゴリズムの適用
遺伝子の定義
まずは遺伝子をどう表すか、です。最終的な解を表現できるものにする必要があります。
そこで盤面上のQueenの位置を表す1次のベクトルを遺伝子として採用します。
各列のQueenの位置を 0~7 で表します。例を示します。
[0, 2, 3, 1, 4, 3, 6, 7]
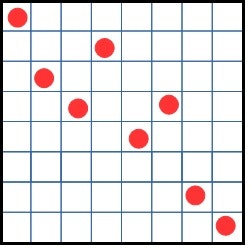
これを踏まえて初期遺伝子を用意します。今回は4つとします。ベクトルの各要素は 0~7 をランダムに選択します。
def makeIniGene():
ini_gene = []
for cnt in range(0, 4):
line = []
for i in range(0, 8):
val = random.randint(0, 7)
line.append(val)
ini_gene.append(line)
return ini_gene
適応度
適応度は遺伝子が目標に対してどの程度近いかを表す指標です。
今回はとある遺伝子=盤面上のQueenの配置において、Queenが互いに取り合っている数を適応度とします。したがって、最終的に解が見つかる状態とは、適応度=0の場合になります。
適応度の計算は、あるQueenの位置から8方向に1マスずつ探索し、ほかのQueenが見つかったら、適応度を+1します。
gVec = [(-1,-1), (-1,0), (-1,1), (0,-1), (0,1), (1,-1), (1,0), (1,1)]
def calcFitness(gene):
fitness = 0
board = []
line = []
for i in range(0, 8):
line = []
for j in range(0, 8):
if j == gene[i]:
line.append(1)
else:
line.append(0)
board.append(line)
for i in range(0, 8):
for j in range(0, 8):
val = getCell(board, (i,j), (0,0))
if val == 1:
for vec in gVec:
for k in range(1, 8):
valofst = getCell(board, (i,j), (vec[0]*k, vec[1]*k))
if valofst == 1:
fitness += 1
elif valofst == -1:
break
return fitness
def getCell(board, pos, ofst):
posx = pos[0] + ofst[0]
posy = pos[1] + ofst[1]
if posx >= 0 and posy >= 0 and posx < 8 and posy < 8:
val = board[posx][posy]
else:
val = -1
return val
計算結果の例を示します。
[0, 1, 2, 3, 4, 5, 6, 7]
=> fitness = 56
遺伝
例として以下の4つの遺伝子を親とし、子の遺伝子を生成します。
G0 = [0, 1, 2, 3, 4, 5, 6, 7]
=> fitness = 56
G1 = [0, 0, 1, 1, 2, 2, 3, 3]
=> fitness = 14
G2 = [0, 2, 4, 6, 7, 5, 3, 1]
=> fitness = 6
G3 = [1, 3, 5, 7, 0, 1, 2, 3]
=> fitness = 20
適応度の小さい順に並べます。順位をrank=0~3とします。
G2 = [0, 2, 4, 6, 7, 5, 3, 1]
=> rank = 0
G1 = [0, 0, 1, 1, 2, 2, 3, 3]
=> rank = 1
G3 = [1, 3, 5, 7, 0, 1, 2, 3]
=> rank = 2
G0 = [0, 1, 2, 3, 4, 5, 6, 7]
=> rank = 3
最初に優秀な遺伝子を残します。適応度の最も大きい遺伝子を最も小さい遺伝子に置き換えます。この場合、G0の値をG2の値で上書きします。
G2 = [0, 2, 4, 6, 7, 5, 3, 1]
G1 = [0, 0, 1, 1, 2, 2, 3, 3]
G3 = [1, 3, 5, 7, 0, 1, 2, 3]
G0' = [0, 2, 4, 6, 7, 5, 3, 1]
次に、交叉を行います。交叉は親の遺伝子から子の遺伝子を生成する手順です。
今回は単純な交叉を考えます。
まずrank=0の遺伝子とrank=1の遺伝子のk番目以降の要素を入れ替えます。
今回はk=5番目以降の要素を入れ替えてみます。
G2' = [0, 2, 4, 6, 2, 2, 3, 3]
G1' = [0, 0, 1, 1, 7, 5, 3, 1]
次にrank=2の遺伝子とrank=3の遺伝子のm番目以降の要素を入れ替えます。
今回はm=7番目以降の要素を入れ替えてみます。
G3' = [1, 3, 5, 7, 0, 1, 3, 1]
G0'' = [0, 2, 4, 6, 7, 5, 2, 3]
以上から子の遺伝子は以下の4つになります。
G2' = [0, 2, 4, 6, 2, 2, 3, 3]
G1' = [0, 0, 1, 1, 7, 5, 3, 1]
G3' = [1, 3, 5, 7, 0, 1, 3, 1]
G0'' = [0, 2, 4, 6, 7, 5, 2, 3]
遺伝の部分をソースコードで表すと以下のようになります。
gene_listは親の遺伝子のリストです。
rank_listはそのindexをrankとして、その要素にgene_listのindexを格納しています。
つまり今回の例の場合、rank_list = [2, 1, 3, 0] というイメージです。
gGaIdx = [4, 6]
def simpleGa(gene_list, rank_list):
new_gene_list = []
for i in range(0, gGeneCnt):
if i == rank_list[3]:
new_gene_list.append(gene_list[rank_list[0]])
else:
new_gene_list.append(gene_list[i])
updated_gene_list = []
line1 = []
line2 = []
for i in range(0, 8):
if i < gGaIdx[0]:
line1.append(new_gene_list[rank_list[0]][i])
line2.append(new_gene_list[rank_list[1]][i])
else:
line1.append(new_gene_list[rank_list[1]][i])
line2.append(new_gene_list[rank_list[0]][i])
updated_gene_list.append(line1)
updated_gene_list.append(line2)
line1 = []
line2 = []
for i in range(0, 8):
if i < gGaIdx[1]:
line1.append(new_gene_list[rank_list[2]][i])
line2.append(new_gene_list[rank_list[3]][i])
else:
line1.append(new_gene_list[rank_list[3]][i])
line2.append(new_gene_list[rank_list[2]][i])
updated_gene_list.append(line1)
updated_gene_list.append(line2)
return updated_gene_list
突然変異
遺伝の手順(交叉)を繰り返すことで優秀な遺伝子を残すように向かいます。
ただ、交叉を繰り返すだけでは、局所解に陥る場合があります。
8-Queen問題の局所解の例を挙げると、適応度=2、つまり残り一組のQueenが互いに取り合う状態から抜け出せない状態になることがあります。
この状態を打破するため、突然変異を設ける必要がります。
今回は単純に4回の遺伝手順ごとに、あるひとつの遺伝子のひとつの要素を0~7のランダムな値に置き換える、という方法をとることにします。
# mutation
if loop % 4 == 0:
geneidx = random.randint(0, 3)
posidx = random.randint(0, 7)
valrand = random.randint(0, 7)
gene_list[geneidx][posidx] = valrand
まとめ
以上をまとめて、ソースコードに起こします。
# ! /usr/bin/env python
# encoding=utf-8
# 8 Queens Problem
import sys
import random
gGeneCnt = 4
gMutationSpan = 4
gRange = 0
gVec = [(-1,-1), (-1,0), (-1,1), (0,-1), (0,1), (1,-1), (1,0), (1,1)]
gGaIdx = [4, 6]
def makeIniGene():
ini_gene = []
for cnt in range(0,gGeneCnt):
line = []
for i in range(0, 8):
val = random.randint(0, 7)
line.append(val)
ini_gene.append(line)
return ini_gene
def calcFitness(gene):
fitness = 0
board = []
line = []
for i in range(0, 8):
line = []
for j in range(0, 8):
if j == gene[i]:
line.append(1)
else:
line.append(0)
board.append(line)
for i in range(0, 8):
for j in range(0, 8):
val = getCell(board, (i,j), (0,0))
if val == 1:
for vec in gVec:
for k in range(1, 8):
valofst = getCell(board, (i,j), (vec[0]*k, vec[1]*k))
if valofst == 1:
fitness += 1
elif valofst == -1:
break
return fitness
def getCell(board, pos, ofst):
posx = pos[0] + ofst[0]
posy = pos[1] + ofst[1]
if posx >= 0 and posy >= 0 and posx < 8 and posy < 8:
val = board[posx][posy]
else:
val = -1
return val
def simpleGa(gene_list, rank_list):
new_gene_list = []
for i in range(0, gGeneCnt):
if i == rank_list[3]:
new_gene_list.append(gene_list[rank_list[0]])
else:
new_gene_list.append(gene_list[i])
updated_gene_list = []
line1 = []
line2 = []
for i in range(0, 8):
if i < gGaIdx[0]:
line1.append(new_gene_list[rank_list[0]][i])
line2.append(new_gene_list[rank_list[1]][i])
else:
line1.append(new_gene_list[rank_list[1]][i])
line2.append(new_gene_list[rank_list[0]][i])
updated_gene_list.append(line1)
updated_gene_list.append(line2)
line1 = []
line2 = []
for i in range(0, 8):
if i < gGaIdx[1]:
line1.append(new_gene_list[rank_list[2]][i])
line2.append(new_gene_list[rank_list[3]][i])
else:
line1.append(new_gene_list[rank_list[3]][i])
line2.append(new_gene_list[rank_list[2]][i])
updated_gene_list.append(line1)
updated_gene_list.append(line2)
return updated_gene_list
def printBoard(gene):
for i in range(0, len(gene)):
line = []
for j in range(0, len(gene)):
if j == gene[i]:
line.append(1)
else:
line.append(0)
print line
def main(argv):
gene_list = makeIniGene()
print "Initial gene = " + str(gene_list)
fitness = []
for i in range(0, gGeneCnt):
fitness.append(0)
loop = 0
while True :
loop += 1
idx = 0
max_fitness_idx = []
min_fitness_idx = []
# mutation
if loop % gMutationSpan == 0:
geneidx = random.randint(0, gGeneCnt-1)
posidx = random.randint(0, 7)
valrand = random.randint(0, 7)
gene_list[geneidx][posidx] = valrand
# compare fitness
for gene in gene_list:
fitness[idx] = calcFitness(gene)
if idx == 0:
max_fitness_idx = (fitness[0], 0)
min_fitness_idx = (fitness[0], 0)
if max_fitness_idx[0] < fitness[idx]:
max_fitness_idx = (fitness[idx], idx)
if min_fitness_idx[0] > fitness[idx]:
min_fitness_idx = (fitness[idx], idx)
print fitness[idx]
idx += 1
min_fitness = min(fitness)
if min_fitness <= gRange:
print "Loop end = " + str(loop) + ", Fitness = " + str(min_fitness)
printBoard(gene_list[min_fitness_idx[1]])
break
ranktemp = []
for i in range(0, gGeneCnt):
if i != max_fitness_idx[1] and i != min_fitness_idx[1]:
ranktemp.append(i)
if fitness[ranktemp[0]] > fitness[ranktemp[1]]:
rank_list = [ min_fitness_idx[1], ranktemp[1], ranktemp[0], max_fitness_idx[1] ]
else:
rank_list = [ min_fitness_idx[1], ranktemp[0], ranktemp[1], max_fitness_idx[1] ]
updated_gene_list = []
updated_gene_list = simpleGa(gene_list, rank_list)
gene_list = updated_gene_list
if __name__ == "__main__":
main(sys.argv)
実行するとダァーッと適応度が表示され、解がみつかると(適応度=0)、Loop回数と盤面上のQueenの位置を示します。
$ ./8-queen.py
...
...
2
2
2
2
2
2
0
2
Loop end = 12964, Fitness = 0
[0, 0, 0, 1, 0, 0, 0, 0]
[1, 0, 0, 0, 0, 0, 0, 0]
[0, 0, 0, 0, 1, 0, 0, 0]
[0, 0, 0, 0, 0, 0, 0, 1]
[0, 0, 0, 0, 0, 1, 0, 0]
[0, 0, 1, 0, 0, 0, 0, 0]
[0, 0, 0, 0, 0, 0, 1, 0]
[0, 1, 0, 0, 0, 0, 0, 0]
次回はN=8から任意のNへ拡張してみます。