はじめに
この記事は
Ten Simple Rules for Better Figures
Nicolas P. Rougier ,Michael Droettboom,Philip E. Bourne
Published: September 11, 2014 https://doi.org/10.1371/journal.pcbi.1003833
を抜粋して翻訳したものです。いわゆる抄訳(しょうやく)です。
以下が本文です。
本文
科学的視覚化は、古典的に、科学データをグラフィカルに表示するプロセスとして定義されています。ただし、このプロセスは直接または自動ではありません。同じデータを表すには、散布図、線形プロット、棒グラフ、円グラフなど、さまざまな方法があります。さらに、同じタイプのプロットを使用した同じデータは、誰が図を見ているかによって、非常に異なって認識される可能性があります。科学的視覚化のより正確な定義は、人とデータの間のグラフィカルインターフェイスです。この短い記事では、このインターフェースについてすべてを説明するふりをするわけではありません。図表のデザインを改善し、一般的な落とし穴のいくつかを説明するための基本的なルールセットを提供することを目指しています。
ルール1:あなたの聴衆を知る
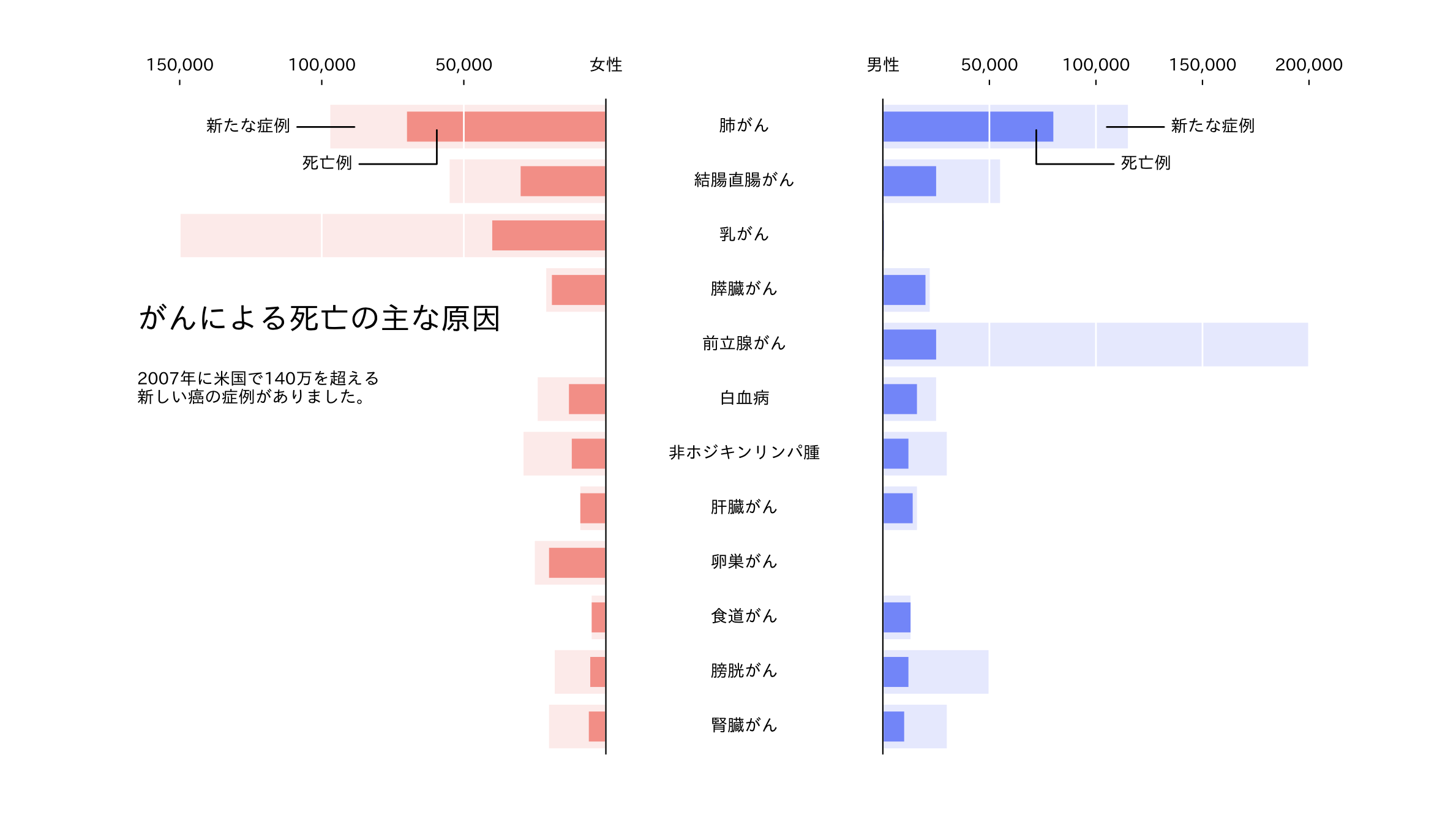
上記の定義を考えると、視覚がどのように知覚されるかが話者の意図と大幅に異なる場合に問題が発生します。したがって、デザインプロセスのできるだけ早い段階で、視聴者とビジュアルが伝えるメッセージを特定することが重要です。ビジュアルのグラフィックデザインは、この意図によって通知されるべきです。あなた自身とあなたの直接の協力者のためにフィギュア(図表)を作っているなら、あなたはおそらく、フィギュアが何であるかを知っているので、デザインプロセスのいくつかのステップをスキップすることができます。ただし、科学雑誌に図を掲載する場合は、図が正しく、関連するすべての情報をより多くの読者に伝える必要があります。その状況の目標は概念を説明することであるため、学生の聴衆は特別な注意を必要とします。その場合、概念を完全に理解するために、追加情報を追加する必要がある場合があります。最後に、あなたはあなたの研究の最も顕著な部分だけを明らかにする単純な、おそらく近似された図を設計する必要があるので、一般大衆はすべての中で最も難しい聴衆かもしれません(図1)。
(図1)
これは、2007年にニューヨークタイムズ(NYT)で最初に公開された図のリメイクです。この新しい図は、近似データを使用してmatplotlibで作成されました。データは、古典的な2列(死亡/ケース)の棒グラフを使用して表示できた4つのシリーズ(男性の死亡/ケース、女性の死亡/ケース)で構成されています。ただし、ここで使用されているレイアウトは、対象読者に適しています。これは、新しいケースの数が常に対応する死亡数よりも多いという事実を利用して、2つの値を混合します。また、中央のラベルが図のメインメッセージ(癌)にすぐにアクセスできるようにしながら、男性と女性の比較を容易にするために、読み方(NYTの英語[左から右])を利用します。これは、癌による死亡について明確なメッセージを伝える自己完結型の図です。ただし、正確ではありません。選択されたレイアウトでは、下部の位置と上部のラベル付きティックの位置が原因で、腎臓がんによる死亡数を推定することは実際には困難です。これは一般向けの出版物には受け入れられますが、実際の数値が記事の他の場所に記載されていない場合、科学的な出版物には受け入れられません。
ルール2:メッセージを特定する
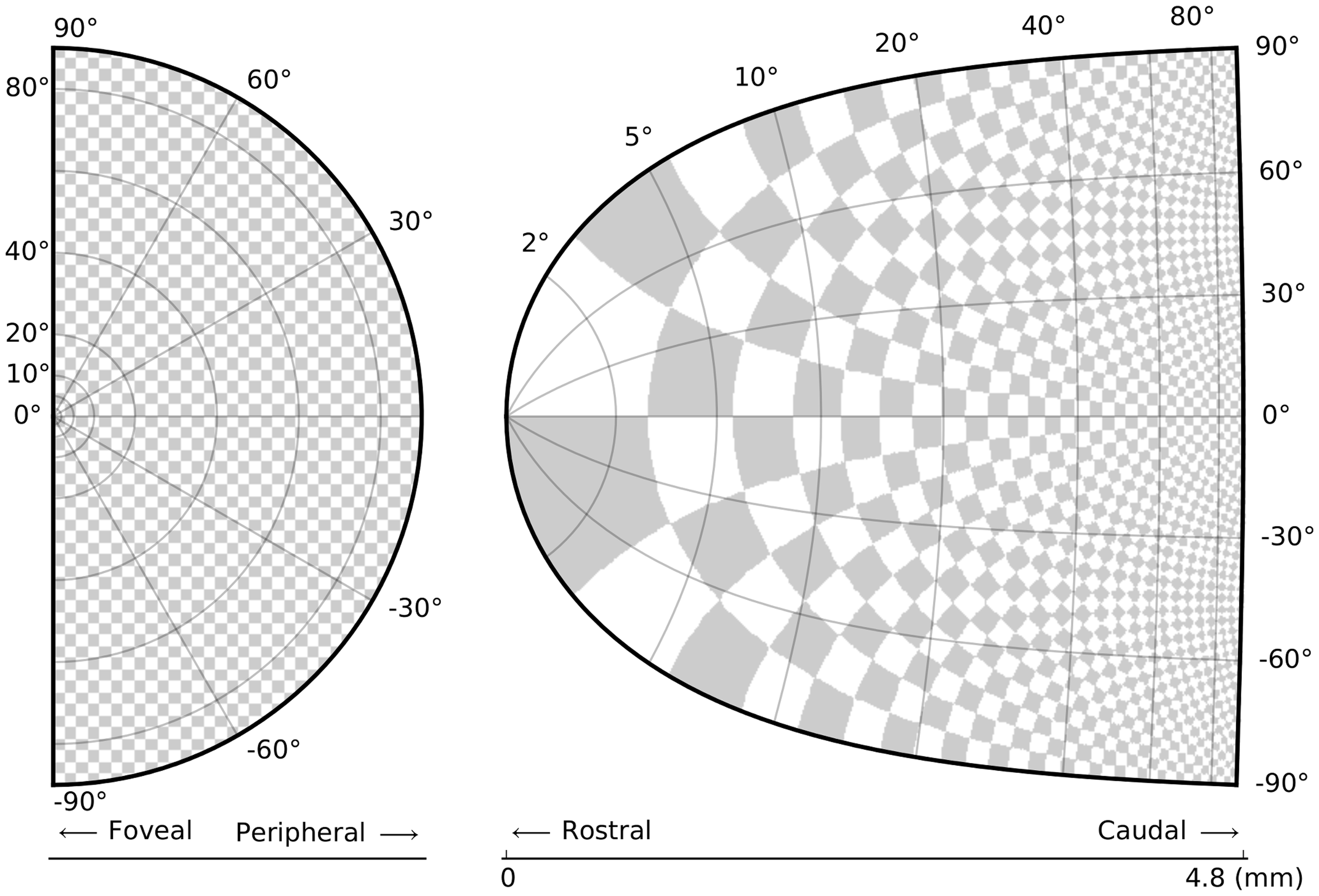
図は、記事であれ、期間限定の口頭発表であれ、言葉だけで説明するには長すぎる(またはほぼ不可能な)アイデアを表現したり、いくつかの事実や結果を紹介したりすることを目的としています。この文脈では、図の役割を明確に特定することが重要です。つまり、根底にあるメッセージは何であり、図はこのメッセージをどのように最もよく表現できるでしょうか。明確に識別されると、このメッセージは、図2に示すように、図の設計の強力なガイドになります。メッセージを特定した後でのみ、テキストの要点を決定した後でのみ記事を書くときに単語や文章を作成するのと同じように、図を作成する価値があります。あなたの姿が一目で印象的なメッセージを伝えることができれば、あなたの記事がコミュニティからより多くの注目を集める可能性が高くなります。
(図2)
上丘(SC)は、複数の機能経路の交差点にある脳幹構造です。いくつかの神経生理学的研究は、SC内のアクティブなニューロンの集団が衝動性眼球運動を誘発する視覚標的の位置をコード化することを示唆しています。網膜表面(左側)から丘状表面(右側)への投影は、対数マッピング関数が網膜座標から丘状座標への投影を保証する標準的かつ定量的なモデルに基づいています。この対数マッピングは、サッカードの決定において主要な役割を果たします。この役割をわかりやすく説明するために、実験中にこのようなパターンは使用されていませんが、人工の市松模様が使用されています。この市松模様は、中心窩領域の極端な拡大を明確に示しています。
ルール3:フィギュアをサポートメディアに適合させる
図形は、ポスター、コンピューターモニター、投影画面(口頭発表の場合)、または単純な紙(印刷物の場合)など、さまざまなメディアに表示できます。これらのメディアはそれぞれ、フィギュアのさまざまな物理的サイズを表していますが、さらに重要なことに、それぞれがフィギュアの表示方法や操作方法も異なります。たとえば、口頭発表では、期間限定でフィギュアが表示されます。したがって、視聴者は、説明を聞きながら、何が表示され、何を表しているのかをすばやく理解する必要があります。このような状況では、図3に示すように、注意を引くために、図を単純に保ち、メッセージを視覚的に目立たせる必要があります。。また、口頭発表では、人物がビデオで映し出され、遠くから見えるため、人物の要素を太く(線)または大きく(点、テキスト)し、色を強くする必要があることにも注意してください。対照的に、縦のテキストなどは避ける必要があります。ジャーナル記事の場合、読者は必要なだけ図を見ることができるため、状況はまったく異なります。これは、キャプションに補足的な説明とともに、多くの詳細を追加できることを意味します。コンピュータ画面で記事を読む人が増えていることを考慮すると、図をズームしたりドラッグしたりすることもできます。理想的には、サポート媒体の種類ごとに異なる図が必要であり、記事から図を抽出してそのまま置くという慣習を放棄する必要があります。
ルール4:キャプションはオプションではない
実験のセットアップを説明する場合でも、新しいモデルを導入する場合でも、新しい結果を提示する場合でも、図自体のすべてを説明することはできません。図にはキャプションを付ける必要があります。キャプションは、図の読み方を説明し、グラフィカルに表現できないものの精度を高めます。これは、口頭発表やポスターの前での説明と考えることができますが、人々が尋ねる質問について事前に考えなければならないという違いがあります。たとえば、棒グラフがある場合は、図の相対的な高さを調べて測定するだけで、読者がさまざまな棒の値を推測することを期待しないでください。数値が重要な場合は、記事の他の場所に記載するか、図に非常に明確に記載する必要があります。同様に、
ルール5:デフォルトを信頼しない
プロットライブラリまたはソフトウェアには、一連のデフォルト設定が付属しています。エンドユーザーが何も指定しない場合、これらのデフォルト設定を使用して、サイズ、フォント、色、スタイル、目盛り、マーカーなどを指定します(図4)。事実上すべての設定を指定でき、これらのデフォルト設定を選択することで、通常、各ソフトウェアパッケージ(Matlab、Excel、Keynoteなど)またはライブラリ(LaTeX、matplotlib、gnuplotなど)の特定のスタイルを認識できます。これらの設定は事実上すべてのタイプのプロットに使用されるため、特定のタイプのプロット用に微調整されることはありません。言い換えれば、それらはどのプロットにも十分ですが、誰にも最適ではありません。すべてのプロットでは、メッセージをより適切に表現するために、さまざまな設定を少なくとも手動で調整する必要があります。これは、正確なプロットを幅広い対象者にとってより目立たせるため、またはデータの性質に最適なカラーマップを選択するためです。
図4
ルール6:色を効果的に使用する
省略(TBA?)
ルール7:読者を誤解させない
省略(TBA?)
ルール8:「Chartjunk」を避ける
省略(TBA?)
ルール9:メッセージは美しさを打ち負かす
省略(TBA?)
ルール10:適切なツールを入手する
省略(TBA?)
図1を生成したPythonプログラム
# -*- coding: utf-8 -*-
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
import matplotlib.patches as patches
import japanize_matplotlib
# ----------
# Data to be represented
diseases = ["腎臓がん", "膀胱がん", "食道がん",
"卵巣がん", "肝臓がん", "非ホジキンリンパ腫",
"白血病", "前立腺がん", "膵臓がん",
"乳がん", "結腸直腸がん", "肺がん"]
men_deaths = [10000, 12000, 13000, 0, 14000, 12000,
16000, 25000, 20000, 500, 25000, 80000]
men_cases = [30000, 50000, 13000, 0, 16000, 30000,
25000, 220000, 22000, 600, 55000, 115000]
women_deaths = [6000, 5500, 5000, 20000, 9000, 12000,
13000, 0, 19000, 40000, 30000, 70000]
women_cases = [20000, 18000, 5000, 25000, 9000, 29000,
24000, 0, 21000, 160000, 55000, 97000]
# ----------
# Choose some nice colors
matplotlib.rc('axes', facecolor = 'white')
matplotlib.rc('figure.subplot', wspace=.65)
matplotlib.rc('grid', color='white')
matplotlib.rc('grid', linewidth=1)
# Make figure background the same colors as axes
fig = plt.figure(figsize=(12,7), facecolor='white')
# ---WOMEN data ---
axes_left = plt.subplot(121)
# Keep only top and right spines
axes_left.spines['left'].set_color('none')
axes_left.spines['right'].set_zorder(10)
axes_left.spines['bottom'].set_color('none')
axes_left.xaxis.set_ticks_position('top')
axes_left.yaxis.set_ticks_position('right')
axes_left.spines['top'].set_position(('data',len(diseases)+.25))
axes_left.spines['top'].set_color('w')
# Set axes limits
plt.xlim(200000,0)
plt.ylim(0,len(diseases))
# Set ticks labels
plt.xticks([150000, 100000, 50000, 0],
['150,000', '100,000', '50,000', '女性'])
axes_left.get_xticklabels()[-1].set_weight('bold')
axes_left.get_xticklines()[-1].set_markeredgewidth(0)
for label in axes_left.get_xticklabels():
label.set_fontsize(10)
plt.yticks([])
# Plot data
for i in range(len(women_deaths)):
H,h = 0.8, 0.55
# Death
value = women_cases[i]
p = patches.Rectangle(
(0, i+(1-H)/2.0), value, H, fill=True, transform=axes_left.transData,
lw=0, facecolor='red', alpha=0.1)
axes_left.add_patch(p)
# New cases
value = women_deaths[i]
p = patches.Rectangle(
(0, i+(1-h)/2.0), value, h, fill=True, transform=axes_left.transData,
lw=0, facecolor='red', alpha=0.5)
axes_left.add_patch(p)
# Add a grid
axes_left.grid()
plt.text(165000,8.2,"がんによる死亡の主な原因", fontsize=18,va="top")
plt.text(165000,7,"""2007年に米国で140万を超える\n"""
"""新しい癌の症例がありました。""", va="top", fontsize=10)
# --- MEN data ---
axes_right = plt.subplot(122, sharey=axes_left)
# Keep only top and left spines
axes_right.spines['right'].set_color('none')
axes_right.spines['left'].set_zorder(10)
axes_right.spines['bottom'].set_color('none')
axes_right.xaxis.set_ticks_position('top')
axes_right.yaxis.set_ticks_position('left')
axes_right.spines['top'].set_position(('data',len(diseases)+.25))
axes_right.spines['top'].set_color('w')
# Set axes limits
plt.xlim(0,200000)
plt.ylim(0,len(diseases))
# Set ticks labels
plt.xticks([0, 50000, 100000, 150000, 200000],
['男性', '50,000', '100,000', '150,000', '200,000'])
axes_right.get_xticklabels()[0].set_weight('bold')
for label in axes_right.get_xticklabels():
label.set_fontsize(10)
axes_right.get_xticklines()[1].set_markeredgewidth(0)
plt.yticks([])
# Plot data
for i in range(len(men_deaths)):
H,h = 0.8, 0.55
# Death
value = men_cases[i]
p = patches.Rectangle(
(0, i+(1-H)/2.0), value, H, fill=True, transform=axes_right.transData,
lw=0, facecolor='blue', alpha=0.1)
axes_right.add_patch(p)
# New cases
value = men_deaths[i]
p = patches.Rectangle(
(0, i+(1-h)/2.0), value, h, fill=True, transform=axes_right.transData,
lw=0, facecolor='blue', alpha=0.5)
axes_right.add_patch(p)
# Add a grid
axes_right.grid()
# Y axis labels
# We want them to be exactly in the middle of the two y spines
# and it requires some computations
for i in range(len(diseases)):
x1,y1 = axes_left.transData.transform_point( (0,i+.5))
x2,y2 = axes_right.transData.transform_point((0,i+.5))
x,y = fig.transFigure.inverted().transform_point( ((x1+x2)/2,y1) )
plt.text(x, y, diseases[i], transform=fig.transFigure, fontsize=10,
horizontalalignment='center', verticalalignment='center')
# Devil hides in the details...
arrowprops = dict(arrowstyle="-",
connectionstyle="angle,angleA=0,angleB=90,rad=0")
x = women_cases[-1]
axes_left.annotate('新たな症例', xy=(.9*x, 11.5), xycoords='data',
horizontalalignment='right', fontsize= 10,
xytext=(-40, -3), textcoords='offset points',
arrowprops=arrowprops)
x = women_deaths[-1]
axes_left.annotate('死亡例', xy=(.85*x, 11.5), xycoords='data',
horizontalalignment='right', fontsize= 10,
xytext=(-50, -25), textcoords='offset points',
arrowprops=arrowprops)
x = men_cases[-1]
axes_right.annotate('新たな症例', xy=(.9*x, 11.5), xycoords='data',
horizontalalignment='left', fontsize= 10,
xytext=(+40, -3), textcoords='offset points',
arrowprops=arrowprops)
x = men_deaths[-1]
axes_right.annotate('死亡例', xy=(.9*x, 11.5), xycoords='data',
horizontalalignment='left', fontsize= 10,
xytext=(+50, -25), textcoords='offset points',
arrowprops=arrowprops)
# Done
plt.savefig('figure-1.pdf')
plt.show()
おわりに
データの可視化 / 視覚化を頑張っていきたいなと思いました。
Notes
All the figures for this article were produced using matplotlib, and figure scripts are available from https://github.com/rougier/ten-rules.