概要
- RPostgreSQLライブラリでPostgresqlからRstudioへデータをインポート
- インポートしたデータをtidyrライブラリを使って主成分分析できるようにピボットする
-
prcompライブラリで主成分分析する
環境
- RStudio
- R
手順
Rstudioのインストール
- RstudioとRは下記でインストールする
デモデータ
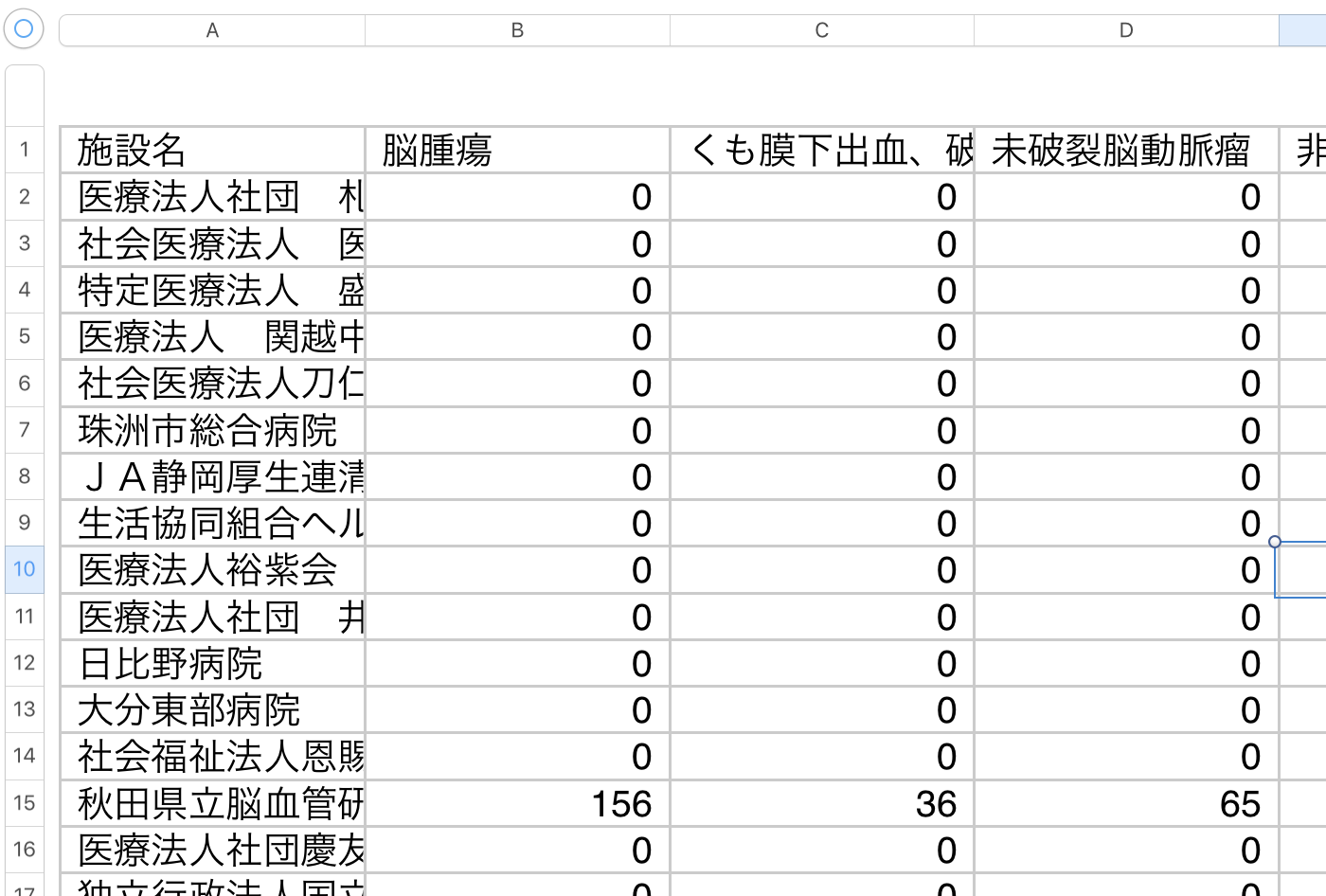
- 全国のDPC病院の疾患別・手術別集計を使います
- 今回の解析の目的は: 神経系疾患病院を分類する際の評価軸の検討としました。
データ前処理
前記集計には、入院期間と患者件数データがあります。今回は、患者件数データを対象とします。
患者件数は様々な治療種類毎のデータが入っています。今回は事前に治療種類毎の単純合計値、主成分解析の解析対象としました。
また、さらに、有効なデータが少ない、病気および病院は除去しました。(詳細は省略)
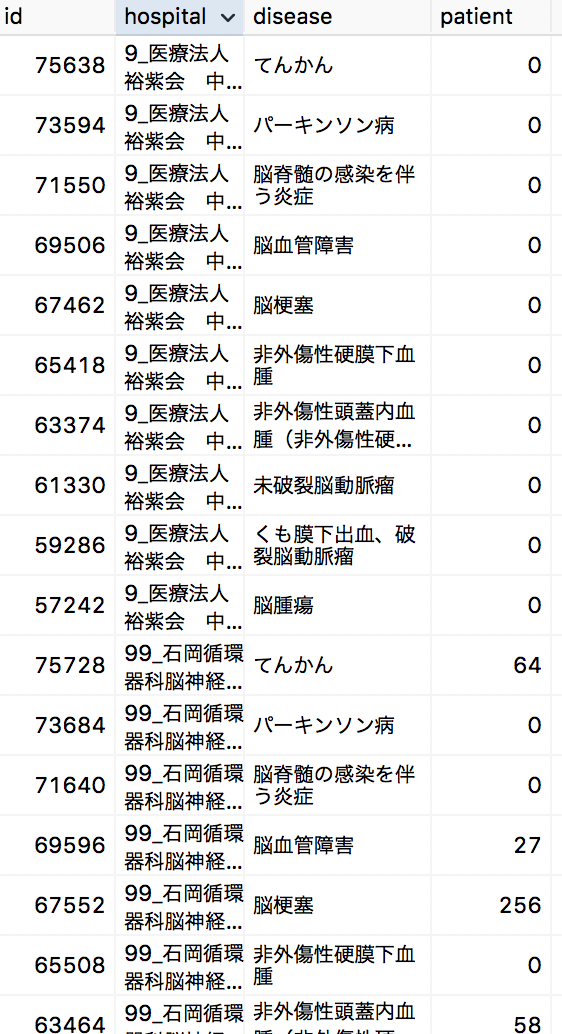
データをPostgresqlへ投入
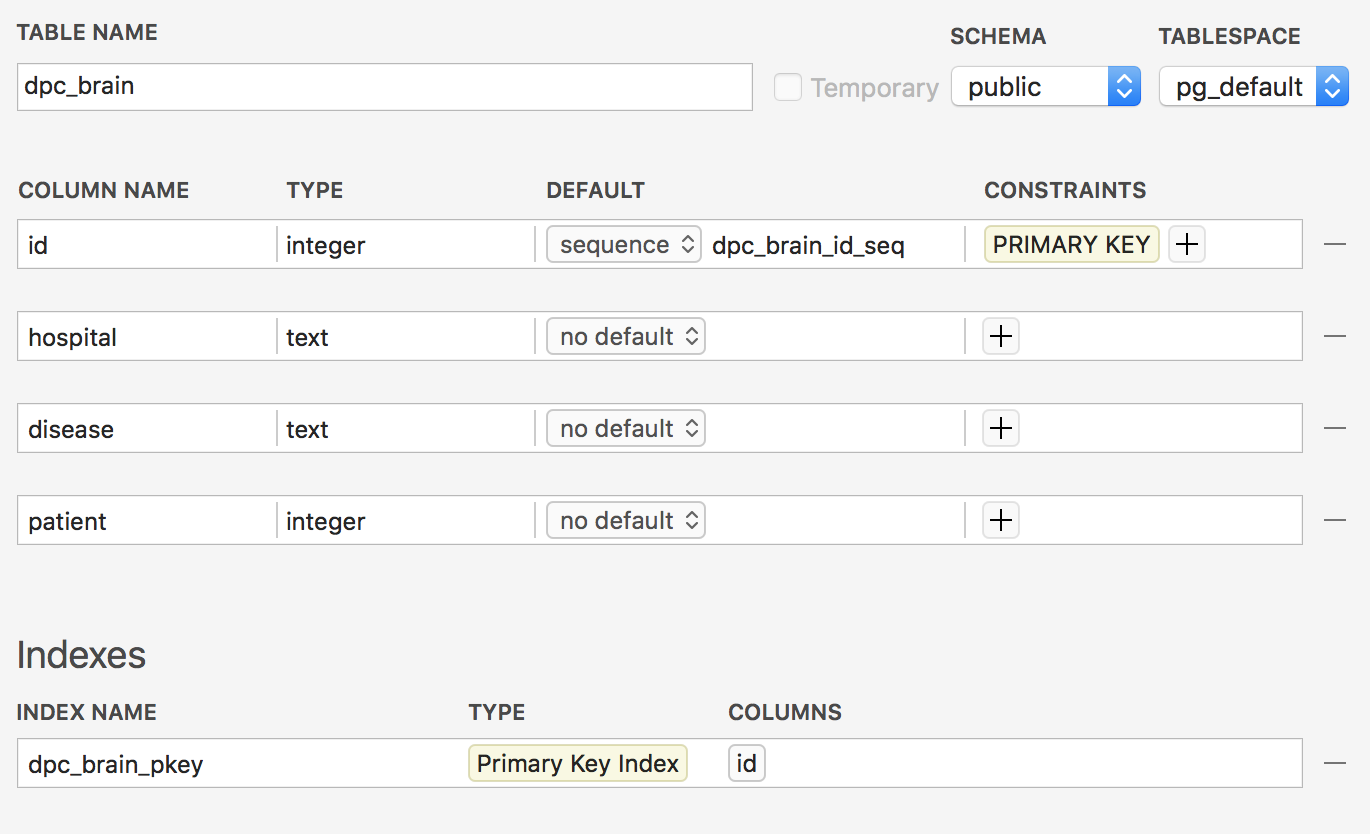
- dpcというユーザー名でdpcというデータベースとdpc_brainというテーブルを用意します
- dpc_brainテーブルには、hospital(病院名)とdisease(病気名)とpatient(患者件数合計)というカラムを用意します。
- データを投入します。
Postgresqlのデータを読み込む
- PostgreSQLのテーブルをRのデータフレームに一瞬で流しこむ方法の通りです。
- dbnameとuserとpasswordは今回のデータベースにあわせます。
install.packages("RPostgreSQL")
require(RPostgreSQL)
drv <- dbDriver("PostgreSQL")
con <- dbConnect(drv,dbname="dpc",host="localhost",port=5432,user="dpc",password="")
dpc_brain<- dbReadTable(con,"dpc_brain")
分析しやすいようにデータを変換する
使用するデータだけを抽出
1列目はidなので、2列名以降(hospital, disease, patient)を選択して selected_dpc_brainに入れる
selected_dpc_brain <- dpc_brain[,2:4]
データをピボットする
主成分分析等、統計解析する際は、各列は変数ごとになっていることを想定してなされることがほとんどです。今回の場合もdisease列の各病気名を列名とするように、selected_dpc_brainをピボットする必要があります。
そのためにtidyrをつかいます。
spreaded_dpc_brain <- tidyr::spread(selected_dpc_brain, key = "disease", value = "patient")
その他、tidyrの細かな使い方は、こちらのtidyr::gather( )とtidyr::spread( )でデータフレームを自在に変形するを参照してください。
ラベルをつける
普通に読むこむと各行のラベルは連番(1, 2, 3...)です。これでも解析上は問題ないのですが、結果をみるとき不便なので、病院名をラベル(行名、rownames)にします。
1列目が病院名なので、それを行名として投入します。
rownames(spreaded_dpc_brain) <- spreaded_dpc_brain[,1]
解析する
グラフ内で日本語が表示できるようにする
こちらのページからコードを抜粋し、実行します。これをやらないと文字が豆腐になってしまいます。
par(family = "HiraKakuProN-W3")
基礎データ
summaryを見ます。
summary(spreaded_dpc_brain[,2:11])
くも膜下出血、破裂脳動脈瘤 てんかん パーキンソン病 脳血管障害 脳梗塞 脳腫瘍 脳脊髄の感染を伴う炎症
Min. : 0.000 Min. : 0.00 Min. : 0.000 Min. : 0.00 Min. : 0.0 Min. : 0.00 Min. : 0.000
1st Qu.: 0.000 1st Qu.: 0.00 1st Qu.: 0.000 1st Qu.: 0.00 1st Qu.: 19.0 1st Qu.: 0.00 1st Qu.: 0.000
Median : 0.000 Median : 16.00 Median : 0.000 Median : 0.00 Median : 54.0 Median : 0.00 Median : 0.000
Mean : 4.446 Mean : 30.97 Mean : 6.996 Mean : 16.46 Mean : 99.6 Mean : 19.68 Mean : 6.393
3rd Qu.: 0.000 3rd Qu.: 43.00 3rd Qu.: 0.000 3rd Qu.: 13.00 3rd Qu.:151.2 3rd Qu.: 16.00 3rd Qu.: 11.000
Max. :157.000 Max. :2428.00 Max. :363.000 Max. :649.00 Max. :882.0 Max. :561.00 Max. :123.000
非外傷性硬膜下血腫 非外傷性頭蓋内血腫(非外傷性硬膜下血腫以外) 未破裂脳動脈瘤
Min. : 0.000 Min. : 0.00 Min. : 0.00
1st Qu.: 0.000 1st Qu.: 0.00 1st Qu.: 0.00
Median : 0.000 Median : 10.00 Median : 0.00
Mean : 5.188 Mean : 25.65 Mean : 14.72
3rd Qu.: 0.000 3rd Qu.: 40.00 3rd Qu.: 10.00
Max. :109.000 Max. :308.00 Max. :721.00
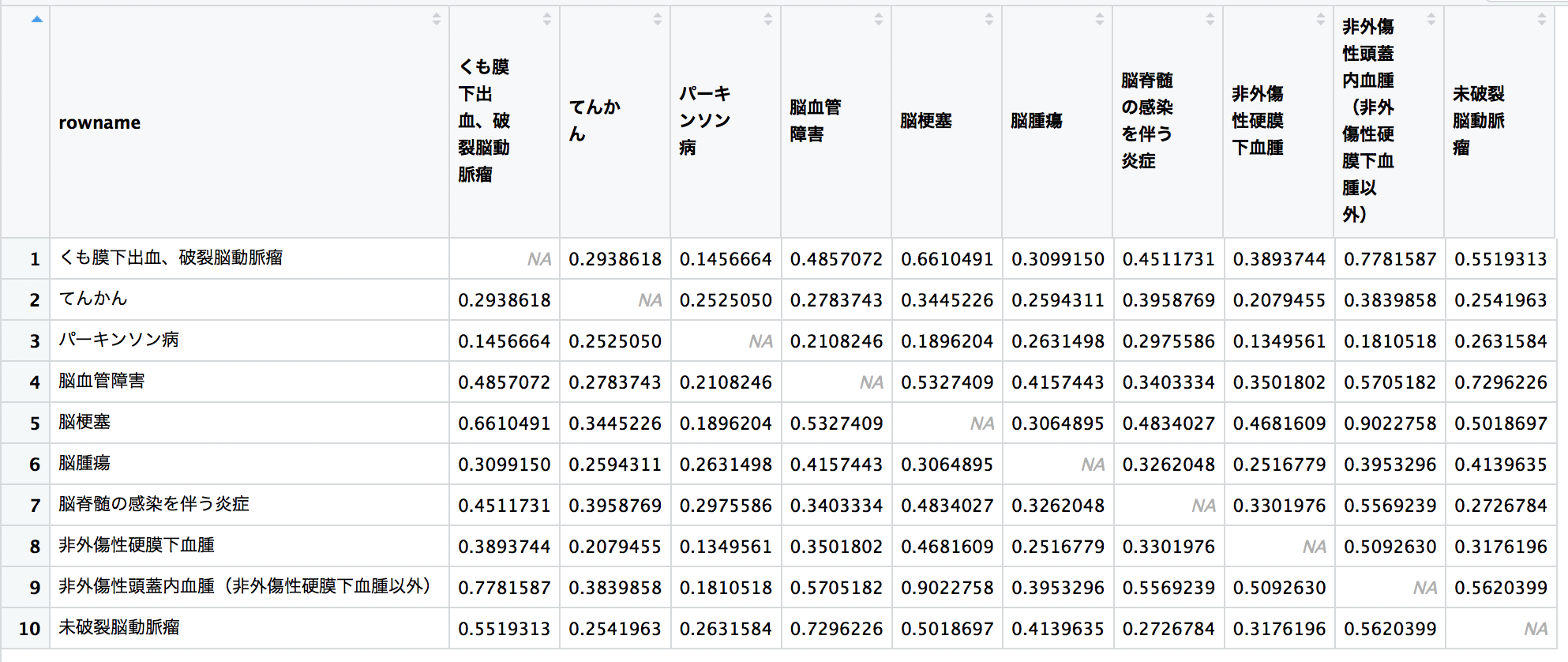
相関係数行列
各病気間の相関係数をみます。
pairs(spreaded_dpc_brain[,2:10])
library(corrr)
dpc_brain_correlate <- correlate(spreaded_dpc_brain[,2:11])
主成分分析
prcompを使うだけ。
pca <- prcomp(spreaded_dpc_brain[,c(2:11)],scale=T)
summaryを見る。
> summary(pca)
Importance of components:
PC1 PC2 PC3 PC4 PC5 PC6 PC7 PC8 PC9 PC10
Standard deviation 2.1683 1.0566 0.99355 0.8526 0.82848 0.79096 0.69449 0.60600 0.48495 0.26669
Proportion of Variance 0.4702 0.1116 0.09871 0.0727 0.06864 0.06256 0.04823 0.03672 0.02352 0.00711
Cumulative Proportion 0.4702 0.5818 0.68051 0.7532 0.82185 0.88441 0.93265 0.96937 0.99289 1.00000
Cumulative Propotionをみると主成分4ぐらいまで軸がありそうです。
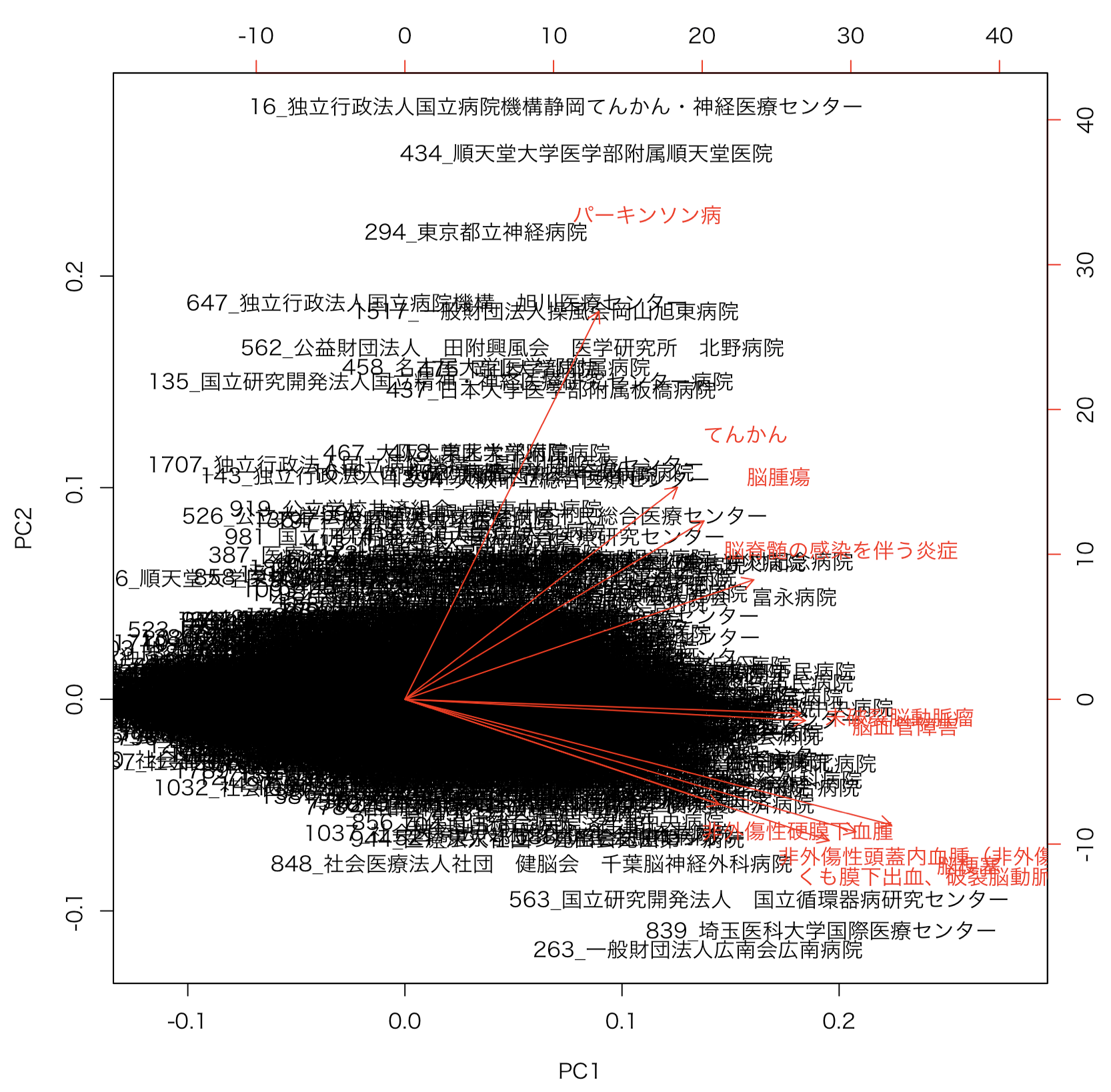
結果をグラフにプロットしてみます。
biplot(pca)
赤い矢印の軸をみると、脳の病気に関して、病院を分類する際は、4種類ぐらい評価軸がありそうなことがわかりました。
Extra 主成分分析はRubyでもできるよ!
主成分分析をrubyでやろうとしても、デファクトスタンダードなライブラリがありません。gem pcaで調べると2つぐらいでますが、スター数が少ないので、不安・・・
しかし、主成分分析の実装は簡単です。計算はたった2つなので。
- 相関係数行列(標準化されたデータの分散共分散行列)を計算する!
- 上記行列の固有値ベクトルを計算する!
(理論は、 主成分分析 固有値ベクトル とかでググってください)
1,2いずれも普通の行列計算ソフトであれば実装されているものです。
今回は、演算が速いとされている日本製のライブラリNumoを使った例を示します。(標準のmatrixでもできます。)
まず、正解例としてRを使って計算します。(なお、計算を簡単にするため今回は相関係数行列ではなく、分散共分散行列を使った例です。)
> a <- data.frame(x1=c(1,4,2), x2=c(4,8,3))
> var(a)
x1 x2
x1 2.333333 3.5
x2 3.500000 7.0
> pca <- prcomp(a,scale=F)
> pca
Standard deviations (1, .., p=2):
[1] 2.9787822 0.6783732
Rotation (n x k) = (2 x 2):
PC1 PC2
x1 0.4718579 0.8816746
x2 0.8816746 -0.4718579
次にruby の例を示します。
irb(main):002:0> require 'numo/narray'
=> true
irb(main):003:0> require 'numo/linalg'
=> true
irb(main):004:0> a = Numo::DFloat.cast([[1,4,2], [4,8,3]])
=> Numo::DFloat#shape=[2,3]
[[1, 4, 2],
[4, 8, 3]]
irb(main):005:0> a.cov
=> Numo::DFloat#shape=[2,2]
[[2.33333, 3.5],
[3.5, 7]]
irb(main):007:0> Numo::Linalg.eig(a.cov)
=> [Numo::DComplex#shape=[2]
[0.46019+0i, 8.87314+0i], nil, Numo::DComplex#shape=[2,2]
[[-0.881675+0i, -0.471858+0i],
[0.471858+0i, -0.881675+0i]]]
値が一致するので、主成分分析ができることがわかりました。