概要
クラスタリングは簡易であり,有益な結果を得やすいデータ分析の手法です.
もともとネットワーク構造のであるデータはもちろんのこと,ネットワーク構造でないデータに関しても距離関数を定義することでネットワーク化し,クラスタリングをすることができます.
このエントリではクラスタリングを行い,その結果を可視化する方法について紹介します.
ネットワークとは
そもそもネットワーク構造とはなんでしょうか?
一般的にはノードとエッジから構成されるデータのことであり,
エッジには方向がついていたりついていなかったりします
方向があるものを有向グラフ, ないものを無向グラフといいます.
エッジに重みがあるものもないものもあり,あるものを重み付きグラフといいます.
クラスタリングとは
データの集合をいくつかのまとまり(部分集合)に分けることです
それぞれの部分集合がある共通の特徴を持つように分けます
ネットワーク分析においてはCommunity Detectionとよばれることもあります.
データを分析する際にそれぞれの関係性を計算し,ネットワーク化しそれをクラスタリングしてまとまりごとに可視化するという手法はそのデータの構造を把握するためによくとられますし,
知見を得やすく有用な手法です.
今回のエントリで紹介するアルゴリズムについて
今回紹介するアルゴリズムはネットワークのモジュラリティが最大になる分割をすることを目的としたアルゴリズムです.
モジュラリティとは,そのネットワークはランダムなネットワークよりどれだけ密集しているかを表す指標です.
2010年の比較論文で,ネットワークからのコミュニティ抽出ではもっともよい評価を得ています(http://arxiv.org/abs/0906.0612)
よく知られているクラスタリングアルゴリズムとしてはK-meansがあります.
K-meansはそれぞれのノードの近さを反映するため,一次のつながりのみが考慮されているアルゴリズムであるといえます.
今回紹介するアルゴリズムはネットワークに対してクラスタリングを行うものであり,
K-meansとの違いとしては
- 事前にクラスタ数を決定する必要がない
- 高次のつながりを反映することができる
という点になります.
ライブラリについて
手法の論文
Fast unfolding of communities in large networks, Vincent D Blondel, Jean-Loup Guillaume, Renaud Lambiotte, Renaud Lefebvre, Journal of Statistical Mechanics: Theory and Experiment 2008(10)
インストール
以下のBitbucketから
サンプルコード
karate_clubと言われる社会ネットワークの代表的なデータセットの可視化を行います.
import community
import networkx as nx
import matplotlib.pyplot as plt
G = nx.karate_club_graph()
partition = community.best_partition(G)
size = float(len(set(partition.values())))
pos = nx.spring_layout(G)
count = 0.
for com in set(partition.values()):
count += 1.
list_nodes = [nodes for nodes in partition.keys() if partition[nodes] == com]
nx.draw_networkx_nodes(G, pos, list_nodes, node_size=20, node_color = str(count/size) )
plt.show()
グラフ可視化ツールを使おう
上記のようにPythonのみでもグラフを描画することは可能ですが,
ネットワークが大規模になった際には,一番大きな連結グラフのみにしたり,一定の重み以下のエッジを消したりするなど,描画おいてインタラクティブな操作ができると便利なことがたくさんあります.
グラフ可視化ツールにはいくつかありますが,今回はCytoScapeを例にクラスタリングの結果を可視化ツールに反映させることについて紹介します.
今回紹介したライブラリはNetworkXベースで表現されており,
NetworkXで表されたネットワークは様々な可視化ソフトと連携できるデータ形式で書きだすことができるようになっています.
今回はGraphML形式で書きだしてみましょう
コミュニティ情報をどう書き出すか?
ノードには様々な属性情報を持たせることができます.
ノードIDをkeyにもつdictを渡すことで与えることが可能です.
これによりそれぞれのノードがどのようにクラスタリングされたかをデータとして保持することができます.
こちらをGML形式で書き出します.
import community
import networkx as nx
G = nx.karate_club_graph()
partition = community.best_partition(G)
labels = dict([(i, str(i)) for i in xrange(nx.number_of_nodes(G))])
nx.set_node_attributes(G, 'label', labels)
nx.set_node_attributes(G, 'community', partition)
nx.write_gml(G, "community.gml")
CytoScapeを使ってデータを読み込む
起動後のダイアログのFrom Network Fileからファイルを指定して読み込みます
(もしくはFile->import->Network->File)
よくわかんないのが表示されたと思いますが,これはレイアウト指定してないからです.
まず表示アルゴリズムを指定しましょう.
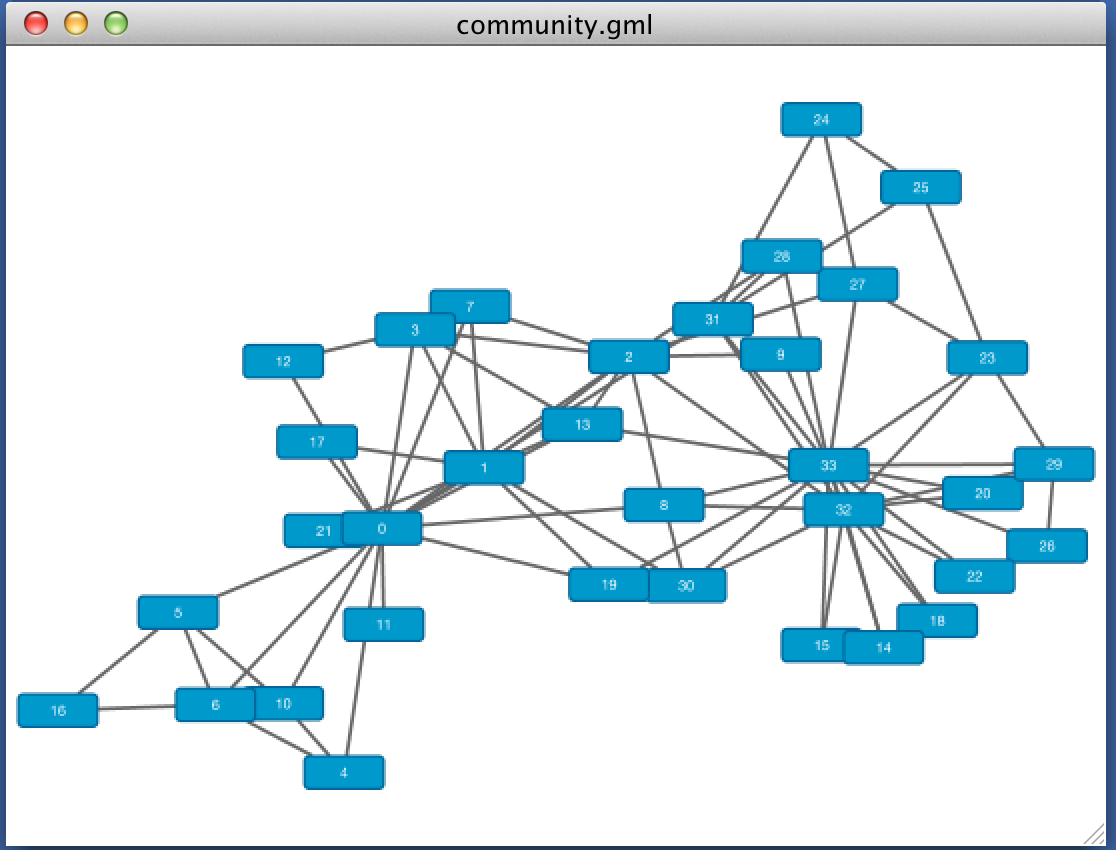
Layout->yFiles Layouts->Organicを選択すると表示が以下のようになります
それっぽい感じで表示されましたね.
クラスタリングの結果を可視化に加える
続いてクラスタリングの結果をこのグラフに反映させます.
今回はクラスタに応じてノードの色を変えてみましょう.
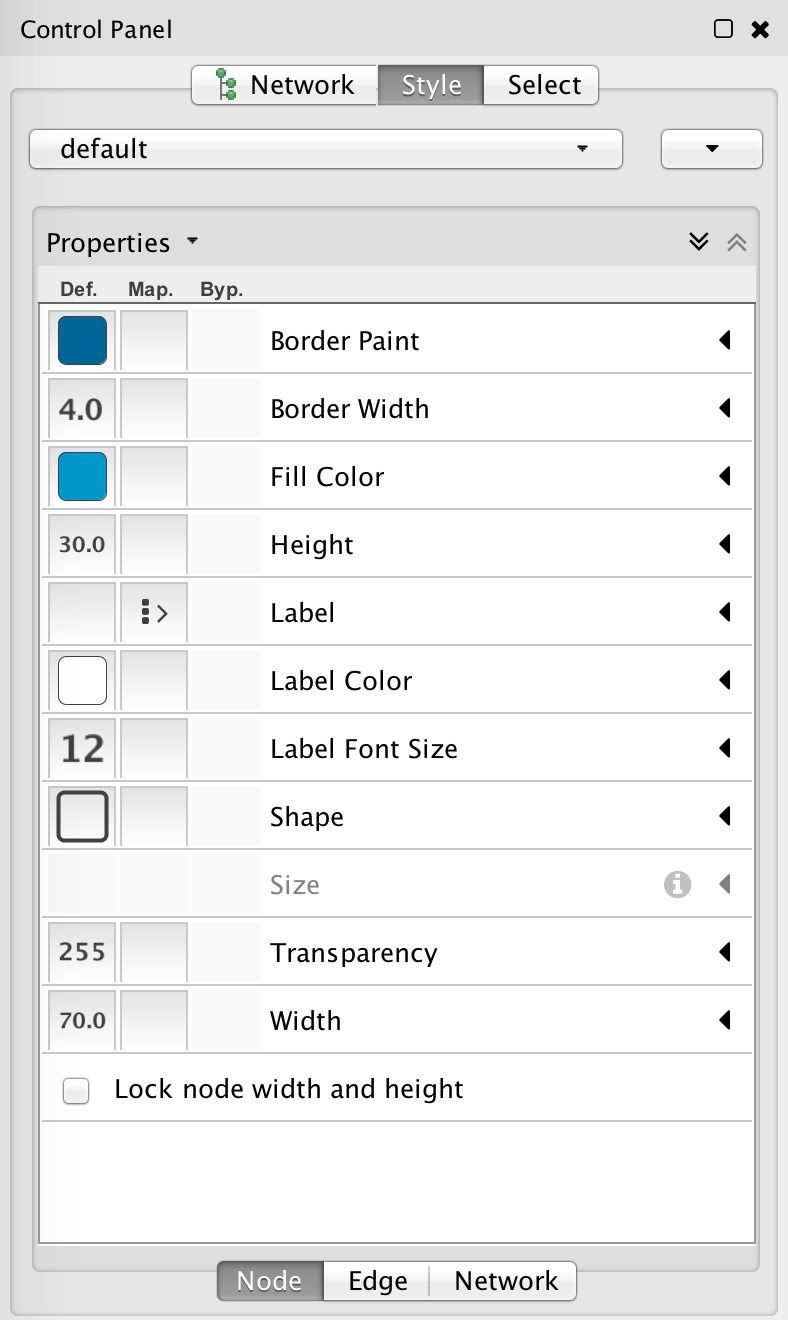
グラフの色や大きさなどを変えるときにはControlPanelのStyleメニューを使います.
Control Panelが出ていない時はView->Show Contorol Panelで表示させてください.
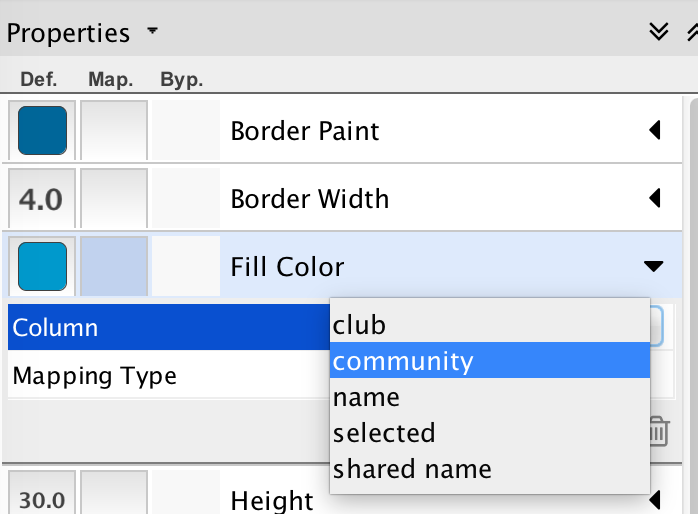
色を変えるにはFill Colorを使います.
mapでどのような条件で設定を割り当てるかを決めることができます.
変えたい項目のMapのところをクリックすると
このような項目が表示されます.
Columnのところに条件になるデータ項目を設定します.
set_node_attributesで設定した項目が選択できます.
communityを選択しましょう
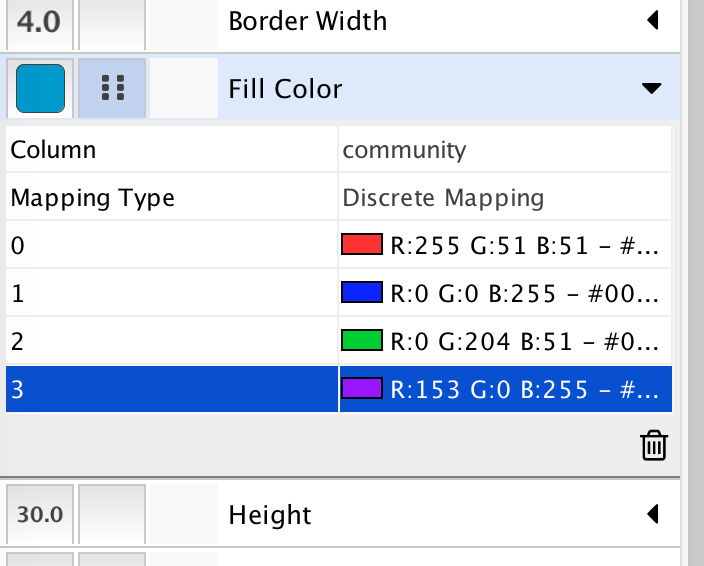
MappingTypeでどのような条件で定義を変えるかを選びます
Typeには3種類あり
- Continuous Mapping: 値を連続値とし,大小によってサイズや色のグラデーションを変える
- Discrete Mapping: 値を離散値とし,それぞれの値に対して色を設定する
- PassThrough Mapping: 値をそのままデータにする.例えばredなどと指定すると赤色になる
今回はクラスタ数も4つと少く,値の大小にも意味がないのでDiscrete Mappingを選択します
選択すると以下のようなプルダウンメニューが出てくるので色を選択して指定します.
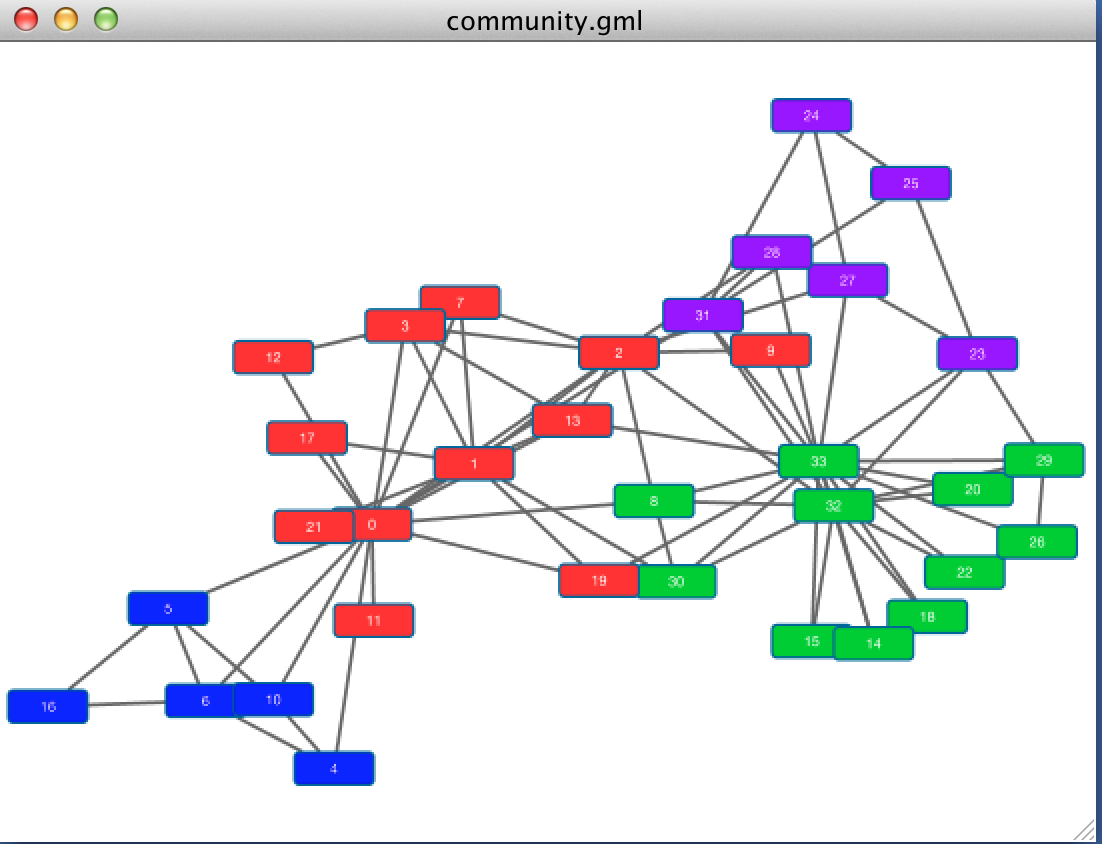
このように設定すると最初のグラフの色がこのように変わっています.
クラスタリングの結果を可視化することができました.
まとめ
本エントリではネットワークのクラスタリングから可視化までの流れについて紹介しました.
ネットワークの分析は比較的容易でありながら,面白い結果が得やすい手法でありますのでぜひ興味を持たれた皆様は挑戦してみてください!