概要
樹木の病気の判定を人手でやっているのだが、人手不足でその業務を自動化できないかということがそもそものお話の始まりです。植物の病気は色々な形で現れますが、とっかかりとして枯れている木を診断できないかどうかを機械学習で作成してみました。画像認識の領域になるので、画像処理で特徴抽出して、SVMのようなロジックで分類器を構成するというのが一般的なやり方になると思います。
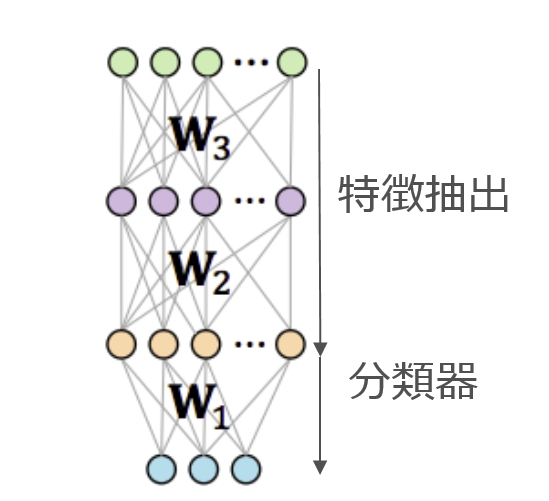
でもそのようなやり方は特徴抽出の方法に依存し、その後の展開に問題が発生しそうです。そのような事情でDeep Learningを使ってみることにしました。ただ、一から、学習させるととても時間がかかります。そのことから転移学習という手法を使ってみることにします。Deep Learrningは大枠特徴抽出している部分と分類している部分にわかれます。分類器に入る直前の最後の全結合層が一番特徴を表していると考えられています。その最終の全結合層を取り出して、分類器にかければ、任意の画像の認識に使えるという手法です。
ResNet
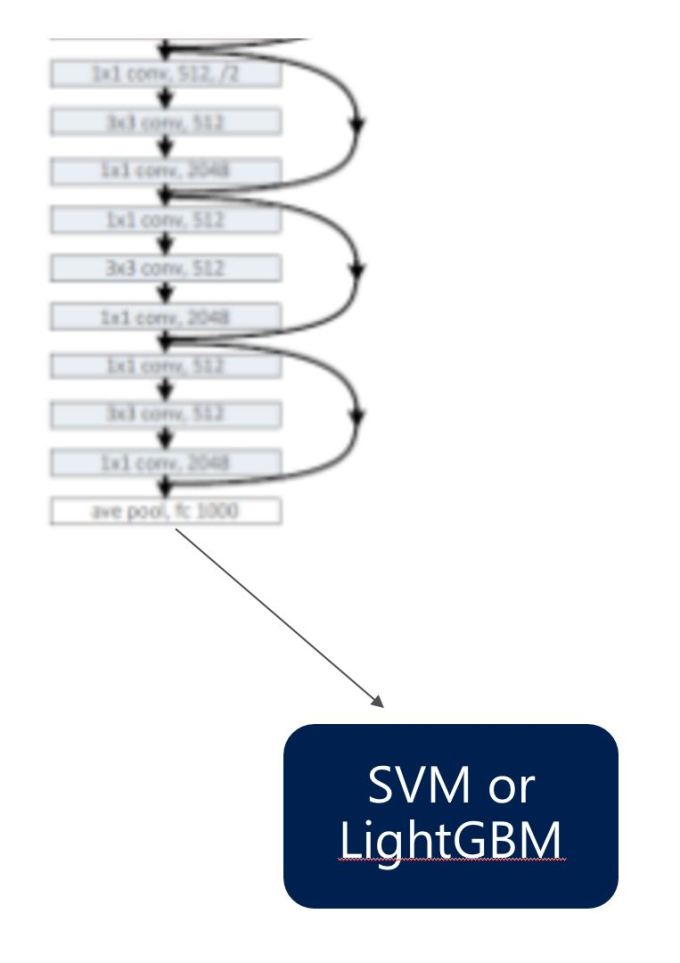
今一番認識率の高いモデルと言われているマイクロソフトで開発されたResNetがあります。ResNetの最終の全結合層は512個のパラメータになります。学習画像から生成された512のパラメータを使って、分類器のみを学習させれば、最初からモデルを学習させる必要はなく、画像認識の仕組みを構築することが可能になります。分類器はNeural Networkである必要はなく、SVMやLightGBMのような機械学習のクラス分類手法の利用が可能です。
処理の方針
1.ResNetのモデルから最後の全結合層を取り出す
2.取り出した特徴とラベルから、分類器を学習させる
最初の全結合層の取り出しですが、CNTKのサンプルにFeastureExtractionというものが付属しています。それを使って特徴を取り出します。
CNTK2.0-rc1\cntk\Examples\Image\FeatureExtractionのFeatureExtraction.pyがそれにあたります。
動かす準備
GPUを使用を明示します。下の2行をファイルの最初に追加します。
from cntk.io import MinibatchSource, ImageDeserializer, StreamDefs, StreamDef
from cntk.device import set_default_device, gpu
また、一部モデルからの返り値の型が、Betaから仕様が変更されているようです。
def eval_and_write(model_file, node_name, output_file, minibatch_source, num_objects):
上記の定義内に
out_values = output[0,0].flatten()
となっていますが、これだと動作しないので、
下記に修正します。
out_values=np.array(output).flatten()
また、下記の場所にGPUを利用するための、デバイス設定の呼び出しを追加します。
if __name__ == '__main__':
set_default_device(gpu(0))
これで、動作可能になります。あとはデータの読み込みを設定します
まず、ResnetのPretrainedモデルをダウンロードします。データもダウンロードされますが、下記のコマンドを実行することでモデルがダウンロードされます。
cd .\CNTK2.0-rc1\Examples\Image\FeatureExtraction
Python install_data_and_model.py
CNTK-2.0-rc1\cntk\Examples\Image\DataSets\Groceryにtext.txtファイルが存在し、こちらに特徴抽出するファイルのリストを書き込みます。ファイルの場所と名前を変更すれば、OKですこのプログラム内でデータを読み込むときにデータ変換のストリーム中に入れてサイズを変更しています。
ImageNetのPretrained Modelを使うことから224×224に変更する必要があります。元データもあまり大きいと読み込みに時間がかかったり、エラーになる可能性もあるので、予めHDサイズ(1920×1024)程度にそろえておくのがよいかと思います。
def create_mb_source(map_file, image_width, image_height, num_channels, num_classes, randomize=True):
transforms = [xforms.scale(width=image_width, height=image_height, channels=num_channels, interpolations='linear')]
return MinibatchSource(ImageDeserializer(map_file, StreamDefs(
features =StreamDef(field='image', transforms=transforms),
labels =StreamDef(field='label', shape=num_classes))),
randomize=randomize)
学習処理
layerOut.txtファイルが生成され、データ画像ごとの512の特徴量がCSV形式で出力されます。
データ画像ごとの枯れているかどうかのラベル
枯れている:1
枯れていない:0
として、layerOut.txtの最初のカラムに追加してCSVファイルを作成します
テスト用のデータはlayerOut2.txtとして
SVMで学習Pythonのコードは以下になります。
from sklearn import svm
import numpy as np
d_tmp = np.loadtxt('layerOut.txt', delimiter=',')
train_data = [x[1:] for x in d_tmp]
label = [int(x[0]) for x in d_tmp]
d_tmp_t = np.loadtxt('layerOut2.txt', delimiter=',')
test_data =[x[1:] for x in d_tmp_t]
treehelth =svm.LinearSVC(C=1.0)
treehelth.fit(train_data, label)
prediction = treehelth.predict(test_data)
print(prediction)
SVM部分をマイクロソフトの開発したLightGBMを使うこともできます。データ量が多い場合は学習が高速になります。
もともとの画像が問題があったようですが、認識率は70%弱というところです。おそらく、元データ側をチューニングする必要がありそうです。画像は学習データの一つです。