OLS、特徴選択、リッジ回帰、ラッソの4つの方法でTrainデータからモデルをFittingし、Testデータを用いて、平均2乗誤差(MSE)を推定して比べるといったことをします。
まずはデータの準備
データはStanford大学の統計学部からprostateのデータをダウンロード、正規化、訓練データ、テストデータに分けて使います。
> # Download prostate data
> con = "http://statweb.stanford.edu/~tibs/ElemStatLearn/datasets/prostate.data"
> prostate=read.csv(con,row.names=1,sep="\t")
>
> # Scale data and prepare train/test split
> prost.std <- data.frame(cbind(scale(prostate[,1:8]),prostate$lpsa))
> names(prost.std)[9] <- 'lpsa'
> data.train <- prost.std[prostate$train,]
> data.test <- prost.std[!prostate$train,]
> y.test <- data.test$lpsa
> n.train <- nrow(data.train)
OLS
単純な最小二乗法でMSEを求めてみます。
> m.ols <- lm(lpsa ~ . , data=data.train)
> summary(m.ols)
Call:
lm(formula = lpsa ~ ., data = data.train)
Residuals:
Min 1Q Median 3Q Max
-1.64870 -0.34147 -0.05424 0.44941 1.48675
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 2.46493 0.08931 27.598 < 2e-16 ***
lcavol 0.67953 0.12663 5.366 1.47e-06 ***
lweight 0.26305 0.09563 2.751 0.00792 **
age -0.14146 0.10134 -1.396 0.16806
lbph 0.21015 0.10222 2.056 0.04431 *
svi 0.30520 0.12360 2.469 0.01651 *
lcp -0.28849 0.15453 -1.867 0.06697 .
gleason -0.02131 0.14525 -0.147 0.88389
pgg45 0.26696 0.15361 1.738 0.08755 .
---
Signif. codes:
0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.7123 on 58 degrees of freedom
Multiple R-squared: 0.6944, Adjusted R-squared: 0.6522
F-statistic: 16.47 on 8 and 58 DF, p-value: 2.042e-12
> y.pred.ols <- predict(m.ols,data.test)
> summary((y.pred.ols - y.test)^2)
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.000209 0.044030 0.116400 0.521300 0.445000 4.097000
>
> mse.ols=sum((y.pred.ols - y.test)^2)
> mse.ols
[1] 15.63822
特徴選択
次に特徴量を減らしてみます。
leap関数でCp(Process Capability Index)をもとに特徴選択を行った上で、MSEを求めます。
whichで確認すると、gleasonを削ったモデルが良さそうだと判断されてます。
> library(leaps)
> l <- leaps(data.train[,1:8],data.train[,9],method='Cp')
> plot(l$size,l$Cp)
>
> # Select best model according to Cp
> bestfeat <- l$which[which.min(l$Cp),]
> bestfeat
1 2 3 4 5 6 7 8
TRUE TRUE TRUE TRUE TRUE TRUE FALSE TRUE
>
> # Train and test the model on the best subset
> m.bestsubset <- lm(lpsa ~ .,data=data.train[,bestfeat])
> summary(m.bestsubset)
Call:
lm(formula = lpsa ~ ., data = data.train[, bestfeat])
Residuals:
Min 1Q Median 3Q Max
-1.65425 -0.34471 -0.05615 0.44380 1.48952
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 2.46687 0.08760 28.161 < 2e-16 ***
lcavol 0.67645 0.12384 5.462 9.88e-07 ***
lweight 0.26528 0.09363 2.833 0.0063 **
age -0.14503 0.09757 -1.486 0.1425
lbph 0.20953 0.10128 2.069 0.0430 *
svi 0.30709 0.12190 2.519 0.0145 *
lcp -0.28722 0.15300 -1.877 0.0654 .
pgg45 0.25228 0.11562 2.182 0.0331 *
---
Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1
Residual standard error: 0.7064 on 59 degrees of freedom
Multiple R-squared: 0.6943, Adjusted R-squared: 0.658
F-statistic: 19.14 on 7 and 59 DF, p-value: 4.496e-13
> y.pred.bestsubset <- predict(m.bestsubset,data.test[,bestfeat])
> summary((y.pred.bestsubset - y.test)^2)
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.000575 0.038000 0.122400 0.516500 0.449300 4.002000
>
> mse.bestsubset=sum((y.pred.bestsubset - y.test)^2)
> mse.bestsubset
[1] 15.4954
リッジ回帰
次にリッジ回帰でやってみます。
リッジ回帰とラッソについて、なんだっけという場合は下記がご参考。
で、さくっとやってみます。
> library(MASS)
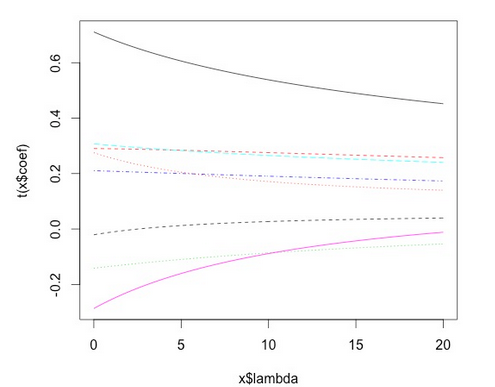
> m.ridge <- lm.ridge(lpsa ~ .,data=data.train, lambda = seq(0,20,0.1))
> plot(m.ridge)
>
> #by using the select function
> select(m.ridge)
modified HKB estimator is 3.355691
modified L-W estimator is 3.050708
smallest value of GCV at 4.9
下記のような図と共に、ベストなλは4.9だと選ばれました。
次に予測値を返させて、MSEを求めます。
> y.pred.ridge = scale(data.test[,1:8],center = m.ridge$xm, scale = m.ridge$scales)%*% m.ridge$coef[,which.min(m.ridge$GCV)] + m.ridge$ym
> summary((y.pred.ridge - y.test)^2)
V1
Min. :0.000003
1st Qu.:0.043863
Median :0.136450
Mean :0.494410
3rd Qu.:0.443495
Max. :3.809051
> mse.ridge=sum((y.pred.ridge - y.test)^2)
> mse.ridge
[1] 14.8323
ラッソ
あまり使っている見ませんが、larsライブラリー([https://cran.r-project.org/web/packages/lars/index.html])を使います。
> library(lars)
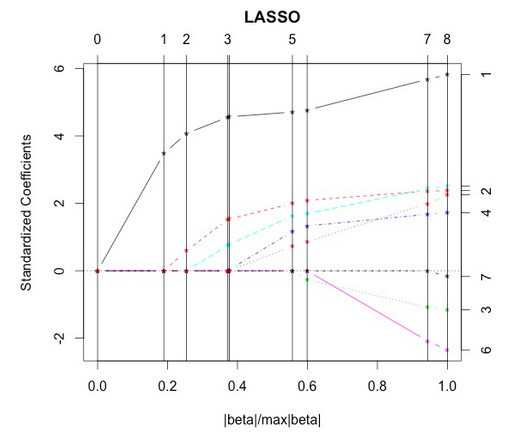
> m.lasso <- lars(as.matrix(data.train[,1:8]),data.train$lpsa,type="lasso")
> plot(m.lasso)
> # Cross-validation
> r <- cv.lars(as.matrix(data.train[,1:8]),data.train$lpsa)
> bestfraction <- r$index[which.min(r$cv)]
> bestfraction
[1] 0.5252525
fractionは0.525あたりと図と出力から判断出来ます。
> # Observe coefficients
> coef.lasso <- predict(m.lasso,as.matrix(data.test[,1:8]),s=bestfraction,type="coefficient",mode="fraction")
>
> # Prediction
> y.pred.lasso <- predict(m.lasso,as.matrix(data.test[,1:8]),s=bestfraction,type="fit",mode="fraction")$fit
> summary((y.pred.lasso - y.test)^2)
Min. 1st Qu. Median Mean 3rd Qu. Max.
0.000144 0.072470 0.128000 0.454000 0.394400 3.565000
>
> mse.lasso=sum((y.pred.lasso - y.test)^2)
> mse.lasso
[1] 13.61874
平均二乗誤差の比較
> # Compare them
> mse.ols
[1] 15.63822
> mse.bestsubset
[1] 15.4954
> mse.ridge
[1] 14.8323
> mse.lasso
[1] 13.61874
今回のデータに関しては、ラッソで行った分析が一番MSEを最小に出来ました。