本更新はR advent calendar 2015: 17日目の記事となります。
####背景・目的
昨年12月より、「続・わかりやすいパターン認識」読書会を主催させていただき、色々と不慣れな部分はありながらもなんとか次回、1月12日に最終回を迎えることが出来そうです(10回あたりからblog更新が途絶えててすいません…)。
しかしほっとするにはまだ早い。最終回の発表担当はこの本を開くまで最終章のテーマ、「共クラスタリング」という単語を聞いたことがない私です。その上、本で紹介されている無限関係モデル (Infinite Relational Model: IRM) は共クラスタリングの中でもその前の章までに勉強してきたノンパラベイズの手法を取り入れた必殺技であり、"共クラスタリング is 何" とか言ってる人がいきなり手をつけるには厳しい内容です。
そこで、まずは最尤法で動くパッケージの動作を見ながら共クラスタリングに慣れつつ、資料のイントロを先に作っておこうというのが今回の目的です。
####パッケージなど
使用するのは以下のパッケージです。
"blockcluster: Coclustering Package for Binary, Categorical, Contingency and Continuous Data-Sets"
こちらはEMアルゴリズムで共クラスタリングを行うためのパッケージです。ベースはC++で書かれているらしく、今回テストで使った12×300くらいのデータなら瞬殺です。EMアルゴリズムについてはぞくパタで@yamano357さんに発表いただいた資料が非常に充実しているので是非ご参照ください。
Vinetteはこちら。またJSSにsubmit済みの論文も著者らのサイトに上がっています。解析対象にはいつぞや作成した自分の尿中代謝物のデータを使います。
####データ読み込み・簡易な前処理
以下のコードでデータを読み込み、転置後にnaを取り除いておきます。合わせてスケーリングも行います。
metabo = read.csv("https://gist.githubusercontent.com/siero5335/c7323407efb2b0b20b55/raw/325390f7c42385357646dd861ee7f774015c4aee/metabolo_test.csv")
metabo <- metabo[,-c(1,2)]
metabo_t <- t(metabo)
metabo_t<-na.omit(metabo_t)
metabo_df <- data.matrix(metabo_t)
metabo_df <- scale(metabo_df)
####共クラスタリングの実行
下記のように実行可能です。
out<-cocluster(metabo_df,datatype="continuous",nbcocluster=c(3,2))
これだけだと何のことやらわからないのでcocluster関数の中身を見ていきます。
cocluster(data, datatype, semisupervised = FALSE,
rowlabels = numeric(0), collabels = numeric(0),
model = character(0), nbcocluster,
strategy = cocluststrategy()
dataにはデータ名、datatypeにはデータの種類("binary" , "contingency", "continuous", "categorical"から1つ指定)を指定します。rowlabels, collabelsそれぞれに各データのクラスタをc(1,2,...1; unknownは-1を入力) で与えるとともに、semisupervisedをTRUEにすることで半教師あり学習を行うことが出来ます。modelにはデータの種類や平均、分散、クラスタ間におけるサンプルサイズの偏りの有無などを見ながら適切なモデルを入力します(help: cocluster参照, 各データの種類についてデフォルトのモデルが指定されているので、記入しなくても動きます)。nbcoclusterにはrow, columnの順で、いくつのクラスタに分けたいかをc(X,Y)の形式で指定します。strategyについては後ほど詳細を示します。
####結果
結果は以下で確認可能です。
summary(out)
plot(out)
summaryは以下の様に出力されます。クラス内の平均や分散に加え、行、列における各クラスタが占める割合が出力されます。また、尤度も合わせて出力されるので、尤度を見ながらクラスタの値を最適化していくことができると思います。
******************************************************************
Model Family : Gaussian Latent block model
Model Name : pik_rhol_sigma2kl
Co-Clustering Type : Unsupervised
Model Parameters..
Class Mean:
[,1] [,2]
[1,] -0.2451047 -0.2544150
[2,] -0.1832020 -0.1823025
[3,] 1.3884466 1.4228915
Class Variance:
[,1] [,2]
[1,] 4.835054e-05 5.201995e-05
[2,] 3.505204e-03 3.596868e-03
[3,] 4.974849e+00 4.860298e+00
Row proportions: 0.3333333 0.3333333 0.3333333
Column proportions: 0.5833333 0.4166667
Pseudo-likelihood: 2.882163
******************************************************************
各行、列がどのクラスタに所属しているかは、結果内の"rowclass"および"colclass"に格納されているので、以下のように読みだす事ができます。
out@rowclass
out@colclass
Plotは以下のように示されます。クラスタの線がちょうどサンプルの境界上に引かれていないのが納得いきませんが…
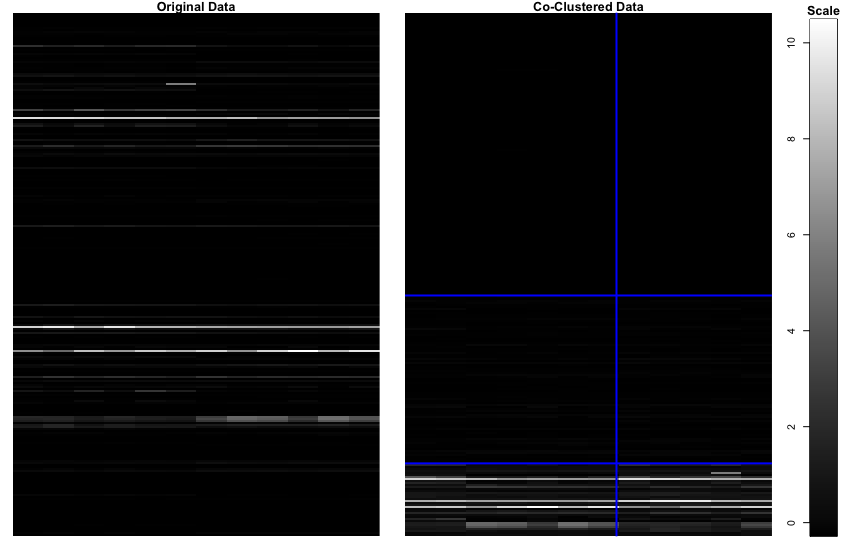
左側のオリジナルが右側の形にクラスタリングされました。元データは食前食後、各n = 6なのでそういうふうにクラスタリングして欲しかった気持ちはありますが、一致率は9/12でした。教師なし学習なのでそんなものかもしれません。
####詳細なチューニング
ここまでくればおおまかに使うことは出来そうですが、cocluststrategy関数からstrategyをいじることでより詳細にチューニングできます。
cocluststrategy(algo = "BEM", initmethod = character(),
stopcriteria = "Parameter", nbiterationsxem = 50,
nbiterationsXEM = 500, nbinititerations = 10,
initepsilon = 0.01, nbiterations_int = 5,
epsilon_int = 0.01, epsilonxem = 1e-04,
epsilonXEM = 1e-10, nbtry = 2, nbxem = 5,
bayesianform = FALSE, hyperparam = numeric(0))
algoでは、"BEM" (Default, Block EM), "BCEM"(Block Classification EM), "BSEM"(Block Stochastic EM)のうちから最適化時のアルゴリズムを選択します。デフォルトのBEMは行列それぞれにEMアルゴリズムを繰り替えし実行し、最適化していく手法のようです。
"BCEM"は complete maximum likelihood approachに基づきEMのステップに加えてC-stepなるstepを追加し、事後確率を最大化するクラスタに割りあてるとのことです。"BEM"は論文中の式(2), (3)に記述されるEM stepを経て最適化をしますが、"BCEM"ではこれに加え式(4)をstepに加える事で潜在ブロックへの尤度を最大化するとのことでした(間違ってたらご指摘ください…)。BSEMはvignetteにも論文にも情報がなかったので中身については今のところ謎です。
収束条件や繰り返しの回数はstopcriteriaやnbiterationsxem(row用), nbiterationsXEM(column用)から指定することが出来ます。また、row, column行き来の回数はnbiterations_intで指定するようです。
その他、epsilon~ではステップのトレランスを設定可能です。bayesianformは現在"binary", "categorical"モデルの対してのみ適用可能なようです。これをTRUEにする際にはhyperparamに事前分布を入れろ、a, b = 1の時はデフォルトと変わらないよとの事なので、おそらくa, bにrow, columnのクラスタ数となりうる値を入れろということだと思うのですが、この点については詳細な記述はありませんでした。
ここで作ったstrategyは、以下のように記述し、cocluster関数内で宣言することで設定を反映することが出来ます。
ST <- cocluststrategy(algo = "BEM", initmethod = character(),
stopcriteria = "Parameter", nbiterationsxem = 50,
nbiterationsXEM = 500, nbinititerations = 10,
initepsilon = 0.01, nbiterations_int = 5,
epsilon_int = 0.01, epsilonxem = 1e-04,
epsilonXEM = 1e-10, nbtry = 2, nbxem = 5,
bayesianform = FALSE, hyperparam = numeric(0))
out<-cocluster(metabo_df,datatype="continuous",nbcocluster=c(3,2), strategy =ST)
今回、共クラスタリングについてのパッケージを紹介することで、少しだけですが共クラスタリングの理解が進んだように思います。最終回に向け、予習を進めたいと思いますので最後までよろしくお願いいたします。
明日の更新は@tetsuroitoさんです!
####参考
@yamano357 『続・わかりやすいパターン認識』 第6章 EMアルゴリズム
Parmeet Singh Bhatia, Serge Iovleff, Gerard Goavert, Vincent Brault, with contributions from Christophe Biernacki and Gilles Celeux. blockcluster: Coclustering Package for Binary, Categorical, Contingency and Continuous Data-Sets
Parmeet Bhatia, Serge Iovleff, Gerard Govaert. blockcluster: An R Package for Model Based Co-Clustering. 2014.
石井 健一郎, 上田 修功. 続・わかりやすい パターン認識 -教師なし学習入門-. 2014