本記事R Advent Calendar 201619日目の記事になります。
まえがき
皆様は今でも現役でrandomforest使っていますか?
xgboost, lightGBM, mxnetなど、新星が次々に登場しており、一時期に比べるとその存在感は薄くなってしまったかなと言う印象もあります。
とは言え、パラメータチューニングが比較的容易、結果が解釈しやすいなどの利点から、現在においても十分に一線級だと考えています。
最近のrandomforest実行パッケージ
最近のRを通じたrandomforest package色々については、
RでランダムフォレストやるならRboristかrangerか
"ranger: A Fast Implementation of Random Forests"のメモ書き
最近のRのランダムフォレストパッケージ -ranger/Rborist-
新型のランダムフォレスト(Random Forest)パッケージ比較:Rborist・ranger・randomForest
などの資料があります。
既報におけるranger, Rboristの特徴
これらにあるように、最近のrandomforest業界ではranger, Rboristなど、従来のrandomforestに比べ高速なパッケージが使われ始めています。
Rborist, rangerの比較という観点から見ると、メモリ消費量ではRboristが有利, 速度では特徴量が多いならranger、少なければRborist,formulaの有無(rangerでは使用可)も報告されています。また、ranger, Rboristいずれもcaretから呼び出して使うことができるので、caret中心に色々やりたい人も安心ですね。
ライブラリ読み込み
library(QSARdata)
library(ranger)
library(Rborist)
library(caret)
実行前にtest dataをつくる。
本テストではQSARdata内に含まれる、化学物質が血液脳関門を通過するかどうかの結果(bbb2_Outcome: 80分子)と、Dragonというソフトフェアにより算出された分子記述子(bbb2_Dragon: 1456記述子)を使うことにする。
data(bbb2)
work <- merge(bbb2_Outcome, bbb2_Dragon)
work <- na.omit(work)
簡易実行例(QSARdataのデータを利用)
#ranger
res_ranger <- ranger(Class ~., work[, -1], mtry = 2,
num.trees = 500, write.forest=TRUE)
table(work$Class, predict(res_ranger,
data=work[, -1])$predictions)
Crosses DoesNot
Crosses 45 0
DoesNot 0 33
#Rborist
res_Rborist <- Rborist(work[ ,-c(1,2)],
work$Class, predProb=0.5, nTree = 500)
table(work$Class, predict(res_Rborist,
work[ ,-c(1,2)])$yPred)
1 2
Crosses 45 0
DoesNot 0 33
どっちでもきれいに分類できます。
もうちょっとranger, Rboristの特徴
さて、randomforestの特徴として、結果が解釈しやすいことを利点の1つとして挙げていました。これが何故なのかというと、変数重要度が算出できることが挙げられます。Japan.Rで私がお話させていただいたように、Gini係数やpermutation baseの変数重要度を算出することができます。Japan.Rで発表させて頂いたrangerにおけるpermutation baseの変数重要度算出法は以下の通りです。
#ranger
res_ranger <- ranger(Class ~., data = work[, -1], mtry = 2,
num.trees = 500, write.forest=TRUE, importance = "permutation")
importance_pvalues(res_ranger, method = "janitza", conf.level = 0.95)
importance pvalue
DragonX_MW -3.276587e-04 0.9515455305
DragonX_AMW -8.528555e-05 0.7518796992
DragonX_Sv 7.642089e-05 0.2681704261
DragonX_Se -3.910256e-04 0.9707602339
DragonX_Sp 8.333333e-06 0.4068504595
DragonX_Ss 3.091283e-04 0.0543024227
...(以下略)
重要なのはimportance = "permutation"と指定することです。
一方、Rboristではrandomforestから感度分析をするパッケージであるforestfloorを呼び出して解析するためのコードがhelpに載っているのですが、何故か上手く行きませんでした。
#Rboristですがだめでした
res_Rborist <- Rborist(work[ ,-c(1,2)], work$Class)
ffe <- ForestFloorExport(res_Rborist)
library(forestFloor)
forestFloor(ffe, work[ ,-c(1,2)])
forestFloor(ffe, work[, -c(1, 2)]) でエラー:
This class is not yet supported, is this a random forest model fit?
ForestFloorExportで上手く形式を変えることはできているようなので、もしかするとforestFloor側の問題なのかもしれません。forestFloorについては以前のTokyo.Rで紹介があるのでそちらを参考にしていただけると幸いです。
これらに加え、@nakamichiさんの記事にもあったedarf: Exploratory Data Analysis using Random Forestsや、inTree(xgboost対応)や、こちらの論文で話題になったdefragTrees(Rのrandomforestの結果を使うことは可能)も面白そうですね。
edarfはrangerにも対応しているので下記のように実行してやれば@nakamichiさんの記事での検証と同様の内容を追試することができると思います。
#ranger
res_ranger <- ranger(Class ~., data = work[, -1], mtry = 2,
num.trees = 500, write.forest=TRUE)
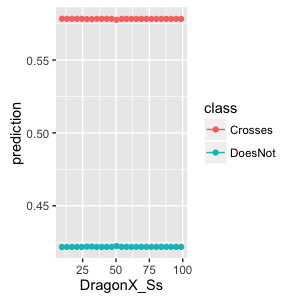
pd <- partial_dependence(res_ranger, data = work[, -1], vars = "DragonX_Ss")
plot_pd(pd)
もともと高い精度がウリだったrandomforestですが、特徴量抽出や変数重要度を用いたシステムの解釈という枠組みでも上手く利用していきたいと思っています。深層学習にもTensorBoardのように学習の中身を可視化していくような流れが出てきていますが、現状この点においてはまだrandomforestが有利かな、と思います。線形モデルだけではどうにもならないけど解釈性はある程度保ちたい。そんなときには深部に踏み込む前に、森を彷徨ってみるのも良いかもしれません。最近は道がある程度整備され、見通しも良くなっているようなので。
明日は@weda_654さんです。
参考文献
RでランダムフォレストやるならRboristかrangerか
"ranger: A Fast Implementation of Random Forests"のメモ書き
最近のRのランダムフォレストパッケージ -ranger/Rborist-
新型のランダムフォレスト(Random Forest)パッケージ比較:Rborist・ranger・randomForest
edarf パッケージ:ランダムフォレストで探索的データ分析
edarf: Exploratory Data Analysis using Random Forests
random forestを予測以外の目的で使う
inTrees: Interpret Tree Ensembles
random forestの感度分析+
forestFloor: Visualizes Random Forests with Feature Contributions
Making Tree Ensembles Interpretable: A Bayesian Model Selection Approach
defragTrees