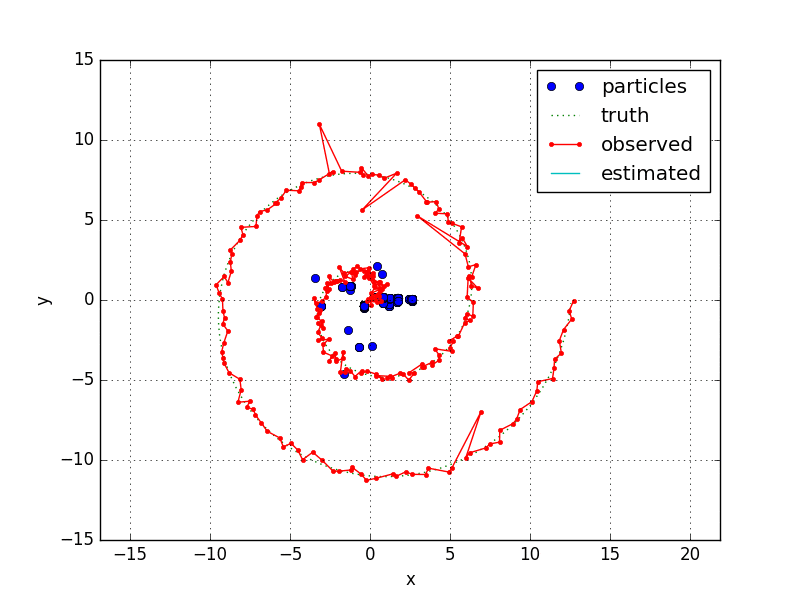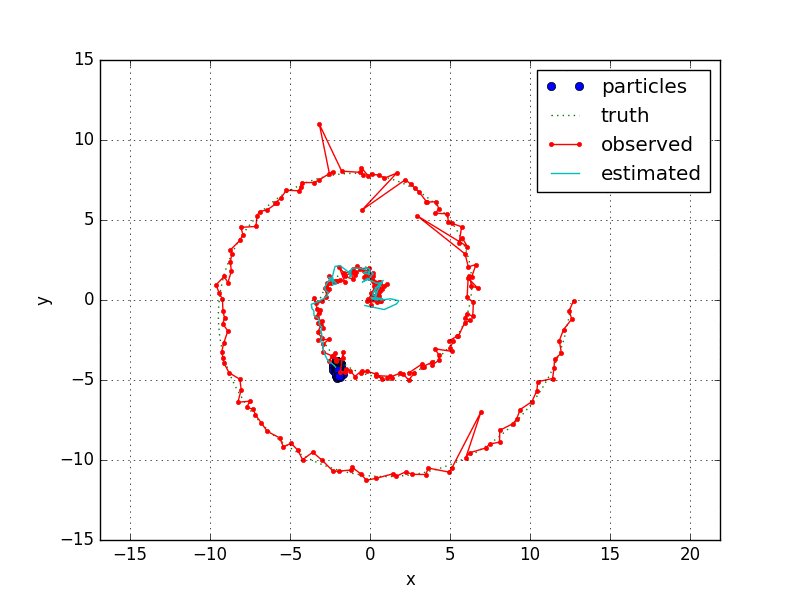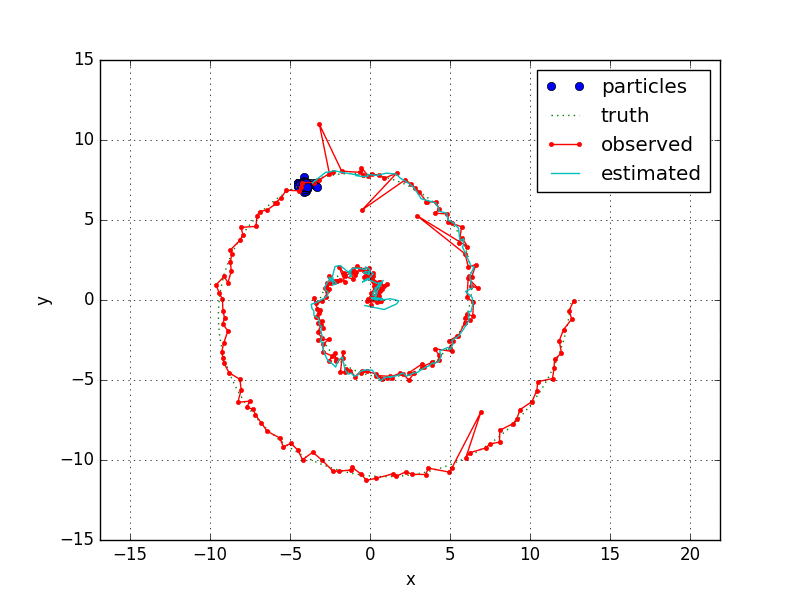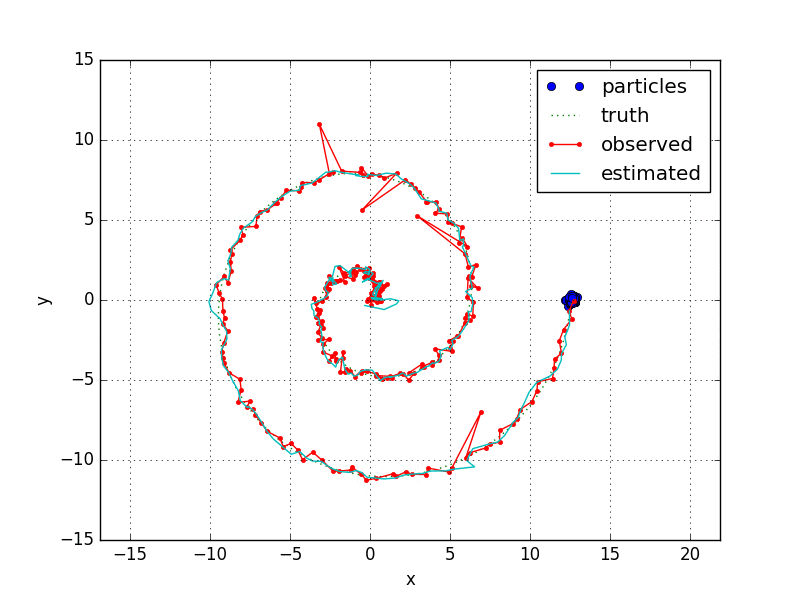
はじめに
ありがちなやつですが、作成したので置いておきます。
気が向いたら解説を追加します。
- はじめロックするまでは変なところをウロウロしてます。
- 何点かわざと外れ値を入れていますが、きちんとフィルタリングしているようです。
こここうした方がいいよとか間違ってね?とかあったら気軽にコメントください。
あと、よくわかんないから教えてとかでも歓迎します。
2016/12/14 修正
- ソースがごちゃごちゃしていたのを書き直し。割りとスッキリかけた気がする
- numpyをforで回すと遅いらしいので、リサンプリングをcythonで書き直してみた(あまり変わらず)
- cupyで動かそうとしたが、案の定うまくインストールできず無事死亡(そもそもNVIDIA製のGPUじゃなくてCUDAが動かなかったみたいです。残念)
- カルマンフィルタをおまけで追加
ソース概要
粒子フィルタ
- particle.py
- test_particle.py
- resample.pyx
- utils.py
カルマンフィルタ(おまけ)
- kalman.py
- test_kalman.py
ソースコード
粒子フィルタ本体
- 重み更新時の尤度計算は、対数尤度の和をとってからexpに戻してます。これは、指数の積とったときにオーバーフローが起きるのを防ぐため。
- リサンプリングは、まずインデックスのリスト
idxsを生成して、pars[:, idxs]とかで一気に撒き直してます。
particle.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
"""粒子フィルタのpython実装
"""
import pyximport
pyximport.install()
import resample
import numpy as xp
# import cupy as xp
class GaussianNoiseModel(object):
"""多次元ガウス分布
"""
def __init__(self, Cov):
"""コンストラクタ
@param ndarray(n,n) Cov : 分散共分散行列
"""
self._Cov = Cov
def generate(self, num):
"""ノイズの生成
@param int num : 粒子数
@return ndarray(n,num) : 雑音行列
"""
n, _ = self._Cov.shape
return xp.random.multivariate_normal(xp.zeros(n), self._Cov, num).T
def logpdf(self, X):
"""対数確率密度関数
@param ndarray(n,num) X
@param int num : 粒子数
@return ndarray(num) : 対数確率密度
"""
Cov = self._Cov
k, _ = Cov.shape
det_Cov = xp.linalg.det(Cov)
Cov_inv = xp.linalg.inv(Cov)
coefs = xp.array([ x.dot(Cov_inv).dot(x.T) for x in X.T ])
return -k*xp.log(2.*xp.pi)/2. -xp.log(det_Cov)/2. -coefs/2.
class CauchyNoiseModel(object):
"""多次元独立コーシー分布
各変数に独立を仮定した多次元コーシー分布
"""
def __init__(self, gma):
"""コンストラクタ
@param ndarray(n) gma : 尺度母数
"""
self._gma = gma
def generate(self, num):
gma = self._gma
uni = xp.random.rand(gma.size, num)
Gma = gma.reshape(gma.size,1) * xp.ones(num)
return xp.arctan(uni / Gma) /xp.pi + 1./2.
def logpdf(self, X):
_, num = X.shape
gma = self._gma
Gma = gma.reshape(gma.size,1) * xp.ones(num)
return xp.sum(xp.log(Gma/xp.pi) - xp.log(X**2 + Gma**2), axis=0)
def _normalize(w):
"""重みの正規化
@param ndarray(num) w : 各粒子の重み
@return ndarray(num) : 各粒子の正規化された重み
"""
return w / xp.sum(w)
class ParticleFilter(object):
"""Particle Filter (粒子フィルタ)
"""
def __init__(self, f, g, h, t_noise, o_noise, pars_init):
"""コンストラクタ
@param ndarray(nx,num) function( ndarray(nx,num) ) f : 状態遷移関数
@param ndarray(nx,num) function( ndarray(nu,num) ) g : 入力伝搬関数
@param ndarray(ny,num) function( ndarray(nx,num) ) h : 観測関数
@param NoiseModel t_noise : システムノイズモデル
@param NoiseModel o_noise : 観測ノイズモデル
@param ndarray(nx,num) pars_init : 粒子の初期値
"""
self._f = f
self._g = g
self._h = h
self._t_noise = t_noise
self._o_noise = o_noise
_, num = pars_init.shape
self._num = num
self._w = _normalize(xp.ones(num))
self._pars = pars_init
def update(self, y, u):
"""フィルタ更新
@param ndarray(ny) y : nでの観測ベクトル
@param ndarray(nu) u : nでの入力ベクトル
"""
self._update_pars(u)
self._update_weights(y)
self._resample()
def _update_pars(self, u):
"""状態遷移モデルに沿って粒子を更新
- 状態方程式
x_n = f(x_n-1) + g(u_n-1) + w
- 状態ベクトル x_n
- 入力ベクトル u_n
- 状態遷移関数 f(x)
- 入力伝搬関数 g(u)
- システムノイズ w
@param ndarray(nu) u : n-1での入力ベクトル (u_n-1)
"""
U = u.reshape(u.size,1) * xp.ones(self._num)
self._pars = self._f(self._pars) + self._g(U) + self._t_noise.generate(self._num)
def _update_weights(self, y):
"""観測モデルに沿って尤度を計算
- 観測方程式
y_n = h(x_n) + v
- 状態ベクトル x_n
- 観測ベクトル y_n
- 観測関数 h(x)
- 観測ノイズ v
@param ndarray(ny) y : nでの観測ベクトル (y_n)
"""
Y = y.reshape(y.size,1) * xp.ones(self._num)
loglh = self._o_noise.logpdf( xp.absolute(Y - self._h(self._pars)) )
self._w = _normalize( xp.exp( xp.log(self._w) + loglh ) )
def _resample(self):
"""リサンプリング
"""
wcum = xp.cumsum(self._w)
num = self._num
# idxs = [ i for n in xp.sort(xp.random.rand(num))
# for i in range(num) if n <= wcum[i] ]
# start = 0
# idxs = [ 0 for i in xrange(num) ]
# for i, n in enumerate( xp.sort(xp.random.rand(num)) ):
# for j in range(start, num):
# if n <= wcum[j]:
# idxs[i] = start = j
# break
idxs = resample.resample(num, wcum)
self._pars = self._pars[:,idxs]
self._w = _normalize(self._w[idxs])
def estimate(self):
"""状態推定
@return ndarray(nx) : 状態ベクトルの推定値
"""
return xp.sum(self._pars * self._w, axis=1)
def particles(self):
"""粒子
@return ndarray(nx,num)
"""
return self._pars
リサンプリングはCythonで
ここはちょっと工夫しました。以下のようにすれば、2重のループになっていますが、ほぼ1重ループ分の処理で済みます。ループ回数が粒子数の2乗でなく1乗に比例するので、粒子数を大きくしていったときに速度に大きく差が出ます。
- $[0, 1]$の一様乱数$n_i$を生成して昇順にソートしておく
- 乱数$n_i$を取る
- 累積重み$w_j$を取る
- 乱数$n_i$が取った累積重み$w_j$より大きい場合は、次の重み$w_{j+1}$に移る
- 小さい場合は、そのインデックス$j$を採用 -> 次の乱数$n_{i+1}$に移る
- ことのき、次の乱数$n_{i+1}$は、$w_{j-1}<n_i<n_{i+1}$なので、$w_j$との比較から始めれば良い
- 累積重み$w_j$を取る
resample.pyx
# -*- coding: utf-8 -*-
# cython: boundscheck=False
# cython: wraparound=False
import numpy as xp
cimport numpy as xp
cimport cython
ctypedef xp.float64_t DOUBLE_t
ctypedef xp.int_t INT_t
def resample(int num, xp.ndarray[DOUBLE_t, ndim=1] wcum):
cdef int start, i, j, length
cdef double n
start = 0
cdef xp.ndarray[INT_t, ndim=1] idxs = xp.zeros(num).astype(xp.int)
cdef xp.ndarray[DOUBLE_t, ndim=1] rand = xp.sort(xp.random.rand(num))
length = rand.size
for i in xrange(length):
for j in xrange(start, num):
if rand[i] <= wcum[j]:
idxs[i] = start = j
break
return idxs
resample_setup.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
# filename_setup.py
from distutils.core import setup
from distutils.extension import Extension
from Cython.Distutils import build_ext as build_pyx
import numpy
setup(
name = 'resample',
ext_modules = [Extension('resample', ['resample.pyx'])],
cmdclass = { 'build_ext': build_pyx },
include_dirs = [numpy.get_include()],
)
$ cython -a resample.pyx # コンパイル
$ python resample_setup.py build_ext --inplace # ビルド
Cythonよくわかってないせいか、あんまり早くなりませんでした。
(詳しい方コメントいただけるとありがたいです)
利用プログラムサンプル
システムノイズ、観測ノイズを多次元の独立コーシー分布とした場合。はずれ値耐性がつく。
参考:自己組織化型状態空間モデルを用いた運動軌跡のフィルタリング
py.test_particle.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
"""particle.pyの利用サンプル
"""
import particle as par
import utils
import numpy as xp
# import cupy as xp
# パラメータ設定 --------------------
num = 3000 # 粒子数
v_sys = 0.01 # システムノイズの共分散
v_obs = 0.1 # 観測ノイズの共分散
v_noise = 0.05
# 初期粒子
mins = -5. * xp.ones(4)
maxs = +5. * xp.ones(4)
pars_init = utils.rand_uniform(mins, maxs, num)
# ---------------------------------
dataset = utils.load_data("testdata")
# dataset = utils.generate_data("testdata")
# 状態モデルの生成 (2次階差モデル)
A = xp.kron(xp.eye(2), xp.array([[2, -1], [1, 0]]))
f = lambda X: A.dot(X) # 状態遷移関数の定義
B = xp.zeros(1)
g = lambda U: B.dot(U)
C = xp.ones(4) * v_sys
sysn_model = par.CauchyNoiseModel(C)
# 観測モデルの生成 (直接観測モデル)
D = xp.kron(xp.eye(2), xp.array([1, 0]))
h = lambda X: D.dot(X) # 観測関数の定義
E = xp.ones(2) * v_obs
obsn_model = par.CauchyNoiseModel(E)
# 初期プロット
lines = utils.init_plot_particle(dataset, pars_init)
# 粒子フィルタの生成
pf = par.ParticleFilter(
f, g, h, sysn_model, obsn_model, pars_init)
x_est = xp.zeros(dataset.x.shape)
for i, y in enumerate(dataset.y):
pf.update(y, xp.zeros(1))
state = pf.estimate()
x_est[i,:] = h(state)
# データのプロット
pars = pf.particles()
utils.plot_each_particle(lines, x_est, pars, i)
# utils.save_as_gif_particle(dataset, pars_init, pf, "particle_selforganized.gif")
プロットとかutility系
utiils.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
import matplotlib.animation as animation
import matplotlib.pyplot as plt
from collections import namedtuple
import numpy as xp
# import cupy as xp
Dataset = namedtuple("Dataset", ["t", "x", "y"])
def load_data(dirname):
t = xp.loadtxt(dirname + "/t.txt")
x = xp.loadtxt(dirname + "/x.txt")
y = xp.loadtxt(dirname + "/y.txt")
dataset = Dataset(t=t, x=x, y=y)
return dataset
def save_data(dirname, dataset):
xp.savetxt(dirname + "/t.txt", dataset.t)
xp.savetxt(dirname + "/x.txt", dataset.x)
xp.savetxt(dirname + "/y.txt", dataset.y)
def generate_data(dirname):
# truth
t = xp.arange(0, 6*xp.pi, 0.05)
x = xp.c_[t*xp.cos(t), t*xp.sin(t)]
# noise
nr, nc = x.shape
idx = xp.random.randint(nr/2, nr, 5)
n = xp.random.normal(0., xp.sqrt(v_noise), x.shape)
n[nr/3:2*nr/3,:] = xp.random.normal(0., xp.sqrt(v_noise*10), (2*nr/3-nr/3,nc))
n[idx,:] += xp.random.normal(0., xp.sqrt(v_noise)*20, (5,nc))
# observation
y = x + n
x_est = xp.zeros(x.shape)
Dataset = namedtuple("Dataset", ["t", "x", "y"])
dataset = Dataset(t=t, x=x, y=y)
save_data(dirname, dataset)
return dataset
def init_plot_particle(dataset, pars):
fig, ax = plt.subplots(1, 1)
ax.hold(True)
ax.axis("equal")
lines = []
lines.append( ax.plot(pars[0,:], pars[2,:], "o") )
lines.append( ax.plot(dataset.x.T[0], dataset.x.T[1], ":"))
lines.append( ax.plot(dataset.y.T[0], dataset.y.T[1], ".-"))
lines.append( ax.plot([0], [0]) )
plt.legend(["particles", "truth", "observed", "estimated"])
plt.xlabel("x")
plt.ylabel("y")
plt.grid(True)
return lines
def plot_each_particle(lines, x_est, pars, i):
lines[0][0].set_data(pars[0,:], pars[2,:])
lines[3][0].set_data(x_est.T[0][:i], x_est.T[1][:i])
plt.pause(1e-5)
def save_as_gif_particle(dataset, pars_init, pf, filename):
x_est = xp.zeros(dataset.x.shape)
lines = init_plot_particle(dataset, pars_init, x_est)
def draw(i):
pf.update(dataset.y[i])
state = pf.estimate()
x_est[i,:] = [state[0], state[2]]
# データのプロット
pars = pf.particles()
plot_each_particle(lines, x_est, pars, i)
ny, _ = dataset.y.shape
ani = animation.FuncAnimation(fig, draw, ny, blit=False, interval=100, repeat=False)
ani.save(filename, writer="imagemagick")
def init_plot_kalman(dataset):
fig, ax = plt.subplots(1, 1)
ax.hold(True)
ax.axis("equal")
lines = []
lines.append( ax.plot(dataset.x.T[0], dataset.x.T[1], ":"))
lines.append( ax.plot(dataset.y.T[0], dataset.y.T[1], ".-"))
lines.append( ax.plot([0], [0]) )
plt.legend(["truth", "observed", "estimated"])
plt.xlabel("x")
plt.ylabel("y")
plt.grid(True)
return lines
def plot_each_kalman(lines, x_est, i):
lines[2][0].set_data(x_est.T[0][:i], x_est.T[1][:i])
plt.pause(1e-5)
def save_as_gif_kalman(dataset, kf, filename):
x_est = xp.zeros(dataset.x.shape)
lines = init_plot_kalman(dataset, x_est)
def draw(i):
kf.update(dataset.y[i])
state = pf.estimate()
x_est[i,:] = [state[0], state[2]]
# データのプロット
plot_each_kalman(lines, x_est, i)
ny, _ = dataset.y.shape
ani = animation.FuncAnimation(fig, draw, ny, blit=False, interval=100, repeat=False)
ani.save(filename, writer="imagemagick")
def rand_uniform(mins, maxs, num):
"""初期粒子の生成用
@param ndarray(nx) mins : 粒子の各最小値
@param ndarray(nx) maxs : 粒子の各最大値
@param int num : 粒子数
@return ndarray(nx,num) : 初期粒子
"""
nx = mins.size
return (mins.reshape(nx, 1) * xp.ones(num)
+ (maxs - mins).reshape(nx, 1) * xp.random.rand(nx, num))
おまけ
カルマンフィルタ本体
kalman.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
"""カルマンフィルタのpython実装
"""
import numpy as xp
# import cupy as xp
class KalmanFilter(object):
def __init__(self, F, H, Q, R, x_init, P_init):
self._x = x_init.reshape(-1,1)
self._P = P_init
self._update_x = lambda x : F.dot(x)
self._update_P = lambda P : Q + F.dot(P).dot(F.T)
self._error = lambda x,y : y - H.dot(x)
self._cov_P = lambda P : R + H.dot(P).dot(H.T)
self._kalman_gain = lambda P,S : P.dot(H.T).dot(xp.linalg.inv(S))
self._estimate_x = lambda x,K,e : x + K.dot(e)
self._estimate_P = lambda P,K : (xp.eye(*P.shape)-K.dot(H)).dot(P)
def update(self, y):
# 予測
x_predicted = self._update_x(self._x)
P_predicted = self._update_P(self._P)
# パラメータ計算
e = self._error(x_predicted, y.reshape(-1,1))
S = self._cov_P(P_predicted)
K = self._kalman_gain(P_predicted, S)
# 更新
self._x = self._estimate_x(x_predicted, K, e)
self._P = self._estimate_P(P_predicted, K)
def estimate(self):
return self._x
利用プログラムサンプル
test_kalman.py
# !/usr/bin/env python
# -*- coding: utf-8 -*-
"""kalman.pyの利用サンプル
"""
import kalman
import utils
import numpy as xp
# import cupy as xp
# パラメータ設定 --------------------
var_Q = 0.01
var_R = 1
x_init = xp.zeros(4)
P_init = xp.ones(4)
# ---------------------------------
dataset = utils.load_data("testdata")
# dataset = utils.generate_data("testdata")
# 状態モデルの生成 (2次階差モデル)
F = xp.kron(xp.eye(2), xp.array([[2, -1], [1, 0]]))
Q = var_Q * xp.eye(4)
# 観測モデルの生成 (直接観測モデル)
H = xp.kron(xp.eye(2), xp.array([1, 0]))
R = var_R * xp.eye(2)
# 初期プロット
lines = utils.init_plot_kalman(dataset)
kf = kalman.KalmanFilter(F, H, Q, R, x_init, P_init)
x_est = xp.zeros(dataset.x.shape)
for i, y in enumerate(dataset.y):
kf.update(y)
state = kf.estimate()
x_est[i,:] = H.dot(state.reshape(-1,1)).T
# データのプロット
utils.plot_each_kalman(lines, x_est, i)
# utils.save_as_gif_kalman(dataset, kf, "kalman.gif")