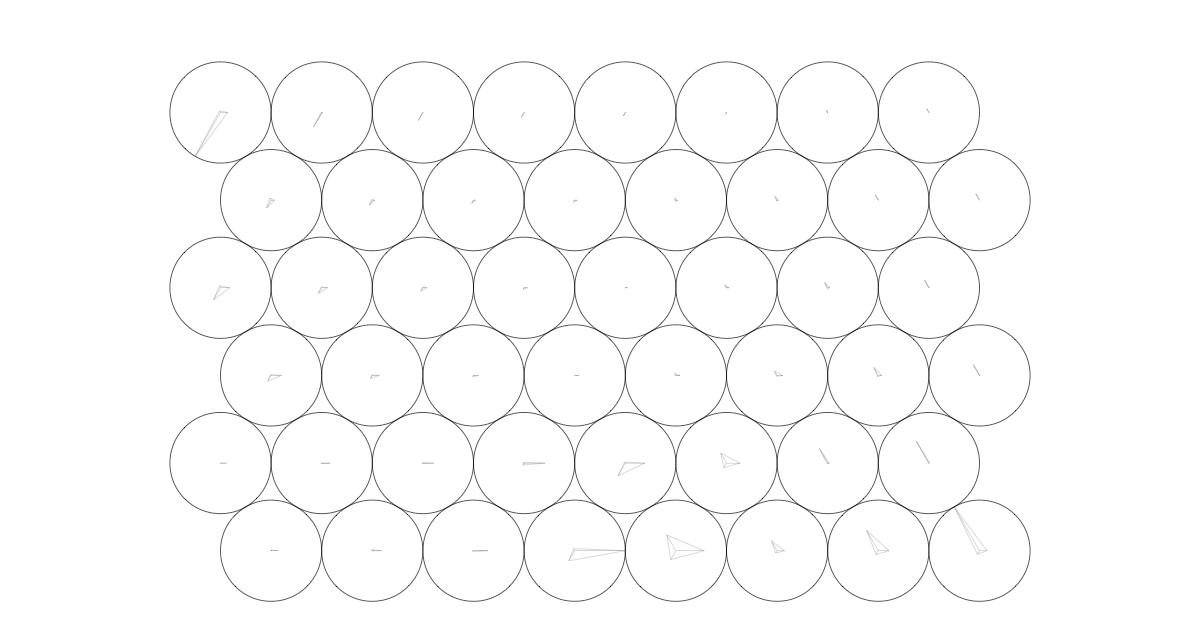
#あらすじ
最近、Rで自己組織化マップ(SOM)をやろうと思って調べてみました。
本投稿ではSOMの解説、SOMの生成からデータの見方まで扱っていきたいと思います。
#SOMって?
自己組織化特徴マップ(SOM)は、ヘルシンキ大学のコホーネン教授により1981年頃に発表された。教師なし学習を行なうニューラルネットワークの代表例と言える。
様々な入力データを、それらの類似度に応じて自動的に分類するクラスタリング能力に優れる。
何種類かのデータを適当に繰返し入力し続けると、いつの間にか、似たようなデータを近くに配置していく。
引用元:自己組織化特徴マップ(SOM) - http://www.sist.ac.jp/~kanakubo/research/neuro/selforganizingmap.html
という解説がされていますが、要は、
データ(特徴ベクトル)の集合をプロットする一つの方法ということです。
この一つ一つの円を「ノード」といい、その中にある図形はレーダーチャートだと思ってください。
このノード一つ一つがそれぞれ値を持っています。
このレーダーチャートは三方向に伸びているので、三次元の値であることを示しています。(4方向に伸びていれば4次元です)
もちろん、レーダーチャートですので、伸び方が大きいほど値は大きいです。
SOMの生成アルゴリズムについては割愛します。他サイトの解説を覗いてみてください。
#SOMを用いる利点
####ビッグデータの可視化
ビッグデータでは途方も無いくらいの時間とメモリを消費する多次元尺度構成法(mdscale)を、データを圧縮することにより適用が可能になる。
3万行くらいのデータになるともうmdscaleなんて使えなくなるのだが、SOMを生成することにより3万行→100~400行とかなりのデータの圧縮が可能になる。
要はmdscaleがビッグデータに対して適用可能になる。(ここ大事)
####高次元データの可視化
mdscale使えば良いといえばそうなんだけど、可視化の一つの選択肢。高次元&行数が多いとかになるとmdscaleでは手がつけられないので。
あとは、クラスタリングに活用できるらしい。
#RでバッチSOMを試してみる
まず、行列かデータフレームで使うデータを用意しておき、その後
library('class');
としてclassパッケージをインポートします。無ければインストールしてください。
準備が終わったら、
gr <- somgrid(xdim = 8, ydim = 6,topo = "hexagonal")
somdata <- batchSOM(useData, gr, c(4, 4, 2, 2, 1, 1, 1, 0, 0))
と入力すると、somが生成されます。ここで、batchSOMに指定したパラメータuseDataは解析したいデータセットの変数名を指定してください。
ここで、somgridのxdimは横方向の長さ、ydimは縦方向の長さを指定しています。用途に応じて適切に設定してください。
c(4, 4, 2, 2, 1, 1, 1, 0, 0) は学習半径ということらしいです。一行のデータをSOMに暴露した時、暴露させる点からどのくらい離れた点まで学習の影響を受けるのかといったところですかね。
あとは、
plot(somdata)
symbols(somdata$grid$pts[,1:2],circles=c(rep(0.5, 6*8)),inches = FALSE,add = TRUE)
でプロットが可能です。
また、元データのどのデータが、SOM上のどの点に対応しているのかを知るためには、
bins <- as.numeric(knn1(somdata$codes, useData, 0:47))
を実行してください。0:47はSOMのデータ点が全部で8*6の48点あることから来ています。もし100点のSOMノードを用意したなら、0:99とすればいいかと思いますね。
ちなみに、各ノードの値はsomdata$codesに格納されています。
しかし、この値の見方には少し癖があって、
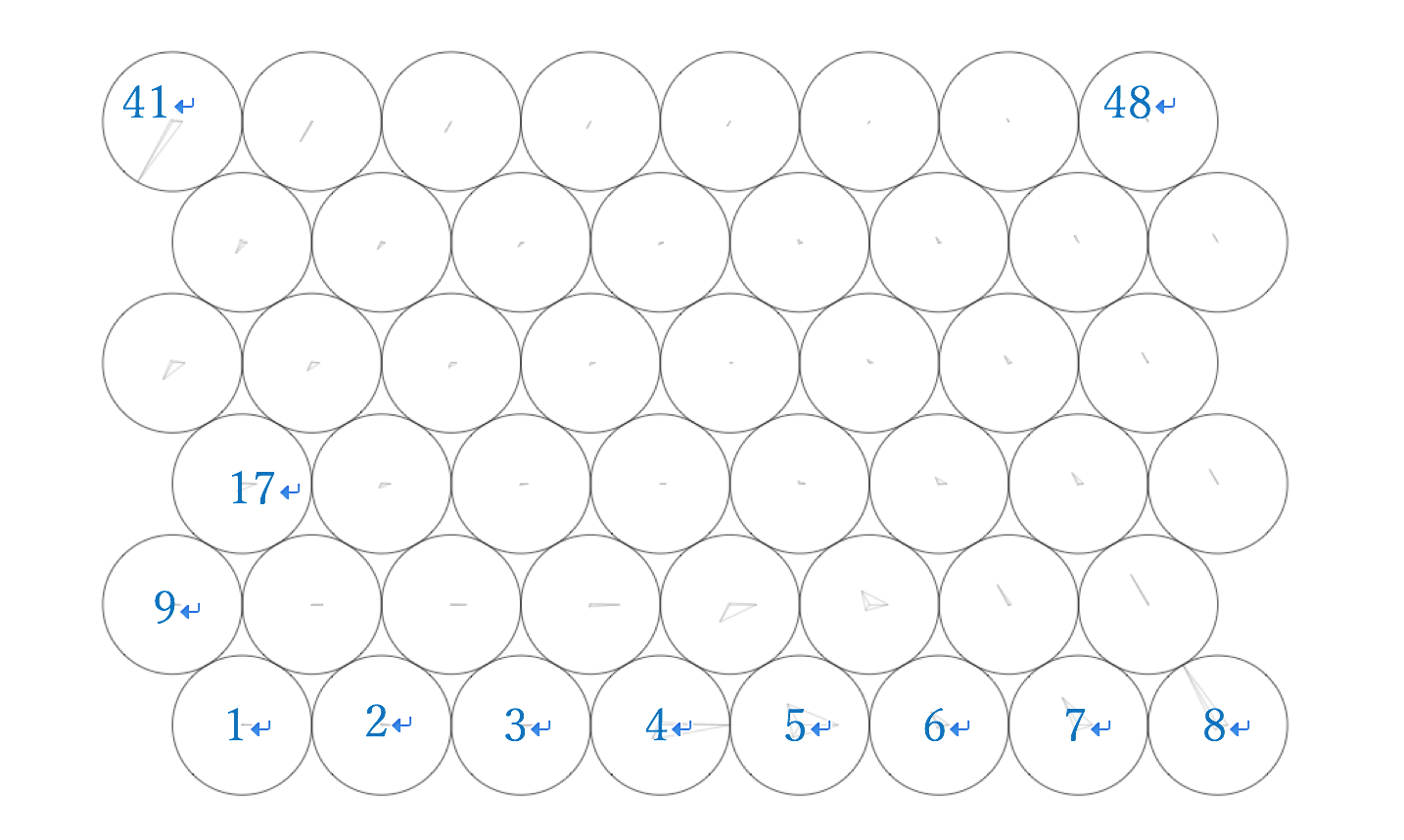
といったように、codesの一行目は左上じゃなくて左下の1と書かれたノード、codesの2行目はノードの2、そこから右に進んでいき、最終行が右上に対応している、といった仕様になっています。
もちろん、codesの値をcmdscaleに用いることも可能です。
最後に、全体をまとめたソースコードを貼っておきます。
library('class');
gr <- somgrid(xdim = 8, ydim = 6,topo = "hexagonal")
somdata <- batchSOM(useData, gr, c(4, 4, 2, 2, 1, 1, 1, 0, 0))
plot(somdata)
symbols(somdata$grid$pts[,1:2],circles=c(rep(0.5, 6*8)),inches = FALSE,add = TRUE)
bins <- as.numeric(knn1(somdata$codes, useData, 0:47))
#参考
自己組織化特徴マップ(SOM)
http://www.sist.ac.jp/~kanakubo/research/neuro/selforganizingmap.html
子供でもわかる「自己組織化マップ」
http://gaya.jp/spiking_neuron/som.htm
Rと自己組織化マップ
https://www1.doshisha.ac.jp/~mjin/R/30/30.html
batchSOM {class} | inside-R | A Community Site for R
http://www.inside-r.org/r-doc/class/batchSOM
somgrid {class} | inside-R | A Community Site for R
http://www.inside-r.org/r-doc/class/somgrid
RでSOM(自己組織化マップ) - RjpWiki
http://www.okadajp.org/RWiki/?R%E3%81%A7SOM(%E8%87%AA%E5%B7%B1%E7%B5%84%E7%B9%94%E5%8C%96%E3%83%9E%E3%83%83%E3%83%97)
Self Organizing Map(SOM)の可視化 - iAnalysis ~おとうさんの解析日記~
http://d.hatena.ne.jp/isseing333/20110224/1298512568