前振り
先に、{biOps}によるフィルタ操作を書きました。
前回、{EBImage}と{biOps}を使った画像生成を比較しました。
[0, 255]しか扱えないimagedataクラス(biOps)と、
数値なんでも扱えるimageクラス(EBImage)に注意ですね。
計算処理には{EBImage}が向いていますが、その代わり出来合いの関数が少ない。
なので道具を自分で作らないといけない事になる。
今回はその例としてCanny Edge detector を作ります。
罠
ただし、ですね。
{EBImage}は方向が変なんですよ。いや、変じゃないんですが…。
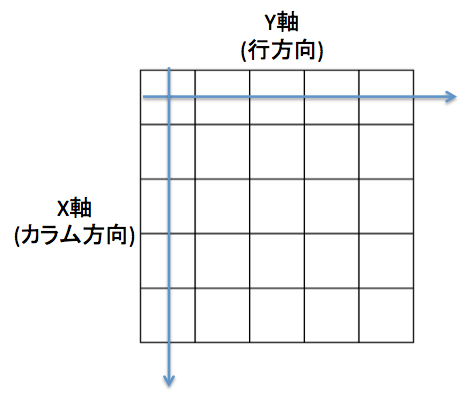
こうなっているんです。
これでフィルタ処理とかすると、X軸・Y軸、プラスマイナスがカオスになって、
大混乱のうえ悶え苦しむので、絵を保存するときは-90度回転させます。
angleはdgreeで入れるのが何ともむず痒い。
rotate(image, angle = -90)
サンプル画像
これは前回のモノを使います。
3x3に拡大内挿(biliner法)したものを使います。
すでに画像になっているものを読み込むのはreadImage。
これでjpeg, tiff, png, bmpなど、代表的な形式は全て読めました。
library("EBImage")
dat <- readImage("hoge.tiff")

Cannyの原理
Canny Edge Detection -アルゴリズム-
こちらのサイトが良くまとまっておりました。
Canny Edge Detectionのアルゴリズムは次の手順。
STEP1:ガウシアンフィルタで平滑化
STEP2:エッジ強度と勾配方向(4方向に量子化)を計算
STEP3:細線化処理(Non-maximum Suppression)
STEP4:ヒステリシス閾処理(Hysteresis Threshold)
これにのっとって進めましょう。
1. ガウシアンフィルタ
以前にも紹介しましたが、
ガウシアンフィルタの特徴
ここのサイトが素晴らしくまとまっています。
要点は、ノイズは高周波成分なので、それをカットしようという発想。
画像に自分で設計したフィルタをかける際、
{biOps}では、imgConvolve(imagedata, filter)を使いました。
{EBImage}では、filter2(image, filter)とします。挙動は同じです。
以前と同様に設計からしてもいいのですが、{EBImage}では、ガウシアンフィルタ用の関数gblurが用意されています。
dat.gau <- gblur(dat, sigma=2)
2-1. エッジ強度
エッジ強度の算出には、sobelフィルタを使います。
X, Y軸方向差分と似たような感じですね。

sobel_x <- matrix(c(1,2,1, 0,0,0, -1,-2,-1), 3)
sobel_y <- matrix(c(-1,-2,-1, 0,0,0, 1,2,1), 3, byrow=T)
え〜と、正負の組み合わせが後の計算に影響するポイントです。
この組か、x, yどちらも正負逆転した組ならOKでしょう。
フィルタをかけます。
dat.sobelx <- filter2(dat.gau, sobel_x)
dat.sobely <- filter2(dat.gau, sobel_y)
で、ここが{EBImage}を使う利点ですが、これには負の値も含まれています。
range(dat.sobelx)は、-0.86 0.86とかになります。
そうすると、こーゆー事ができます。
dat.edgeAbs <- sqrt(dat.sobelx^2 + dat.sobely^2)

これを{biOps}でやろうとすると、差分画像が[0, 255]になっちゃうので、
2乗すると意味不明になります。しかもこんな気軽に四則演算は出来ません。
2-2. エッジ勾配方向
勾配方向は、tangentで求めます。
dat.edgeTan <- dat.sobely / dat.sobelx
で一瞬。
これは、エッジの向きに対して垂直になるはずですね?
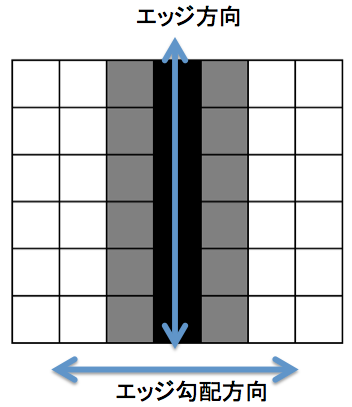
これを水平・鉛直・45度・-45度の4段階に離散化します。
格好良く書けるかもしれませんが、まぁ、ゴリ押しで十分でしょう。
A <- tan(pi/8)
B <- tan(pi*3/8)
dat.edgeTan2 <- as.Image(matrix(0, nrow(dat.edgeTan), ncol(dat.edgeTan)))
dat.edgeTan2[dat.edgeTan > -A & dat.edgeTan <= A ] <- 0 # 水平
dat.edgeTan2[dat.edgeTan > A & dat.edgeTan < B ] <- 0.3 # 45度
dat.edgeTan2[dat.edgeTan > -B & dat.edgeTan <= -A ] <- 0.6 # -45度
dat.edgeTan2[abs(dat.edgeTan) >= B ] <- 1 # 鉛直

エッジの勾配(垂線方向)が、指定の4色で塗り分けられています。
3. 細線化処理(Non-maximum Suppression)
ガウシアンフィルタでぼやけているエッジを細くします。
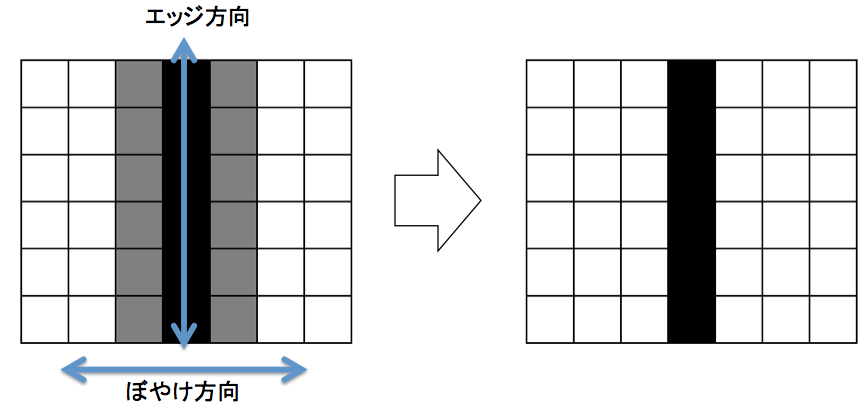
となっているわけですので、エッジに対して垂直方向の隣接ピクセルを拾ってきて、
その中でmaxになっていればエッジ、そうでないものを捨てます。
どうやって隣接ピクセルを拾いますか?
これも、恐らく格好良く書けるんでしょうけれど…。
原始的な発想では、
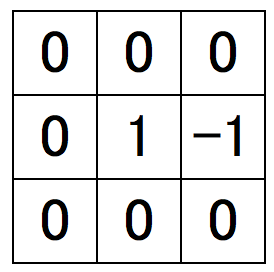
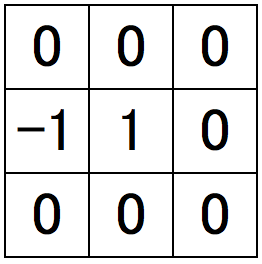
この2つのフィルタを被せて、両方で正の値を取っていれば、隣接水平方向でmaxですね。
それでも、少しだけ格好良くやりたい。
# 水平フィルタ
fil <- matrix(0, 3,3)
fil[2,2] <- 1
fil_1.1 <- fil; fil_1.1[2,3] <- -1
fil_1.2 <- fil; fil_1.2[2,1] <- -1
# 2.1で作ったエッジ強度に、水平フィルタをかける
dat1.1 <- filter2(dat.edgeAbs, fil_1.1)
dat1.2 <- filter2(dat.edgeAbs, fil_1.2)
# 水平隣接中で最大値のpixelを1、他を0にしたマスクを作る
mask1 <- as.Image(matrix(0, nrow(dat1.1), ncol(dat1.1)))
mask1[dat1.1 > 0 & dat1.2>0] <- 1
# 2.2で作った離散化エッジ勾配が 水平 == TRUE/FALSE
a <- dat.edgeTan2 == 0
# aの位置にだけmask1を適用する。
dat.edgeAbs2 <- dat.edgeAbs
dat.edgeAbs2[a] <- dat.edgeAbs2[a] * mask1[a]
 という事で、水平方向の細線化ができました。
という事で、水平方向の細線化ができました。
これを4方向(水平・鉛直・45度・-45度)にやります。
もちろん、格好良く出来るんでしょう。
しかし、私はそろそろ自伝を出そうと思っているぐらいのコピペエンジニアなので、
今回も嵐のようなコピペで乗り切ります(従ってコードは割愛)。

4. ヒステリシス閾処理(Hysteresis Threshold)
個人的には、こんなに根性いれて2値化しなくていいんですけどねぇ。。
まぁ、しょうがない。この処理も組んでみましょう。
えーと、まず、2つの閾値でハイパス・ローパスフィルタを掛けます。
この閾値は天から降ってきた値です。
high <- 0.35
low <- 0.25
dat2 <- dat.edgeAbs2
dat2[dat2 >= high] <- 100
dat2[dat2 <= low] <- 0


こうなっているので、エッジに接続していればエッジにする。
では、接続ってどうすんだ。
いや要するに、

こうなっていたら、ドウするんですかね。
この黒はワタッているのか、キレているのか。囲碁みたいですね。
囲碁だと一見トビも繋がっているかもしれませんが、ここでは除きます。
まぁ、この場合は繋がっているとしましょう。
すると、周囲8マスにエッジがあればエッジとします。
先ほど、わざわざ
dat2[dat2 >= high] <- 100
としたのはこのためで、周囲8マスを合計するフィルタをかけて、100を超えていれば接続とみなしましょう。
これが1だと、エッジがなくても閾値を超えてしまう可能性がある。
フィルタは、真ん中だけ0で、他を1にすれば合計になります。
というか、真ん中1でもいいですよね、誤差みたいなもんです。
fil_hist <- matrix(rep(1, 9), 3, 3)
repeat{
mask_hist <- filter2(dat2, fil_hist)
mask_hist[mask_hist < 100] <- 0
mask_hist[mask_hist >= 100] <- 100
dat3 <- dat2
dat3[mask_hist !=0 & dat2 != 0] <- 100
if(!all(dat2 == dat3)) dat2 <- dat3
else break
}
mask_histは、「エッジもしくはエッジに8方のいずれかで隣接する点」。
dat3は、maskが0で無く、dat2の0で無い点を100にしたもの。
それが元のdat2に完全に一致するまでrepeatをかける。

2値化しておしまい。
dat_canny <- dat2
dat_canny[dat2 < 100] <- 0
dat_canny[dat2 >= 100] <- 1

まとめ
空から降ってきた値は、
・ガウス平滑化のパラメータ: σ
・閾値2種類: high, low
・接続の定義: 8方向で接続とみなす。
この辺りは、実際のデータと付き合わせて調整ではないですかね〜。
ただ、最後までやるならbiOps::imgCannyでおしまいで良いと思います。
私の場合、手で組んだのは、2値化する前の段階で止めたかったからです。




