はじめに

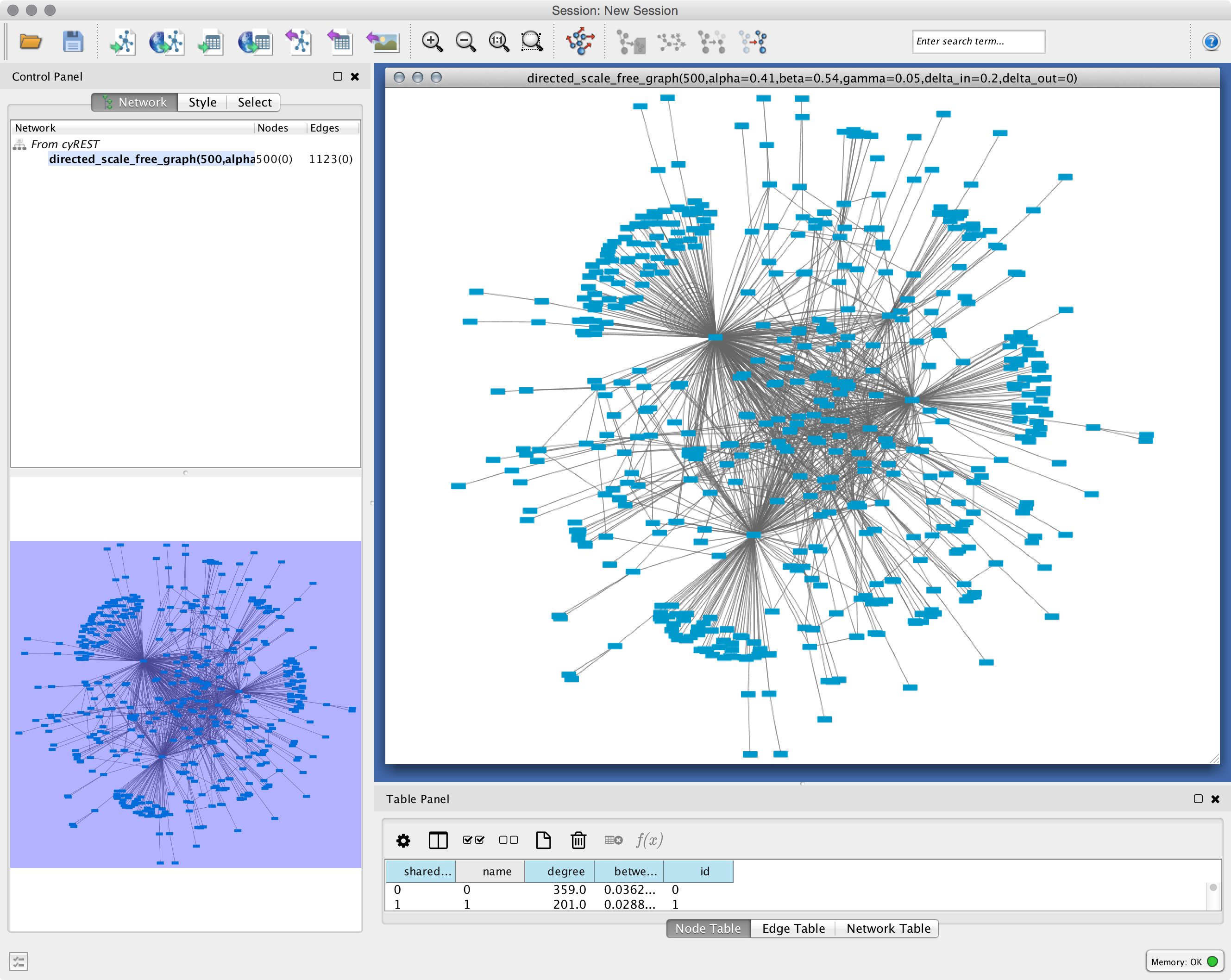
(酵母菌のタンパク質間相互ネットワークをCytoscapeとcyRESTにて可視化)
IPython Notebook1を用いてグラフデータの可視化を行う場合、以下のようなグラフ解析ライブラリに付属する可視化モジュールを用いて行うのが一般的です:
しかし最近のウェブベースの可視化ツールや、デスクトップの可視化ツールに比べると機能的な部分やインタラクティブ性の面で少々物足りなさがあるのもまた事実です。そこでこれらのライブラリの解析機能と、Cytoscapeのインタラクティブな描画機能などを同時に利用できるようにするために作ったのが、今回紹介する__cyREST__と__py2cytoscape__ライブラリです。基本的な考え方はとてもシンプルで、PythonやRといったデータ解析のための標準的な環境からCytoscapeの描画機能などにアクセスできるようにしようというものです。
cyRESTとは?
cyRESTはCytoscape用の拡張機能2で、Cytoscapeの各種データや機能に、RESTfulなAPIからアクセスできるようにしたものです。簡単な例として「今Cytoscape内にある全てのネットワークのID」にアクセスしたい場合、
http://localhost:1234/v1/networks
と言うURLに対してGETリクエストを行うことで可能です。同じように、Cytoscape内にあるリソースには
# NETWORK_IDに対応するネットワークをJSONとして取得する
http://localhost:1234/v1/networks/NETWORK_ID
# NETWORK_IDに対応するネットワーク内の全てのノードを取得する
http://localhost:1234/v1/networks/NETWORK_ID/nodes
# 同エッジ
http://localhost:1234/v1/networks/NETWORK_ID/edges
# ネットワーク内の特定のノードを取得する
http://localhost:1234/v1/networks/NETWORK_ID/nodes/NODE_ID
# ネットワークのノードに関する全プロパティ(テーブル)を取得する
http://localhost:1234/v1/networks/NETWORK_ID/tables/defaultnode
# スタイルのリストを取得する
http://localhost:1234/v1/styles
# 特定のネットワークに指定された自動レイアウトアルゴリズムを適用する
http://localhost:1234/v1/apply/layouts/LAYOUT_NAME/TARGET_NETWORK_ID
基本的なAPI設計はCRUDオペレーションに各種リソースをマッピングするというリソース志向のデザインになっていますので、各種Web APIを利用したことのある方には比較的わかりやすいと思います。つまり、これは言語非依存のAPIであり、HTTPクライアントさえあればどの言語からでもアクセスできます。
py2cytoscapeとは?
このように一般的なHTTPクライアントを用いれば、どの言語からもCytoscape内のリソースにアクセスできるのですが、生のWeb APIをコールするコードがどうしても冗長になってしまいます。特に、___PUT/POST___を行う場合、どのような形でデータを送れば良いのかというフォーマットに関してはその都度ドキュメントを参照するしかありません。これをできる限り解消するために作られたPython用ラッパー兼ユーティリティー集がpy2cytoscapeです。
具体的に、ラッパーを使う場合と使わない場合では以下のような差が出ます。これはCytoscape側にからのネットワークを生成するためのコードです:
# Popular HTTP client for Python
import requests
# Built-in JSON library
import json
# Basic Setup
PORT_NUMBER = 1234
BASE = 'http://localhost:' + str(PORT_NUMBER) + '/v1/'
# Header for posting data to the server as JSON
HEADERS = {'Content-Type': 'application/json'}
# Define dictionary of empty network
empty_network = {
'data': {
'name': 'I\'m empty!'
},
'elements': {
'nodes':[],
'edges':[]
}
}
res = requests.post(BASE + 'networks?collection=My%20Collection', data=json.dumps(empty_network), headers=HEADERS)
new_network_id = res.json()['networkSUID']
print('Empty network created: SUID = ' + str(new_network_id))
一方で、py2cytoscapeを用いると以下のようになります:
from py2cytoscape.data.cyrest_client import CyRestClient
cy = CyRestClient()
network = cy.network.create(name='My Network', collection='My network collection')
print('Empty network created: SUID = ' + str(network.get_id()))
このように、冗長性を減らすのと、IDEなどの文法サポートなどが得られやすいようにするのがこのライブラリの目的です。このほかにも、
- Canvasを使った描画ウィジェットの埋め込み(後述)
- NetworkX/igraphなどのオブジェクトからCytoscapeスタイルのJSONへの相互変換
などの機能が含まれます。
現在はPython用のラッパーしかありませんが、R用も順次公開予定です。
実際の作業
ここからは実際の利用例を紹介します。Cytoscapeがどのようなものかご存じでない方には、こちらの記事が参考になると思います。
動作環境
以下の環境にて動作確認を行いました。
- Mac OS X 10.10.3
- Java SE 8 u45
- Cytoscape 3.2.1
- cyREST 1.1.0
- Python 3.4.3 (Anaconda distributionを利用)
- py2cytoscape 0.4.4
Python側の準備
IPython Notebookなどのインストールはあらかじめ行ってあるという前提で話を進めます。一応3.4/2.7系でテストしていますが、問題があれば連絡ください。
実行に必要なのはpy2cytoscapeのみで、これは以下のコマンドでインストール可能です
pip install py2cytoscape
DockerやEC2などでNotebookサーバー走らせる場合、ノートブックのサーバーとCytoscapeを実行しているマシンが異なることに注意してください。Cytoscapeを実行しているマシンのIPアドレスとポートを適切に設定しない場合、これらのサンプルは動きません。
Cytoscape側の準備
こちらの準備はシンプルで、App ManagerからcyRESTをインストールするのみです。メニューの___Apps→App Manager___からApp Managerダイアログを呼び出し、cyRESTを選択してInstallボタンをクリックするだけです。
__デフォルトのポートは1234です。__変更は、___Edit-Preferences-Properties...___からプロパティ設定画面を呼び出し、___rest.port___に数値を与えることで変更できます。
実行の手順
準備が整ったら、ちゃんと動いているのかを確認します。
- Cytoscapeをスタートする
- ブラウザを開いて以下のURLを開く
3.http://localhost:1234/v1 - 以下のような結果が返ってくればcyRESTが作動しています。
次にノートブックを作成します
- IPython Notebookを実行する
- ブラウザを開いて新しいノートブックを作成する
- 以下のコードを実行する
import json
from py2cytoscape.data.cyrest_client import CyRestClient
cy = CyRestClient()
print(json.dumps(cy.status(), indent=4))
{
"apiVersion": "v1",
"numberOfCores": 8,
"memoryStatus": {
"maxMemory": 28217,
"usedMemory": 513,
"freeMemory": 2645,
"totalMemory": 3159
}
}
IPアドレスとポート
実際にデータ解析をする場合、IPython NotebookはAWSなどのクラウドで、Cytoscapeとブラウザはローカルで、という場合も多いと思います。この場合、CyRestClientにアドレスとポートを渡すことで、Cytoscapeが走っているマシンの場所を指定することが可能です
cy = CyRestClient(ip='127.0.0.1', port=1234)
これで全ての準備が整いました。ここからは実際にCytoscapeをIPython Notebookの外部描画エンジンとして利用するごく簡単な例を紹介したいと思います。
Hello (Graph) World
ここではごく簡単な例を紹介します。できれば環境を整えて、実際にノートブックを実行してみてください。
NetworkXによるランダムネットワーク作成
使うグラフデータはなんでも構わないのですが、ここではNetworkXを利用してスケールフリーネットワークを生成します。
import networkx as nx
g = nx.scale_free_graph(500) # 500ノードのネットワーク
各種統計量計算
このグラフのDegreeとBetweennessを計算します。
deg = nx.degree(g)
btw = nx.betweenness_centrality(g)
nx.set_node_attributes(g, 'degree', deg)
nx.set_node_attributes(g, 'betweenness', btw)
Cytoscapeに送信
一つのコマンドで、直接NetworkXのグラフオブジェクトをCytoscape側に送ることが可能です。この際、ノートやエッジ、ネットワークのプロパティ(attributes)も同時に送信できます。
g_cy = cy.network.create_from_networkx(g)
自動レイアウトアルゴリズムを適用
ここではKamada-Kawaiアルゴリズムを用いて、Cytoscape側でレイアウトを計算させて描画します。
cy.layout.apply(name='kamada-kawai', network=g_cy)
この時点である程度の描画が行われているのが確認できると思います。
別のVisual Styleを適用する
Cytoscapeでは、プロパティから視覚要素への各種マッピングを定義したものを___Visual Style___と呼びます。ここではプリセットの中から、___Directed___というものに切り替えます。
directed = cy.style.create('Directed')
cy.style.apply(directed, network=g_cy)
少々分かりにくいですが、Styleを_create_で呼び出すことにより、そのStyleへのリファレンスを作る形になっています。
エッジを束ねて見やすくする
これはスケールフリー・ネットワークなので、一部のノードに接続が集中しています。これを見やすくするために、力学シミュレーションを使ってエッジを束ねます(Edge Bundling)
cy.edgebundling.apply(g_cy)
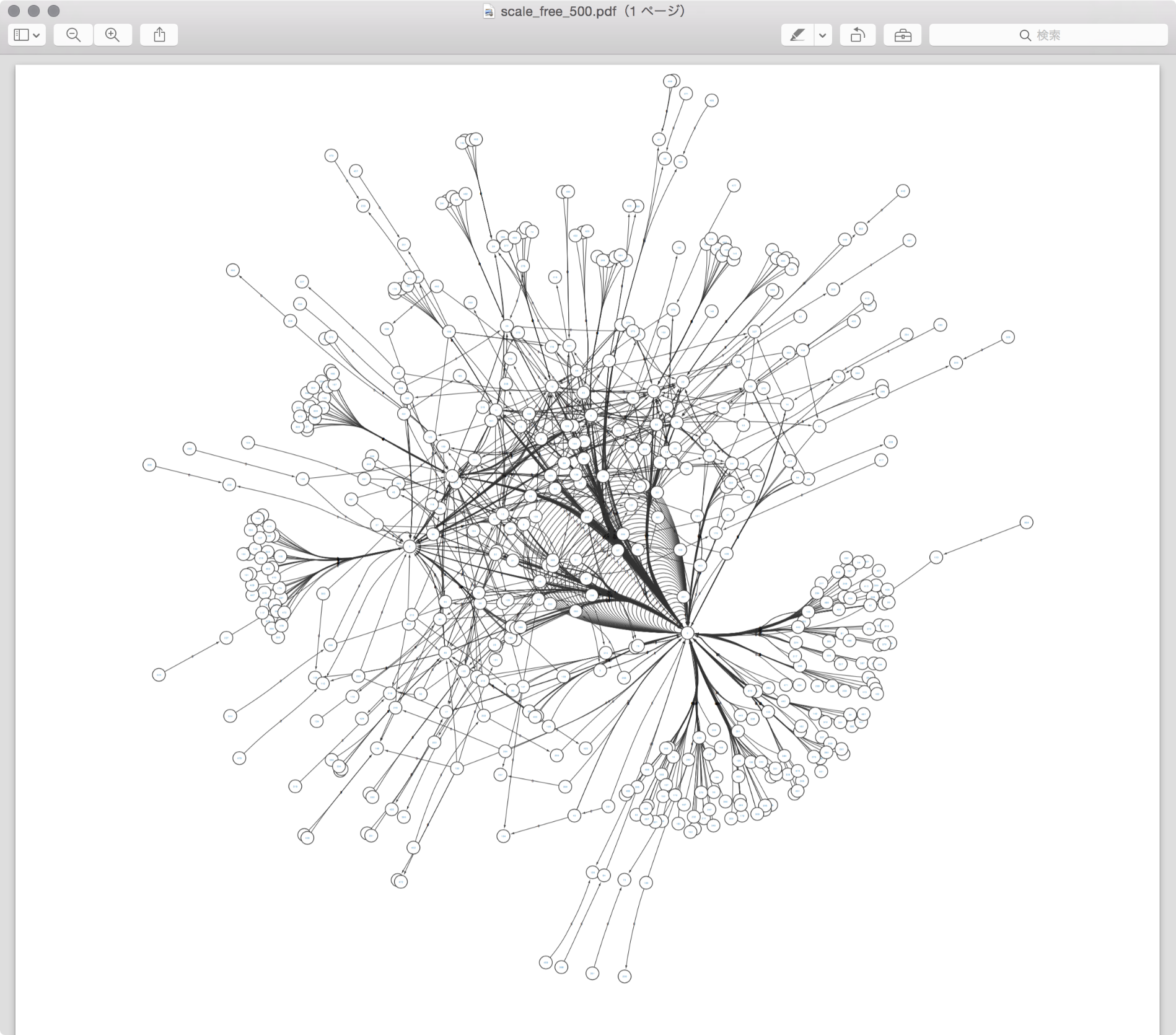
画像としてノートブックに埋め込む
Cytoscapeに内蔵された画像出力機能を使って、画像ファイルとしてネットワークを埋め込んだりファイルに出力したりすることが可能です。現在のバージョンでは、PNG, SVG, PDFをサポートしています。
# PNG
network_png = g_cy.get_png()
from IPython.display import Image
Image(network_png)
# SVG
network_svg = g_cy.get_svg()
from IPython.display import SVG
SVG(network_svg)
# PDFをファイルに保存
network_pdf = g_cy.get_pdf()
f = open('scale_free_500.pdf', 'wb')
f.write(network_pdf)
f.close()
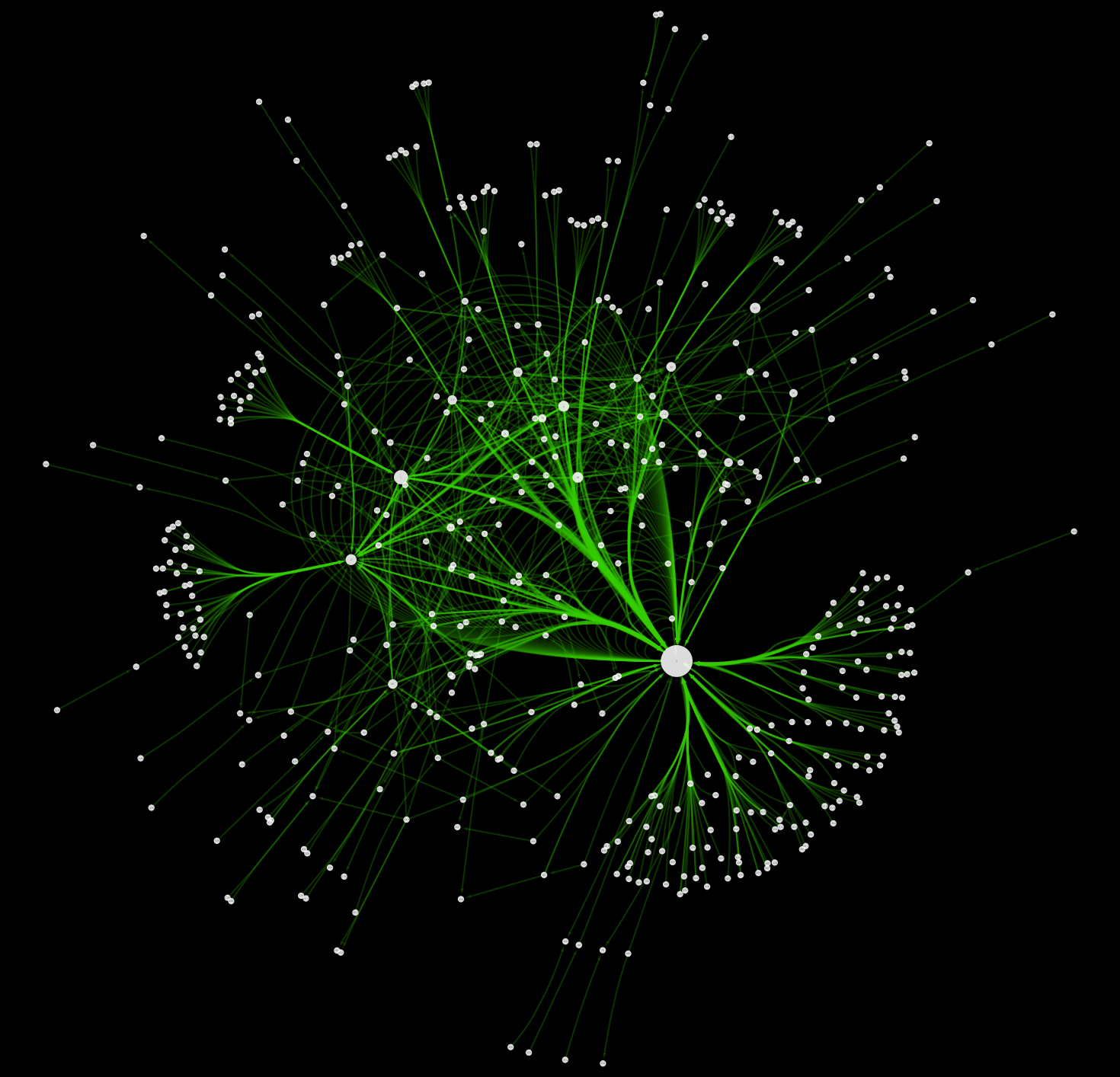
JavaScriptのインタラクティブなオブジェクトとして埋め込む
py2cytoscapeライブラリにはCytoscape.js3を利用したHTMLウィジェットが内蔵されているので、これを利用すれば簡単なコードでJavaScriptによるCanvasを用いたネットワーク描画が可能です。
import py2cytoscape.cytoscapejs as renderer
view = g_cy.get_first_view()
# style_for_widget = cy.style.get(my_yeast_style.get_name(), data_format='cytoscapejs')
renderer.render(view, 'Directed', background='radial-gradient(#FFFFFF 15%, #DDDDDD 105%)')
現在、Edge Bundlingを含む一部の機能がまだ実装されていませんので、完全に再現されるわけではありませんが、それらも徐々にサポートする予定です。
手動でこの結果をさらに編集する
ここまではコードにより描画してきましたが、その結果をGUIからインタラクティブに眺めたり修正したりするのも容易です。これは静止画像を生成するだけのライブラリでは困難な部分です。例えば、ノードの位置の微妙な調整や、細部へのズームインなどがそれにあたります。
上の例は、バックグラウンドの色を変えたり、Betweennessをノードの大きさにマッピングしただけの簡単なエディットですが、コードを書いて再現性のある可視化を行うことと、GUIからの直感的なエディットで直接見ながら可視化結果をいじるという双方のメリットが享受できます。
まとめ
今回は駆け足で基本的な機能を紹介しただけですが、実際のデータ解析を行うには、スタイルのコードを使った生成や、各種グラフ解析ライブラリの結果の利用などが便利です。次回以降にこれらCytoscape + Pythonならではの使い方を紹介したいと思います。
-
現在はJupyter Notebookとしてリブランディング中 ↩
-
_App_と呼ばれます。Cytoscape App Storeから入手可能。 ↩
-
ネットワーク描画と解析に特化したJavaScriptライブラリ。Cytoscapeの開発チームが開発とメンテナンスを行っている。 ↩