はじめに

(ただし実際の実行にはBeakerサーバーが必要ですので、以下の手順を参考にしてください)
前編では、Jupyterで埋め込みカスタムJSウィジェットを作成する場合の具体例と、Beaker Notebookの紹介を行いました。今回は実際にBeakerを使う場合の手順を見て行きたいと思います。
環境の準備
ほとんどの場合において、「素のPython/R/JS」で作業が完結するというのは稀です。たとえば、データの下準備の時は、pandasを使いたいことが多いでしょうし、matplotlibなどで描画を行いたい場合も多いでしょう。ノートブック型アプリケーションは作業の過程を残すのには適していますが、その作業がどのような環境で実行されたかまでは記録することができません。
Dockerを使った可視化環境のポータブル化
そこで、ノートブック型アプリケーションと組み合わせて利用することによりさらに再現性を高めることができるのが、Dockerをはじめとするコンテナ技術です。上記の通り、多言語利用での可視化・解析作業で問題になるのは、__いったいどんな環境でその作業を行ったのか__という記録を残すのが難しいという点です。もちろん仮想マシンイメージという手はありますが、一つのノートブックに対して一つのコンテナ環境という形で記録が残せる上、それらを継承して新たな作業環境を簡単に構築できるという点で、この手の作業とDockerの相性は事情に良いです。
Beaker Notebookプロジェクトでも、サポートされているPython, Node.js, R, Juliaといった言語を即使えるDockerイメージを公開しています。今回は、このイメージを拡張する形で作業を進めます。今回のテーマはDockerの学習ではありませんのでDockerに関する詳細は割愛しますが、Docker Toolboxのリリース後は、前提知識がほぼない方でもかなり気軽に利用できるようになってきましたので、是非挑戦してみてください。
イメージに必要なものを追加する
公式ページにあるイメージには一通りのプログラミング言語があらかじめ用意されているのですが、ここに追加のパッケージを加えてみます。基本的にはインストールに必要なコマンドをRUNで呼び出すだけです。
今回利用するDockerfile
FROM beakernotebook/beaker
# Add Extra library for data analysis
# R Libraries
RUN Rscript -e "install.packages('igraph',,'http://cran.us.r-project.org')"
# Python 3 libraries
RUN /home/beaker/py3k/bin/pip install requests networkx py2cytoscape
# Add a new directory for user's notebooks
RUN mkdir /home/beaker/notebooks
この例では、R言語でよくグラフ解析に使われるigraphと、同じくPythonで使われるNetworkX、そしてCytoscape.jsへのデータ変換ユーティリティーを含むpy2cytoscapeをインストールしています。
Beaker Notebookの実行
BeakerもJupyterと同じく、JSON形式のファイルとしてノートブックを管理しています。このため、ローカルのファイルシステムにDockerファイルとノートブックを置き、それをマウントする形でコンテナを実行すると非常に便利です。
イメージのビルド
docker build -t keiono/beaker .
イメージの実行
docker run -p 8800:8800 -v $PWD:/home/beaker/notebooks -t keiono/beaker
これで現在のディレクトリがコンテナにマウントされた状態でノートブックサーバーが実行されます。
この後にログインのためのパスワードが表示されますので、それを使ってhttpsでアクセスします。Dockerのホストが192.168.99.100の場合、ブラウザで以下のアドレスにアクセスするとパスワードの入力を求められるので、ターミナル上に表示されたそれを入力します。
https://192.168.99.100:8800
今回の例で用いたDockerfileもこちらにありますので、forkしたのちに自由にコマンドを追加して、自分の作業に必要な環境に改造してください。
ノートブックでの作業
サンプルノートブックを開く
こちらのレポジトリにサンプルのノートブック(graph-final.bkr)が入っているのでそれを開きます。

これを実行します。セルの実行方法はJupyterと同じで、___CTR+Return___です。順番に実行していくと、最後のセルがこのような状態になると思います:

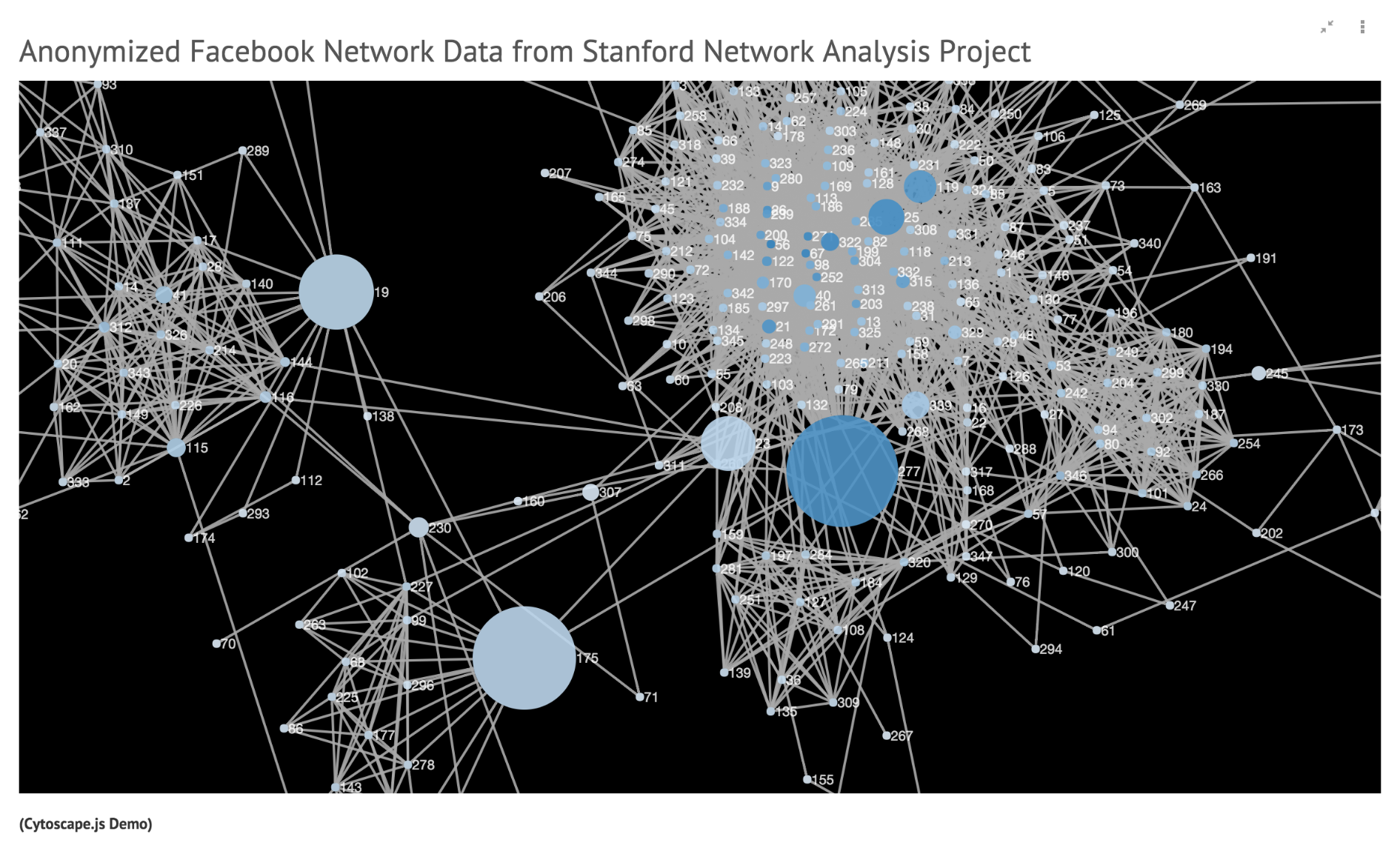
これは、実際の可視化のセルが実行中になっているということですので、一つ上のセルへ戻ると、このようなものが表示されているはずです
その中のノードをクリックすると、表示が変わって、そのノードに直接接続されているものだけが浮かび上がるはずです
このインタラクティブなセルは、Cytoscape.jsを埋め込みながら、ノートブック上で作り上げたものです。この例では、カスタムのネットワークレンダリング用セルを作成しましたが、実際には何を使っても構いません。D3.jsならば最初から用意されていますので、いきなりJavaScriptのセルにコードを書き始めることも可能です。
この先は、実際の工程をもう少し詳しく見てみます。
データの共有: Autotranslation
この例では、Beakerの主要な機能である___Autotranslation___を強調するために、R, Python3, JavaScriptの三つの言語間でデータをやり取りしています。これがBeakerの特徴的な機能で、複数言語間でこの機能を使い、データの直接的なやり取りが可能です。
この例では、最初のセルでRを使い、データフレームにエッジリストを読み込ませています
df1 <- read.table('/home/beaker/notebooks/fbnet.edges', header=FALSE, sep=' ')
colnames(df1) <- c('source', 'target')
# Save it as a shared beaker object
beaker::set('df1', df1)
最後の行に見慣れない記法があると思いますが、これがBeakerのAutotranslationのすべてです。要するに、グローバルに露出したこのbeakerオブジェクトに対し、各言語からオブジェクトや値を渡すことによって、それが自動的にJSONへと翻訳され、各セル間でデータのやり取りが可能になっているのです。
このデータにPythonからアクセスする場合は、pandasのDataFrameとして自動的に変換されたものが使えます。
RのDataFrame → PandasのDataFrame → NetworkXのグラフ → Cytoscape.jsのJSON
df2 = beaker.df1 # これだけで自動的にPandasのDFへ変換される
df2[["source", "target"]] = df2[["source", "target"]].astype('str')
# そのデータフレームからNetworkXのオブジェクトへ変換
g2 = nx.from_pandas_dataframe(df2, "source", "target")
# Calculate Betweenness Centrarity of the graph
cent_1 = nx.betweenness_centrality(g2)
deg = nx.degree(g2)
nx.set_node_attributes(g2, 'b-cent', cent_1)
nx.set_node_attributes(g2, 'deg', deg)
# Convert networkX data into Cytoscape.js JSON
net1 = nxutil.from_networkx(g2)
# Save it into Beaker object
beaker.net1 = net1 # 最終的に度数などが含まれるグラフをもう一度登録する
# Save range
beaker.net1_deg_range = (min(deg.values()), max(deg.values()))
最後の部分で、私が以前作ったユーティリティーでNetworkXのオブジェクトをCytoscape.jsでレンダリングできる形に変換しています。これで可視化に使えるデータができました。
HTMLのひな形を作る
データができたら、それを描画するためのセルをHTMLで作ります。とは言っても、普通は簡単なCSSと、描画ターゲットとなるタグのみを含むシンプルなものになるはずです:
<style>
# cyjs {
width: 100%;
height: 600px;
background-color: #000000;
}
h1 {
color: #555555;
}
</style>
<h1>Anonymized Facebook Network Data from Stanford Network Analysis Project</h1>
<div id="cyjs"></div>
<h3>(Cytoscape.js Demo)</h3>
可視化コンポーネントを作る
ここまできたら、あとは試行錯誤しながらデータを可視化するのみです。Cytoscape.jsの詳細は今回の話題から外れますので深入りはしませんが、ポイントとなるのはここです:
elements: beaker.net1["elements"]
Pythonを使ってCytoscape.js形式に変換したものを、beakerオブジェクトを通じて直接取り込んでいます。これを使えば、可視化に新たなデータを盛り込みたいと思った場合も、前のPythonやRのセルでデータを加工し直し、そのままJSのセルへ戻ってまた可視化部分のコーディングを続けることができます。これが現状のJupyterでは出来ない部分で、Beakerの最大のアドバンテージだと思います。
最後に
このようにDockerとBeaker Notebookを使えば、複数言語を使いながら一つのノートブックで、データ加工から可視化までをすべてを再現できる形で記録することが可能です。ブラウザとエディタで作る場合は、データの変更がまた別のアプリケーションで行わなければならないことも多いですが、これだとすべてをノートだけで完結できます。
まだ利用例が少ないですが、複数言語を使いたい場合、複雑なJSコンポーネントでの可視化を行いたい場合は、ぜひ利用してみてください。