最近kerasは深層学習(ディープラーニング)を使う人達の間でますます人気が上がってきています。
kerasを簡単に紹介しておくと、深層学習(ディープラーニング)のモデルをTensorFlowまたはCNTK、Theano上でより簡単に素早く作って実行できるニューラルネットワークのライブラリです。
keras自体はpythonで実装されているのですが、RstudioがRから直接kerasを呼べるRパッケージkerasを出しています。
今日は、Exploratoryの中でこのRパッケージを通してkerasを使って予測モデルを作ってみます。
問題としては、kaggleの練習問題である、タイタニック号の乗客の生存予測をしてみましょう。
こちらは、年齢、性別、乗船チケットの種類などの乗客の情報から、その乗客がタイタニック号の事故を生き残ることができたのかを予測する問題です。
keras Rパッケージをインストール
プロジェクトリストページのこちらから、Rパッケージメニューを選びます。
パッケージインストールダイアログのこちらで、kerasを指定してインストールします。
プロジェクトの中からも、Rパッケージメニューを選びます。
kerasのチェックボックスをチェックして、このプロジェクトからkerasを使用出来るようにします。
kerasのバックエンドをインストール
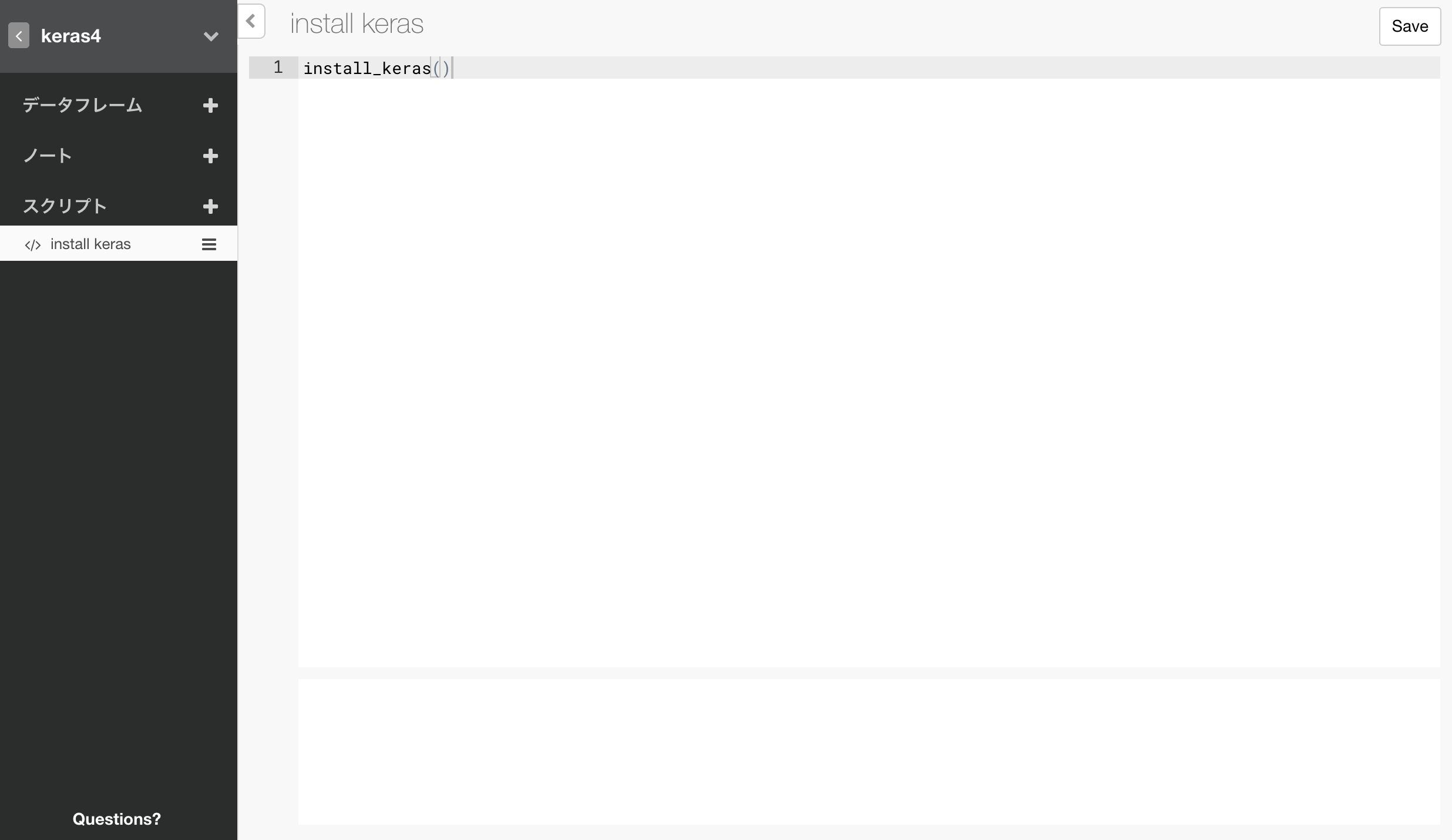
スクリプトを一つ作成して、TensorFlow、python上のkerasパッケージを含めた、kerasのバックエンドをインストールする、以下のコマンドを実行しておきます。
install_keras()
以下のように実行成功のメッセージが出れば成功です。
このコマンドは一度実行すればよいのですが、このスクリプトを残しておくとプロジェクトを開く度に実行されてしまうので、これを避けるため、一度成功したらこのスクリプトを削除しておきます。
Exploratoryのカスタムモデルとしてkerasを使用出来るようにする
Exploratoryの中では、すでに多くの機械学習モデルがUI越しにサポートされていますが、それ以外にも自分の使いたいものをRスクリプトを書くことによって追加できます。
こちらのフレームワークを使って、kerasによるニューラルネットワークモデルを構築するためのコードが以下になります。
以下のコードをコピーして、Exploratoryプロジェクト内にスクリプトを作成します。
# kerasによる二値予測ニューラルネットワークモデルを作成するfunctionを、
# ExploratoryのCustom Model Functionとして実装。
# https://docs.exploratory.io/user-defined-model-function.html
build_keras_model <- function(formula, data, validation_split = 0.2, epochs = 20) {
# dataをvalidation_splitの割合でtraining_dataとvalidation_dataに分割。
data <- data %>% mutate(validation = runif(n()) < validation_split)
training_data <- data %>% filter(!validation)
validation_data <- data %>% filter(validation)
# training output dataを準備
y <- training_data[,colnames(training_data) %in% all.vars(lazyeval::f_lhs(formula))]
y <- data.matrix(y) # data frameからmatrixに変換
# validation output dataを準備
y_v <- validation_data[,colnames(validation_data) %in% all.vars(lazyeval::f_lhs(formula))]
y_v <- data.matrix(y_v) # data frameからmatrixに変換
# training input dataを準備
x <- training_data[,colnames(training_data) %in% all.vars(lazyeval::f_rhs(formula))]
x <- data.matrix(x) # data frameからmatrixに変換
# validation input dataを準備
x_v <- validation_data[,colnames(validation_data) %in% all.vars(lazyeval::f_rhs(formula))]
x_v <- data.matrix(x_v) # data frameからmatrixに変換
# 3層のニューラルネットワークモデルを作成
model <- keras_model_sequential()
model %>%
layer_dense(units = 7, activation = 'relu', input_shape = c(7)) %>% # 1層目に7個のニューロンを設置
layer_dropout(rate = 0.2) %>% # 2割のデータをdropoutする。
layer_dense(units = 5, activation = 'relu') %>% # 2層目に5個のニューロンを設置
layer_dropout(rate = 0.1) %>% # 1割のデータをdropoutする。
layer_dense(units = 1, activation = 'sigmoid') # 3層目で1つの生存確率にまとめ上げる。
model %>% compile(
loss = loss_binary_crossentropy,
optimizer = optimizer_adam(),
metrics = c('accuracy')
)
# モデルにトレーニングデータを学習させる
history <- model %>% fit(
x, y,
epochs = epochs, batch_size = 20,
validation_data = list(x_v, y_v),
verbose = 0
)
# モデル、学習経過(history)、formulaを一つのオブジェクトとして返す。
ret <- list(model = model, history=history, formula = formula)
class(ret) <- c("keras_model")
ret
}
# 学習済みのモデルに新しいデータを渡して予測させるfunction
augment.keras_model <- function(x, data = NULL, newdata = NULL, ...) {
formula <- x$formula
if (is.null(data)) {
data <- newdata
}
data_x <- data[,colnames(data) %in% all.vars(lazyeval::f_rhs(formula))]
data_x <- data.matrix(data_x)
y <- x$model %>% predict_proba(data_x)
ret <- data
ret$y <- as.numeric(y)
ret
}
# モデルからデータフレームとしてモデルサマリビューで表示する情報を抽出するfunction
glance.keras_model <- function(x, ...){
params <- x$history$params
params$metrics <- NULL
params$steps <- NULL
as.data.frame(params)
}
# モデルからデータフレームとしてモデルサマリビューで表示する情報を抽出するfunction
# ここでは学習経過情報を出力する。
tidy.keras_model <- function(x, ...){
as.data.frame(x$history)
}
以下のように、新しくスクリプトをつくって、上のコードをペーストし、Saveボタンをクリックします。
データの読み込み
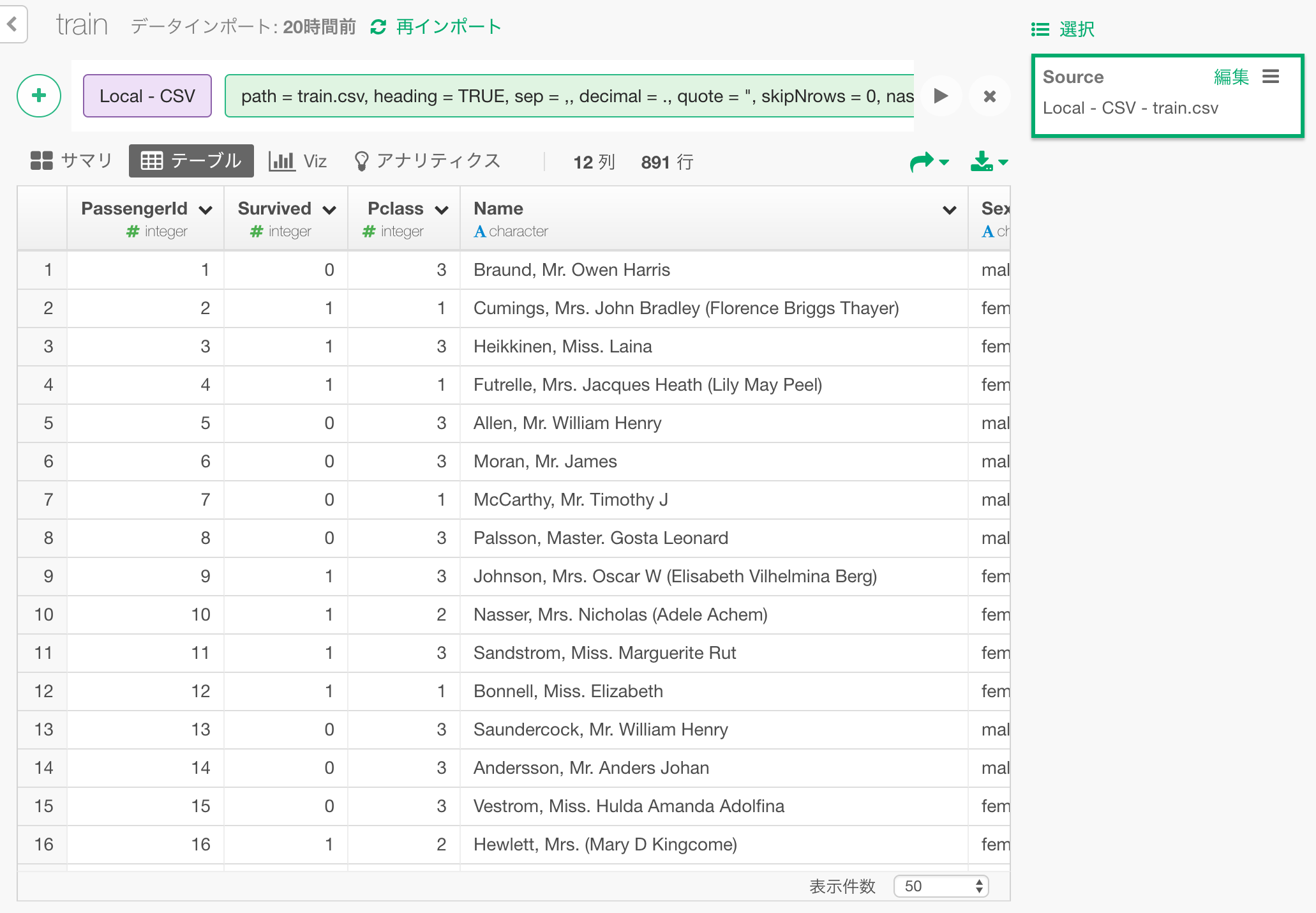
タイタニック号の乗客のデータをこちらのkaggleのcompetionページからダウンロードして、Exploratoryにインポートします。
こちらから、train.csvというデータファイルをダウンロードします。
これを読み込んだところが以下になります。
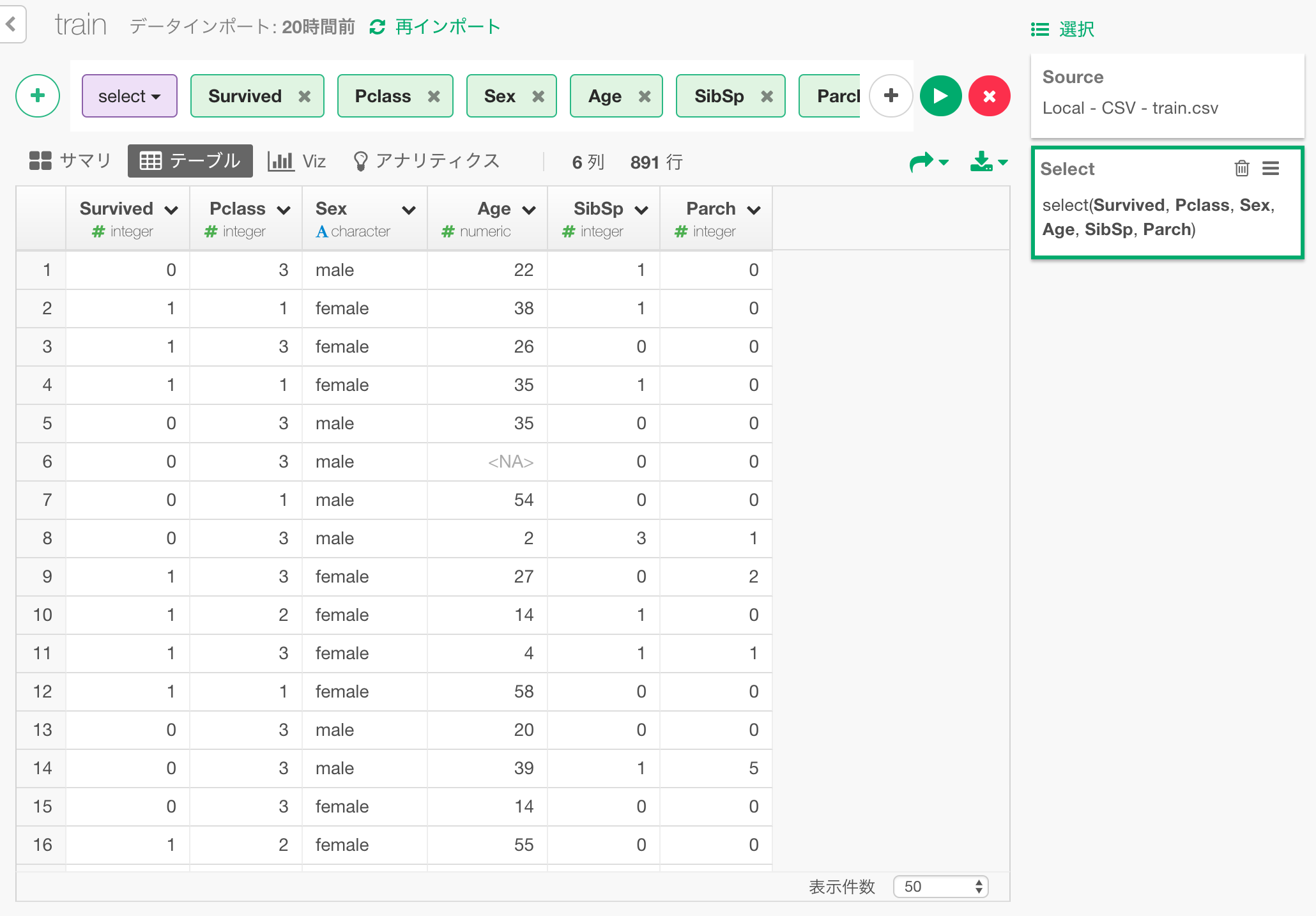
今回、予測に使う列だけを残して他の列は消しておきます。
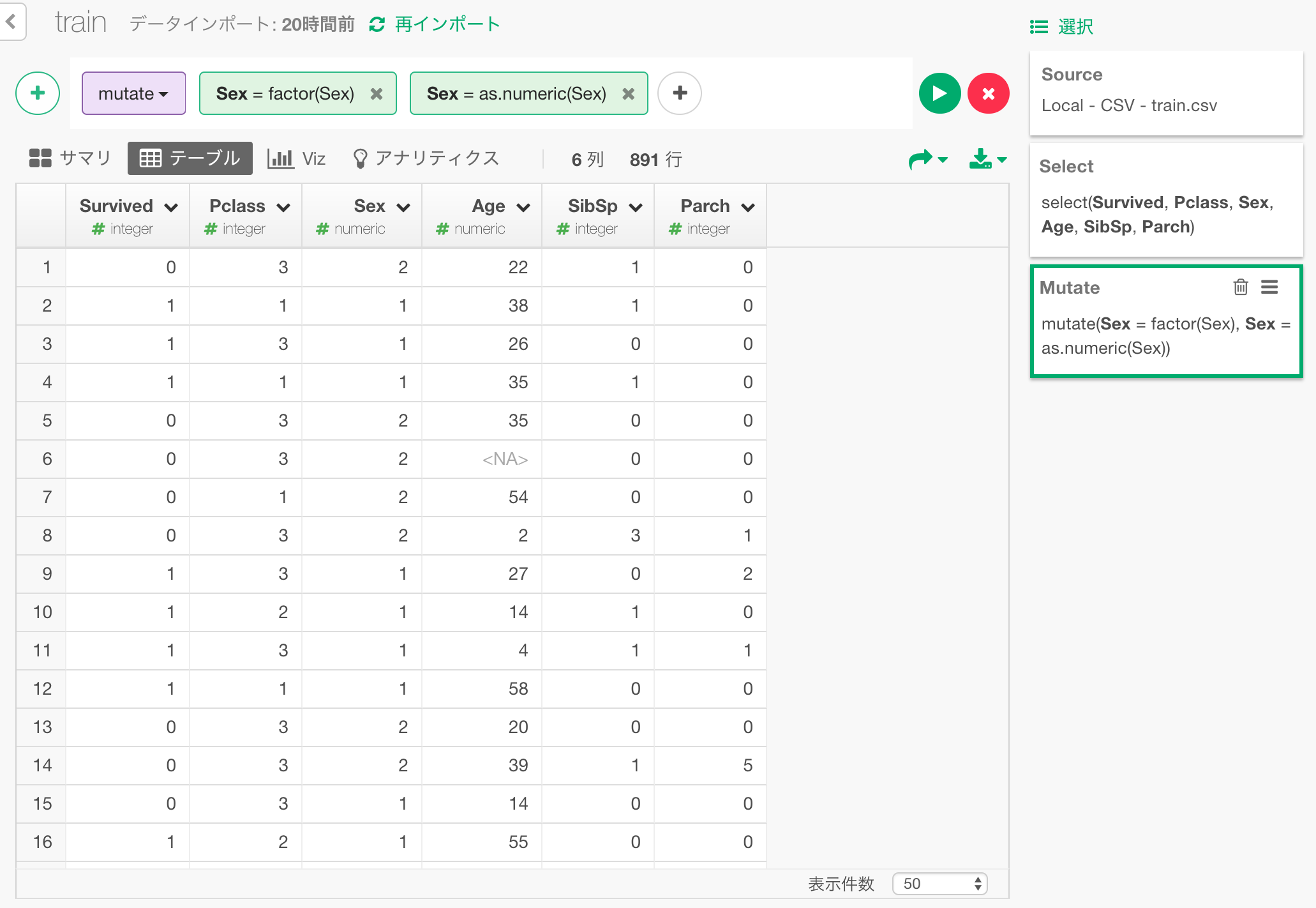
性別データを数字に
kerasのニューラルネットワークモデルに入力するデータは、全て数値である必要があります。
以下のようにして、Factor化して、そのあと数値化することにより、男女を、1と2の二つの数値で表現しなおします。
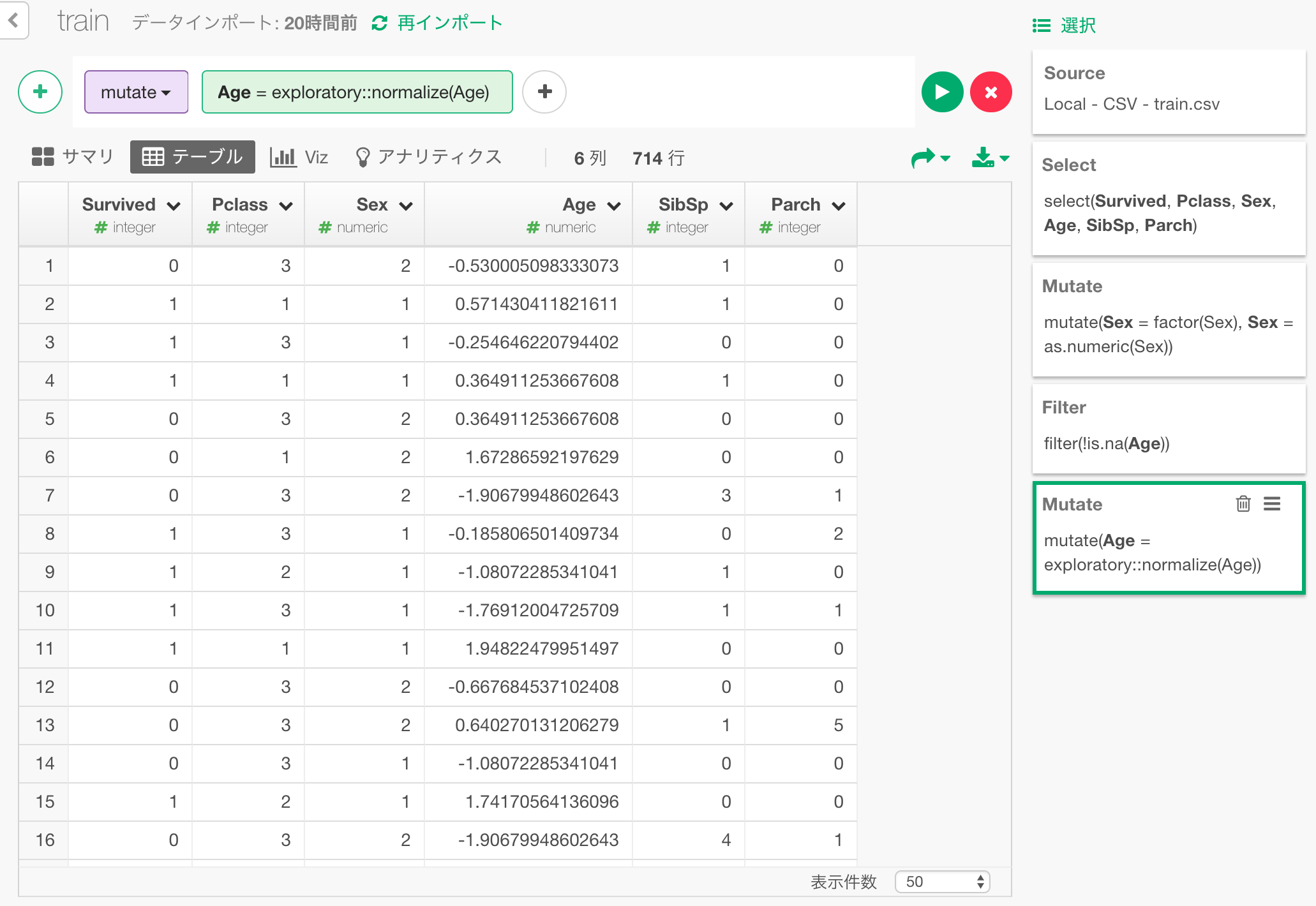
年齢データをfilter、normalize
年齢データにはNA(年齢が記録に残っていない)が混じっていますが、そのままではkerasはこれを扱えません。
今回は単純に、年齢がNAのデータは分析対象から外してしまうことにします。
さらに、年齢以外のデータは1や2などの一桁の数値におさまっていますが、年齢だけが幅の大きい数値データになっています。
学習効率、精度を上げるために、年齢の値の範囲を他と合わせるために、exploratoryパッケージにあるnormalizeという関数を使って標準化(normalize)します。
(kerasにもnormalizeというfunctionがあるため、それと区別するためにexploratory::normalizeとして関数を呼んでいます。)
exploratory::normalize(Age)
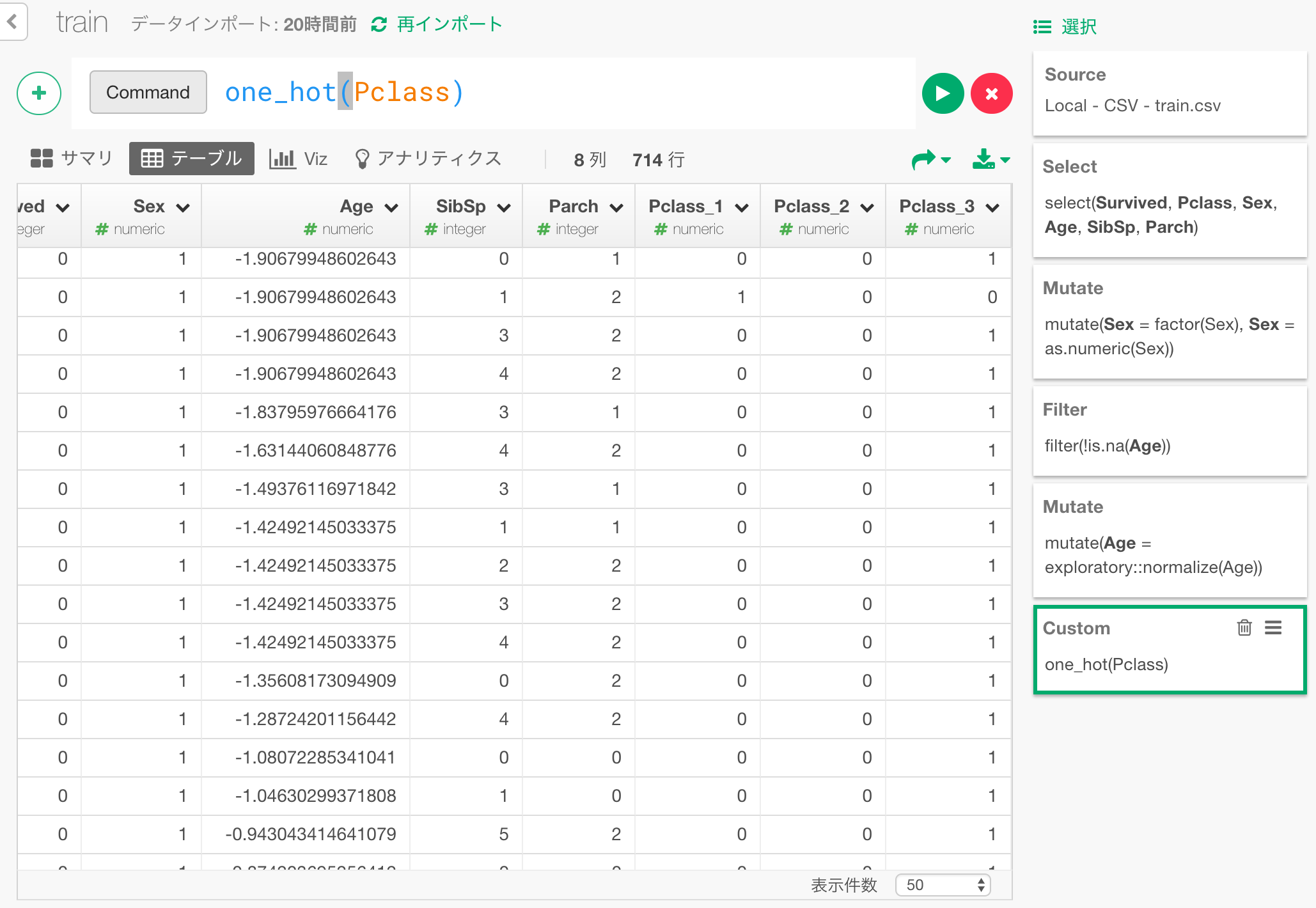
乗船チケットタイプをone-hot encoding
乗船チケットタイプは、大小関係のある数値として扱ってもいいのかもしれませんが、ここではカテゴリーとして扱いましょう。
Pclass列を3つの列、Pclass_1,Pclass_2,Pclass_3に分割して、例えば1st classであるときにはPclass_1に1、その他の列に0が入るといった形に表現しなおします。
こういった変換はone-hot encodingと呼ばれます。
これを行う、one_hotという関数をこちらに用意しましたので、これをコピーしてスクリプトを作成した上で、使用します。
# One-hot encodingのためのutility function
one_hot <- function(df, key) {
key_col <- dplyr::select_var(names(df), !! rlang::enquo(key))
df <- df %>% mutate(.value = 1, .id = seq(n()))
df <- df %>% tidyr::spread_(key_col, ".value", fill = 0, sep = "_") %>% select(-.id)
}
kerasでニューラルネットワークモデルを作成
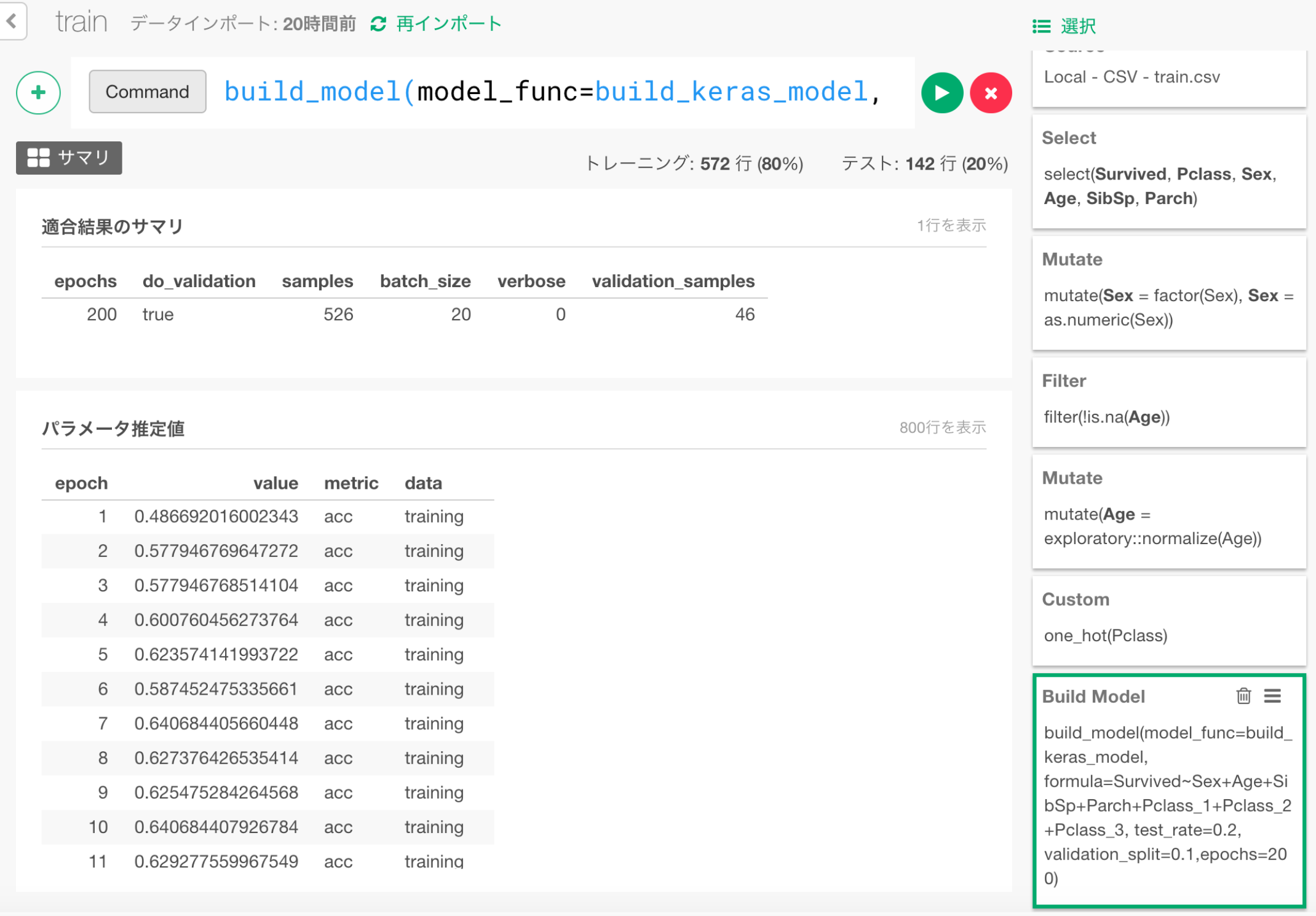
以下のカスタムコマンドで、ここまでに整理、変換してきたデータをもとに、ニューラルネットワークモデルを作成します。
build_model(model_func=build_keras_model, formula=Survived~Sex+Age+SibSp+Parch+Pclass_1+Pclass_2+Pclass_3, test_rate=0.2, validation_split=0.1,epochs=200)
formula=...の部分は、Survived変数を、Sex,Age...といったここに上げられた変数で予測せよ、という意味です。
test_rate=0.2は、データの2割を、あとで予測能力をテストするためのデータとして学習につかわずに取っておくということを指定しています。
validation_split=0.1は、データの1割を、validation(学習しながら途中段階で行うテスト)に使用するため学習に使わないということを意味します。
モデルの作成が完了すると以下のようになります。
学習経過をデータフレームとして抽出、可視化
kerasは、さきほど少し話題に出たvalidationデータをつかって、学習を繰り返すに従って、どのようにモデルが賢くなっていくのか経過を出力してくれますので、それを可視化してみましょう。
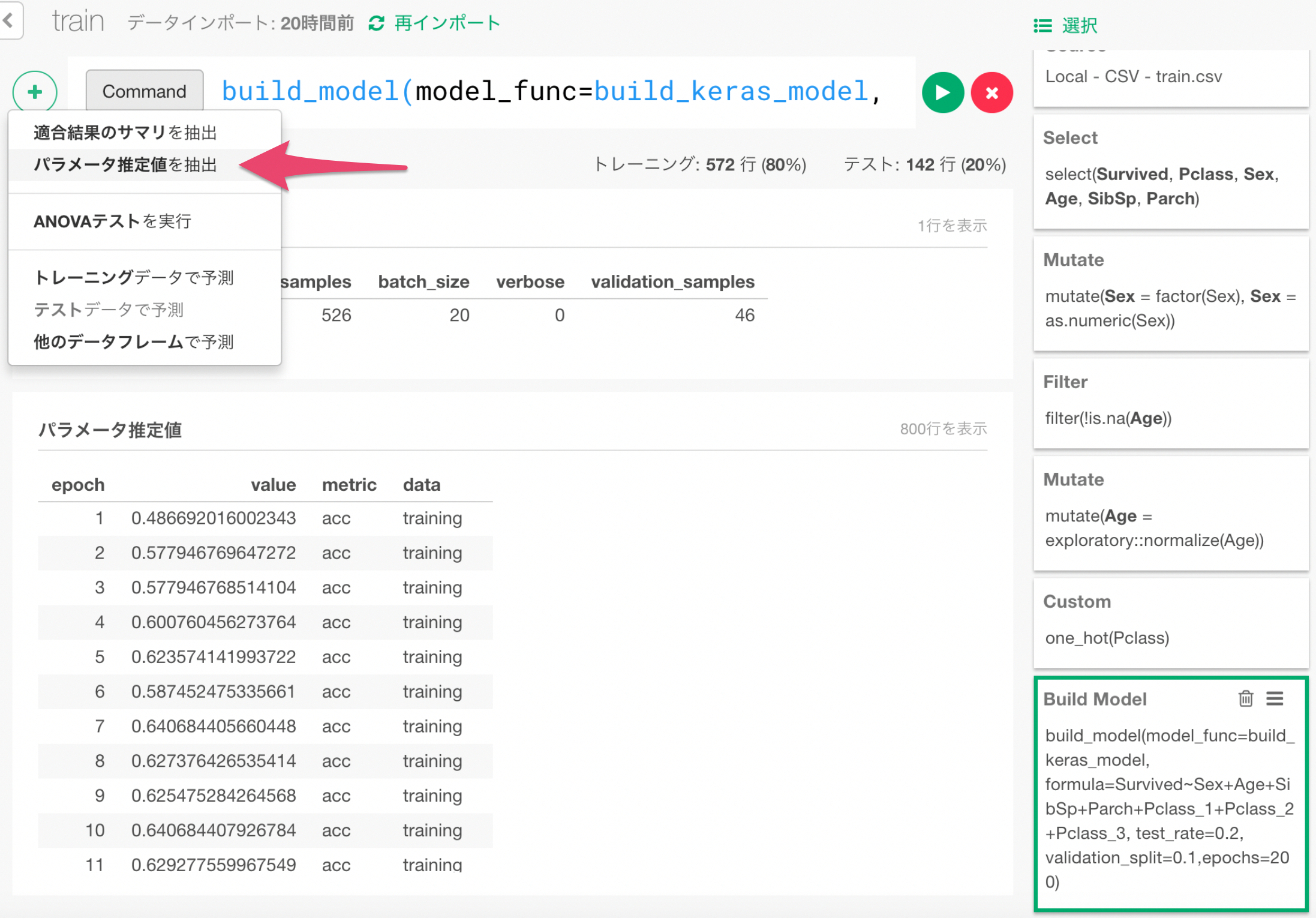
+ボタンメニューから、「パラメータ推定値を抽出」メニューを選びます。
このように、学習経過データをデータフレームとして抽出できます。
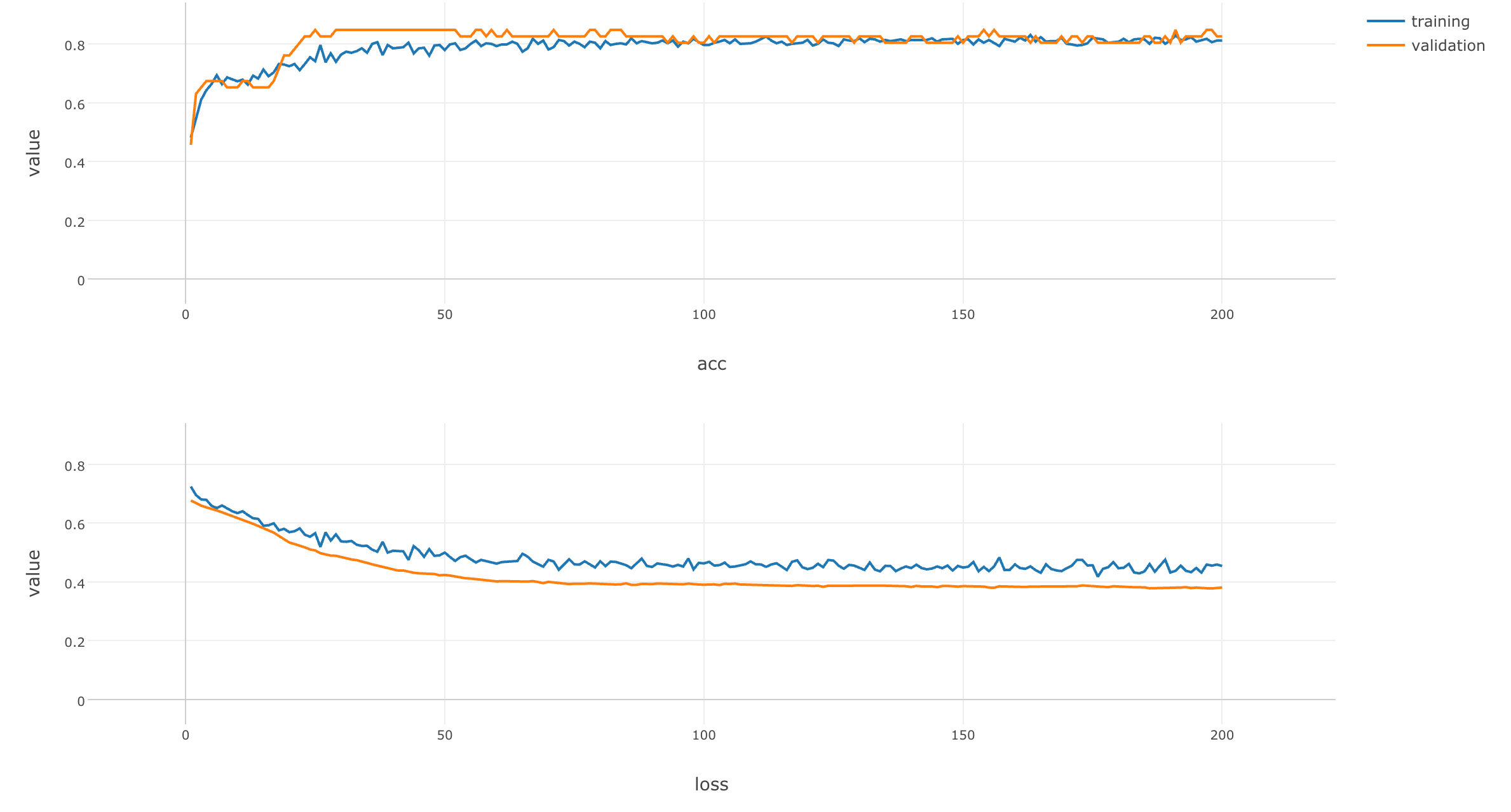
ここには、学習を繰り返すにつれ、accuracy(高いほどモデルは賢い)とloss(低いほどモデルは賢い)がどのように変化したかの情報が入っています。
- epoch : 学習を繰り返した回数
- value : accuracyまたはlossの値
- data : validation dataを元にした値なのか、training dataを元にした値なのか。
- metric : accuracyなのかlossなのか。
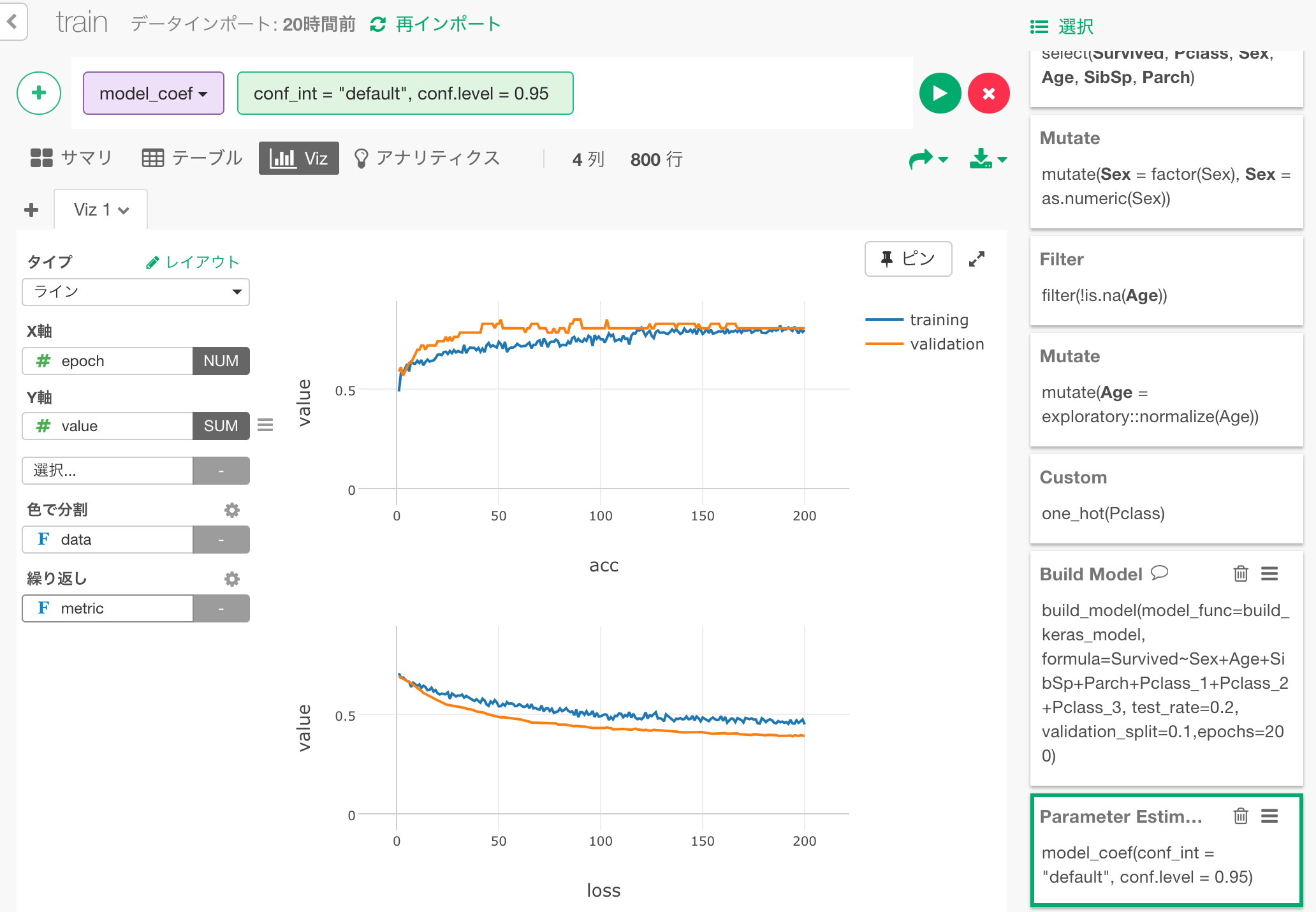
このデータをラインチャートで可視化します。
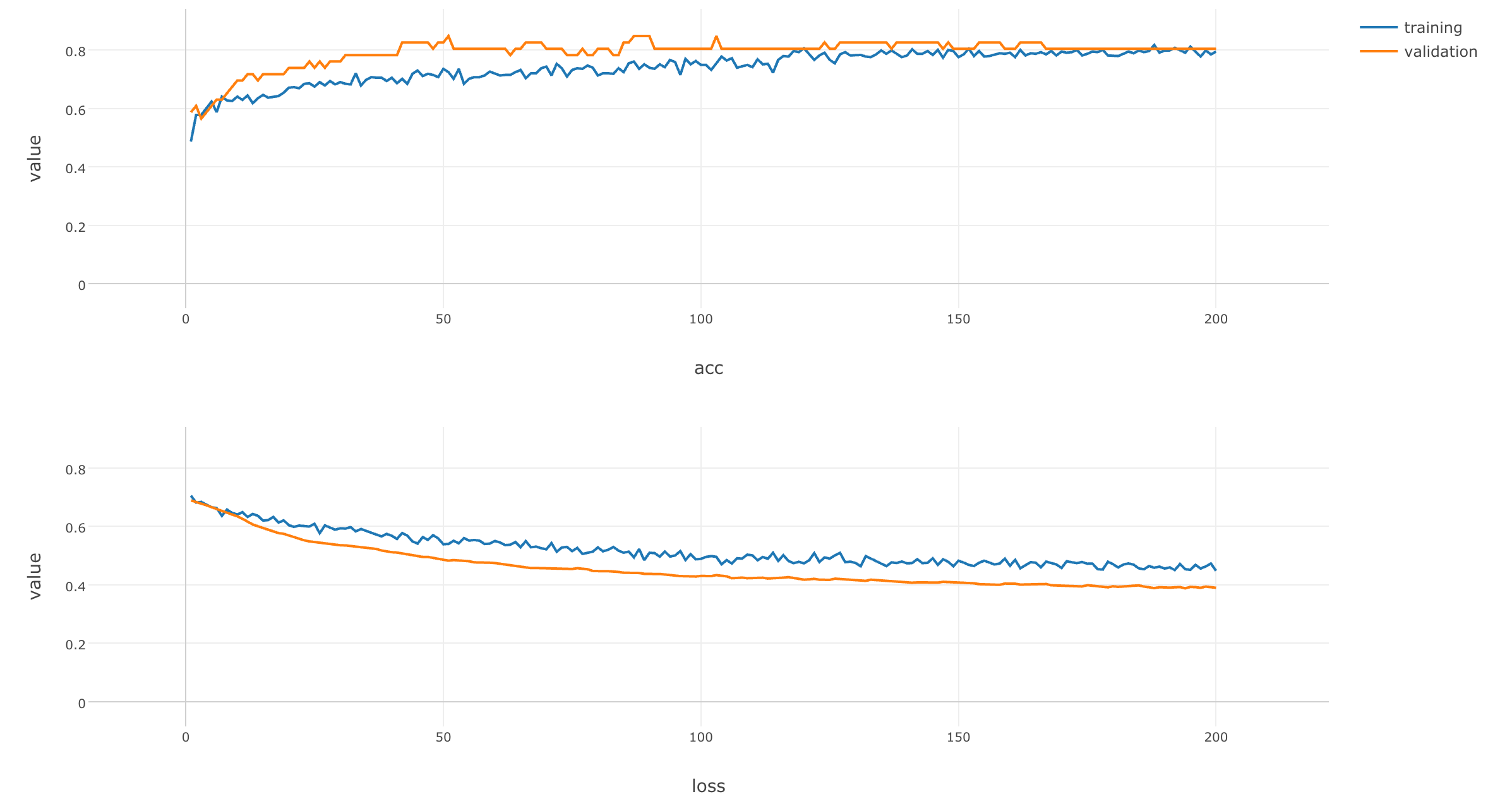
チャートを拡大して見てみましょう。
学習が繰り返されるにつれ、training、validationともに、lossは小さく、accuracyは大きくなっているのが見て取れます。また、これらの変化は学習の終盤ではあまり変化しておらず、このモデルでは、これ以上学習を繰り返しても予測能力の向上はみこめないだろうことも分かります。
モデルにテストデータを入力して予測
さて、このモデルを作成する前に、2割のデータを学習に使わず、テスト用にとっておくという指定をしました。
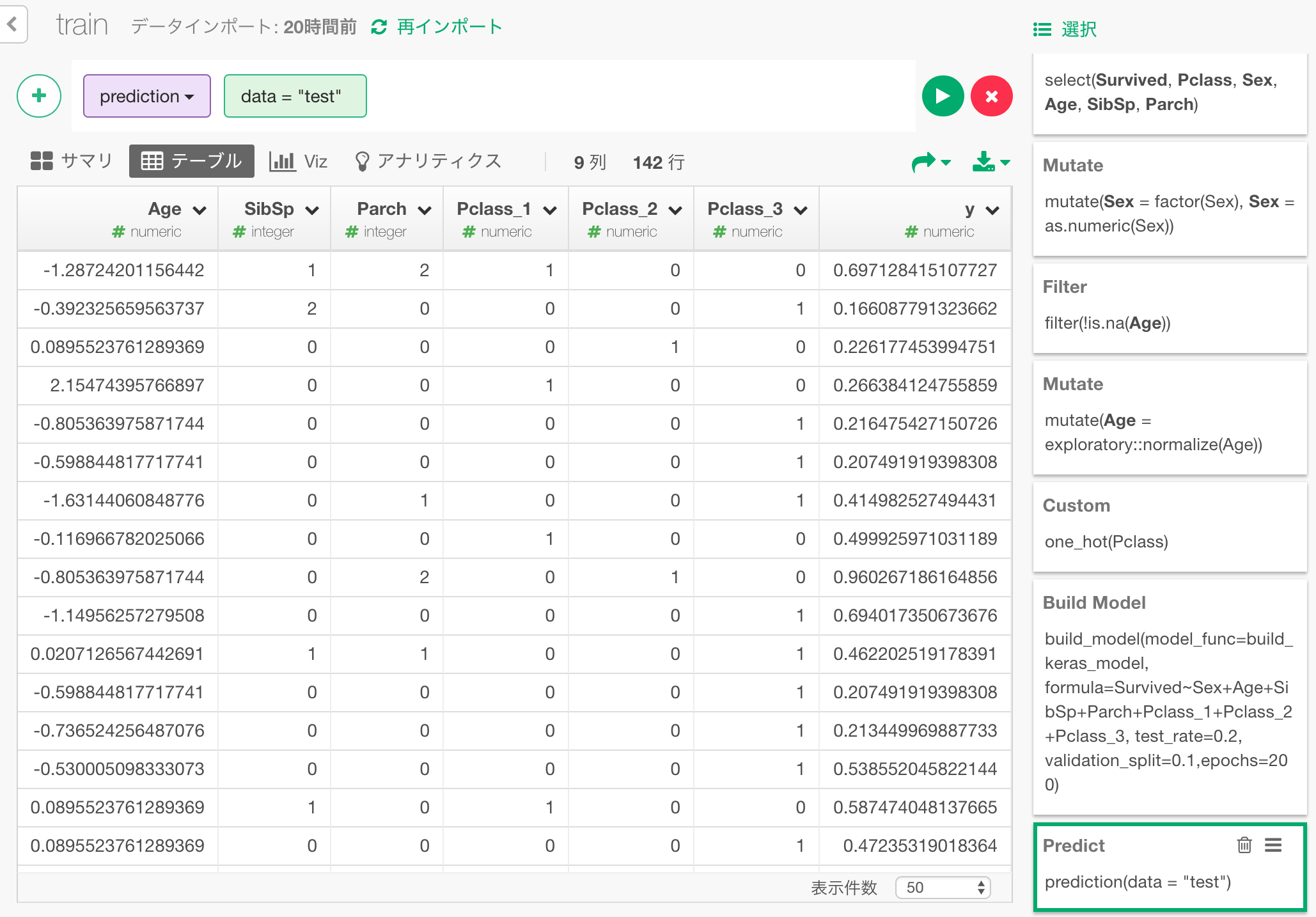
その2割のテストデータについて、このモデルで乗客の生存を予測してみましょう。
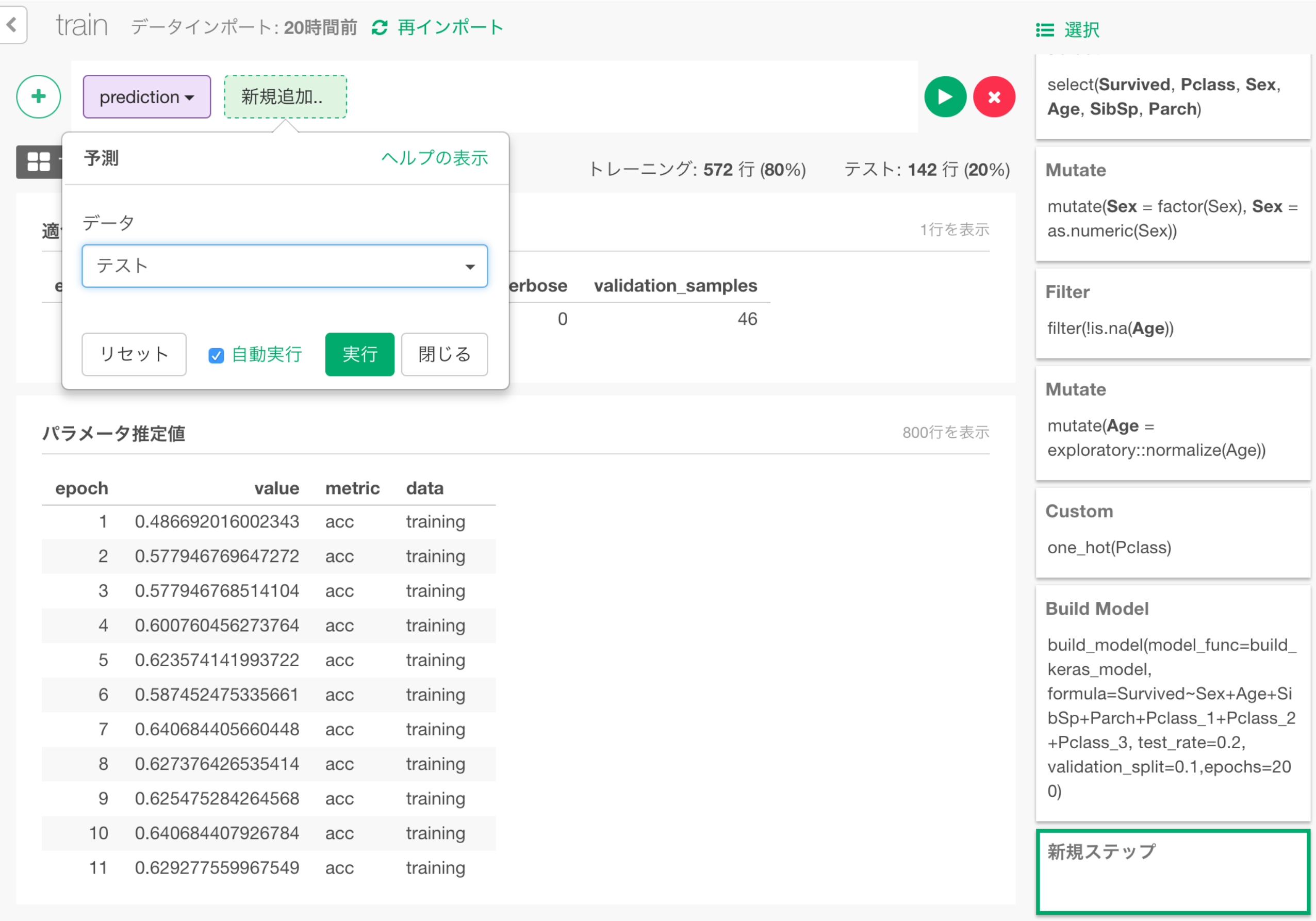
+メニューから予測ダイアログを呼び出し、データとして「テスト」を選択します。
y列にニューラルネットワークモデルが予測した生存確率が入っているのがわかります。
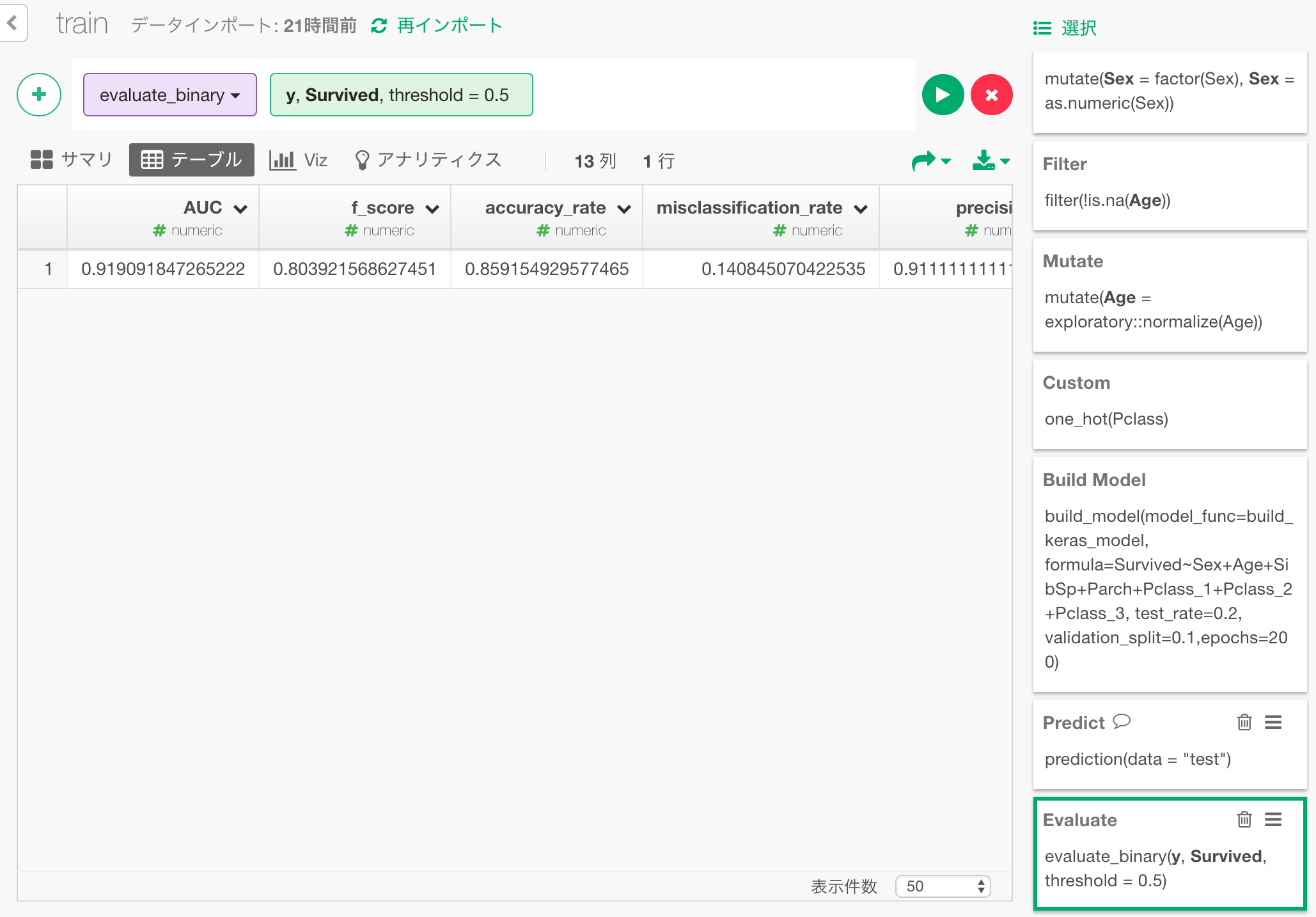
予測の精度を評価
この予測は、どの程度当たっているのでしょうか?予測結果を評価してみましょう。
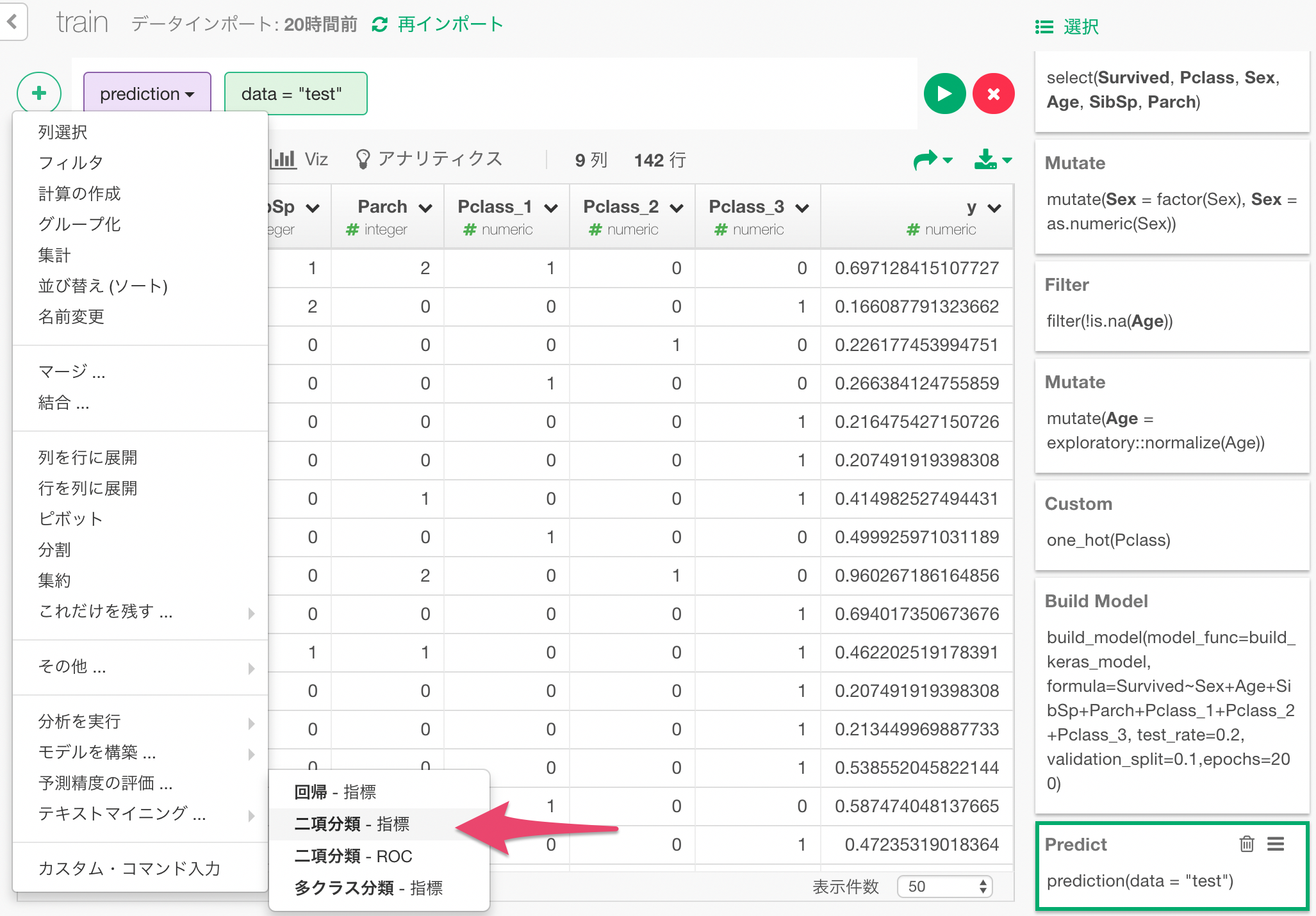
+メニューから、「二項分類 - 指標」を選びます。
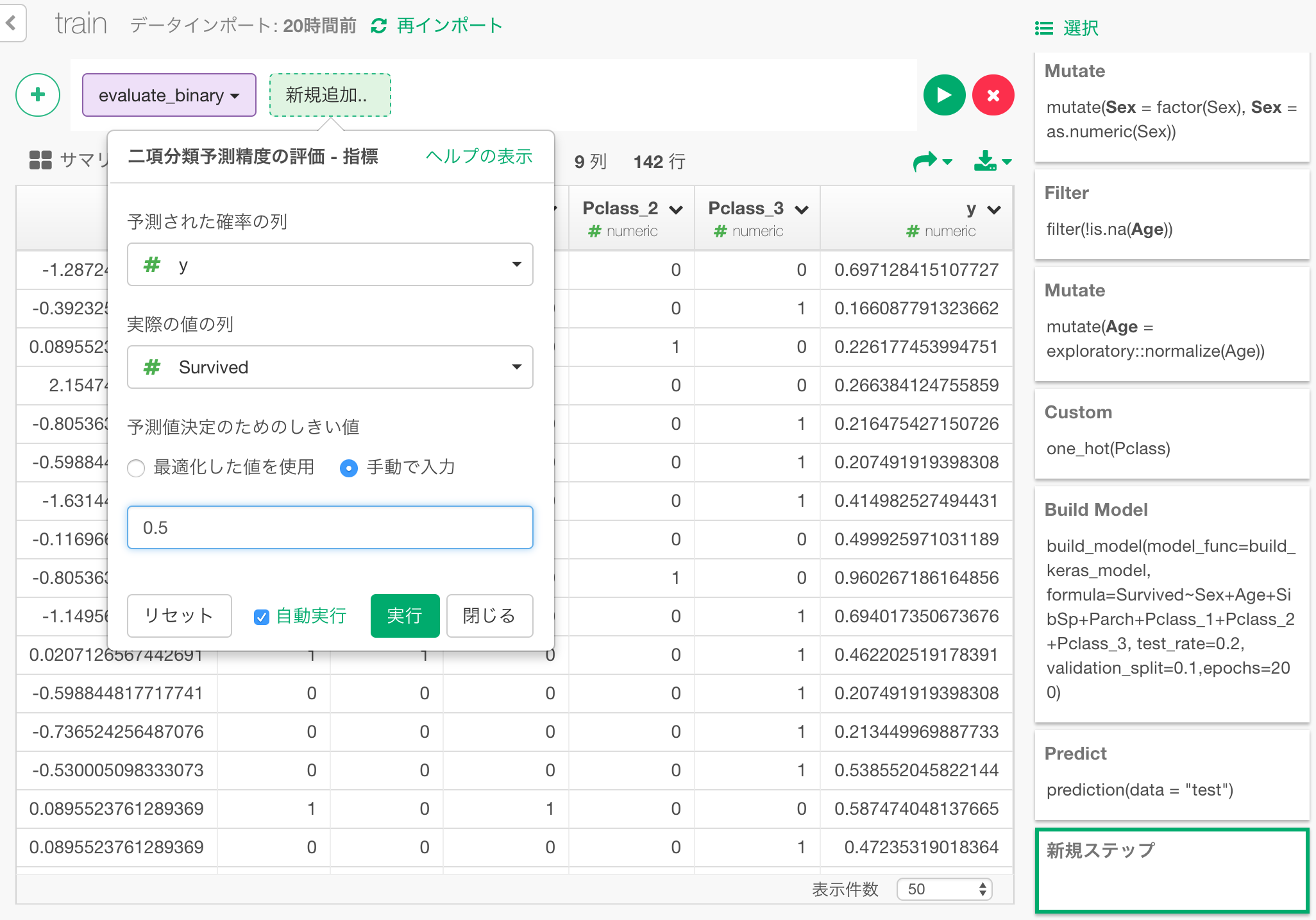
ダイアログで、次のように値を指定して、予測された生存確率yと実際の生存結果Surviveを比較して、予測を評価します。
すると、以下のような評価結果が出力されます。
もっとも分かりやすいのはaccuracy rateでしょう。テストデータの内の86%の場合が正解だったということを示しています。
また、他の指標として、予測された生存確率が、生存した人と生存できなかった人をどれだけクリアに分けているかをみる、AUCというものがあり、約92%となっています。
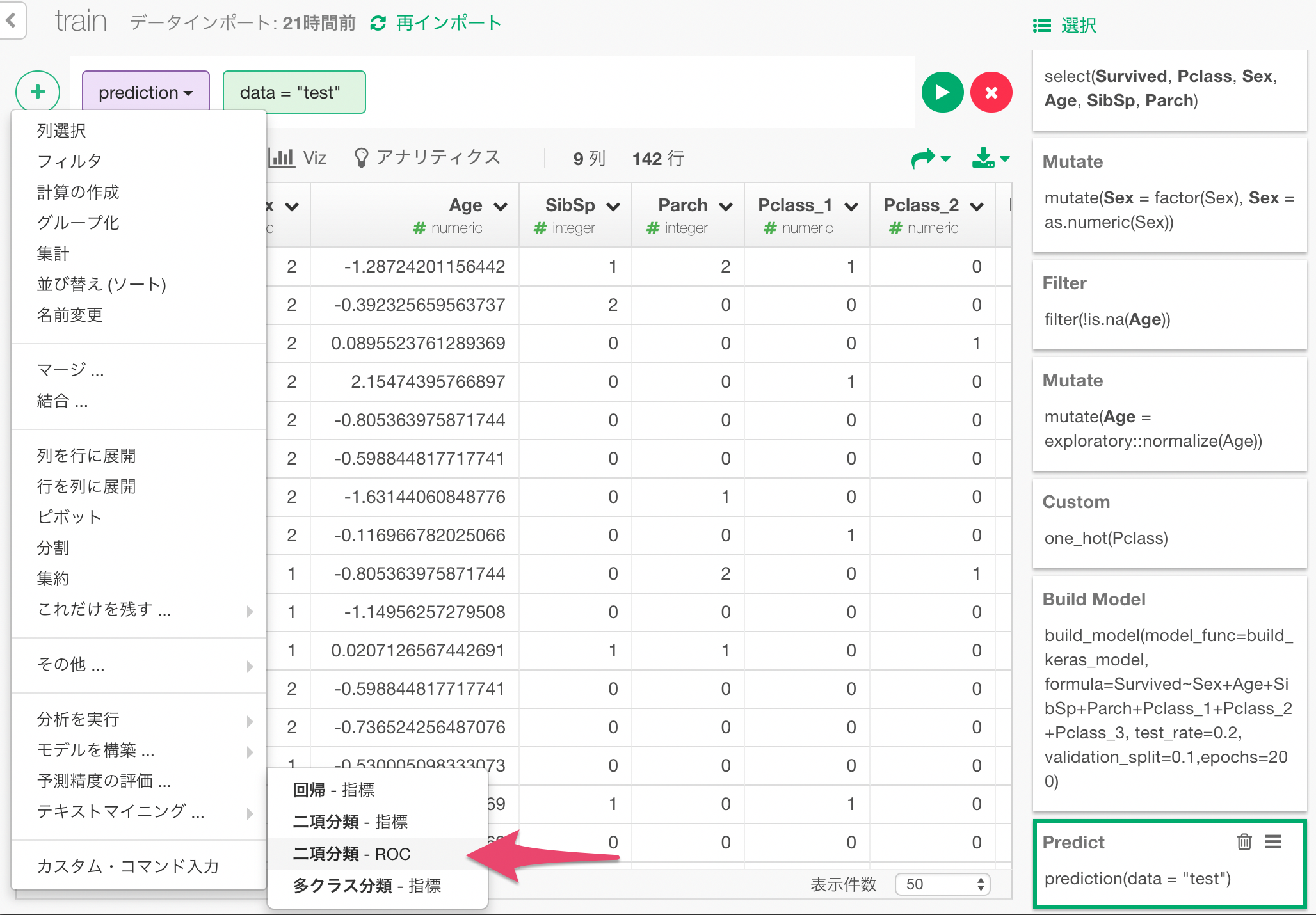
こちらを算出した根拠となるROCカーブを可視化してみましょう。
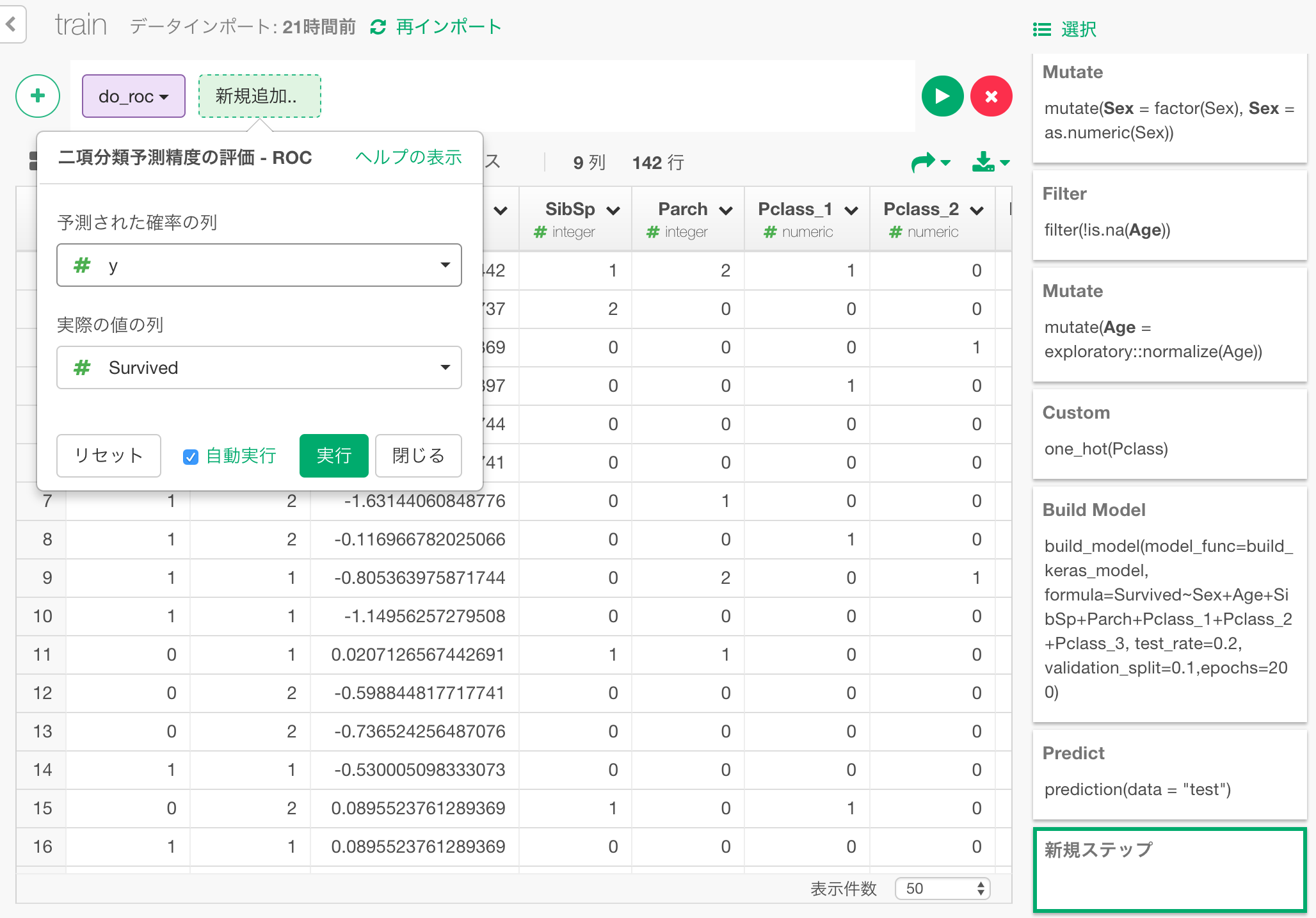
一つステップをもどして予測結果のステップに戻り、+メニューから、「二項分類 - ROC」を選びます。
ダイアログで、yとSurvivedを選択し、これらの比較によって、ROCカーブを計算するよう指定します。
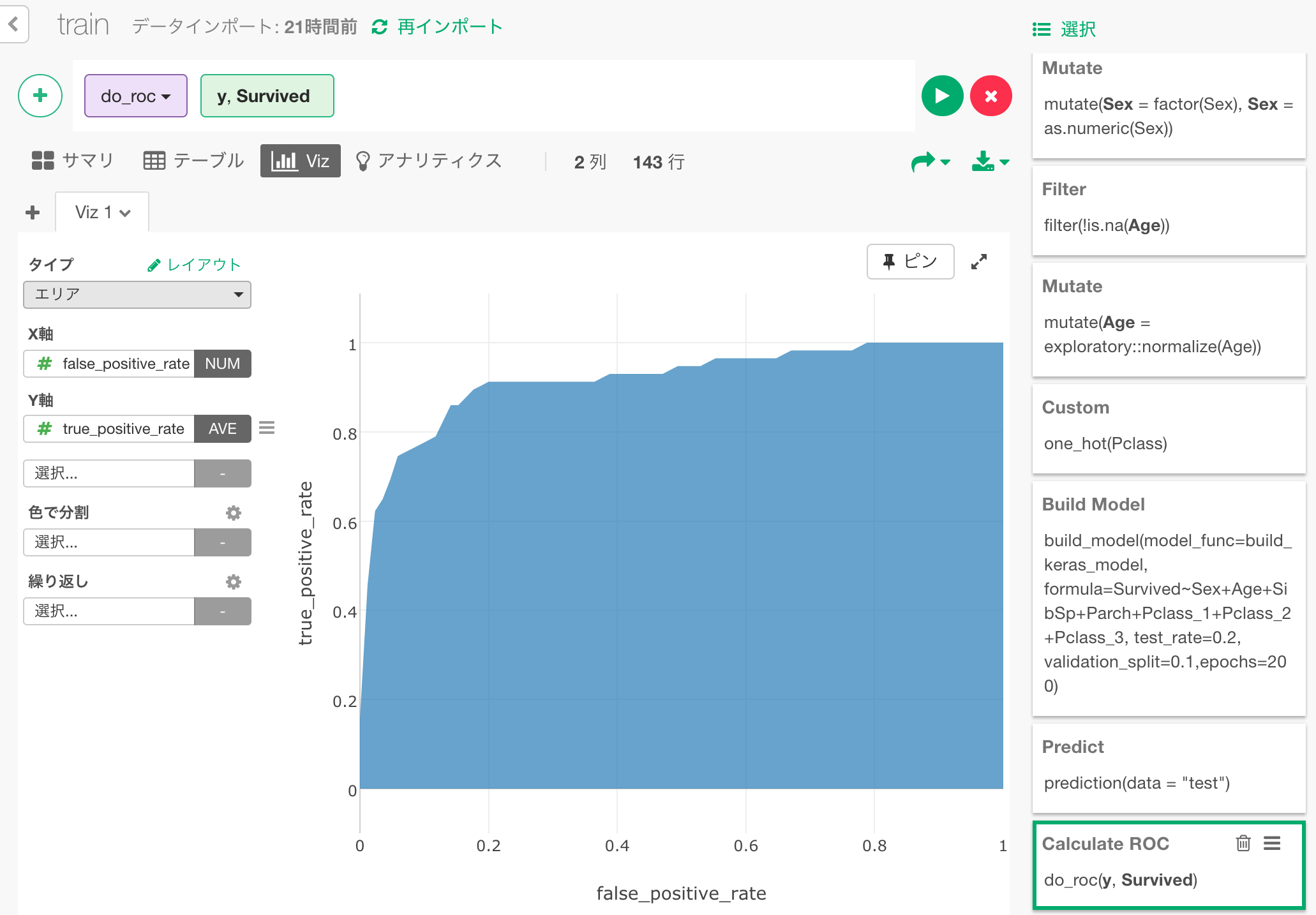
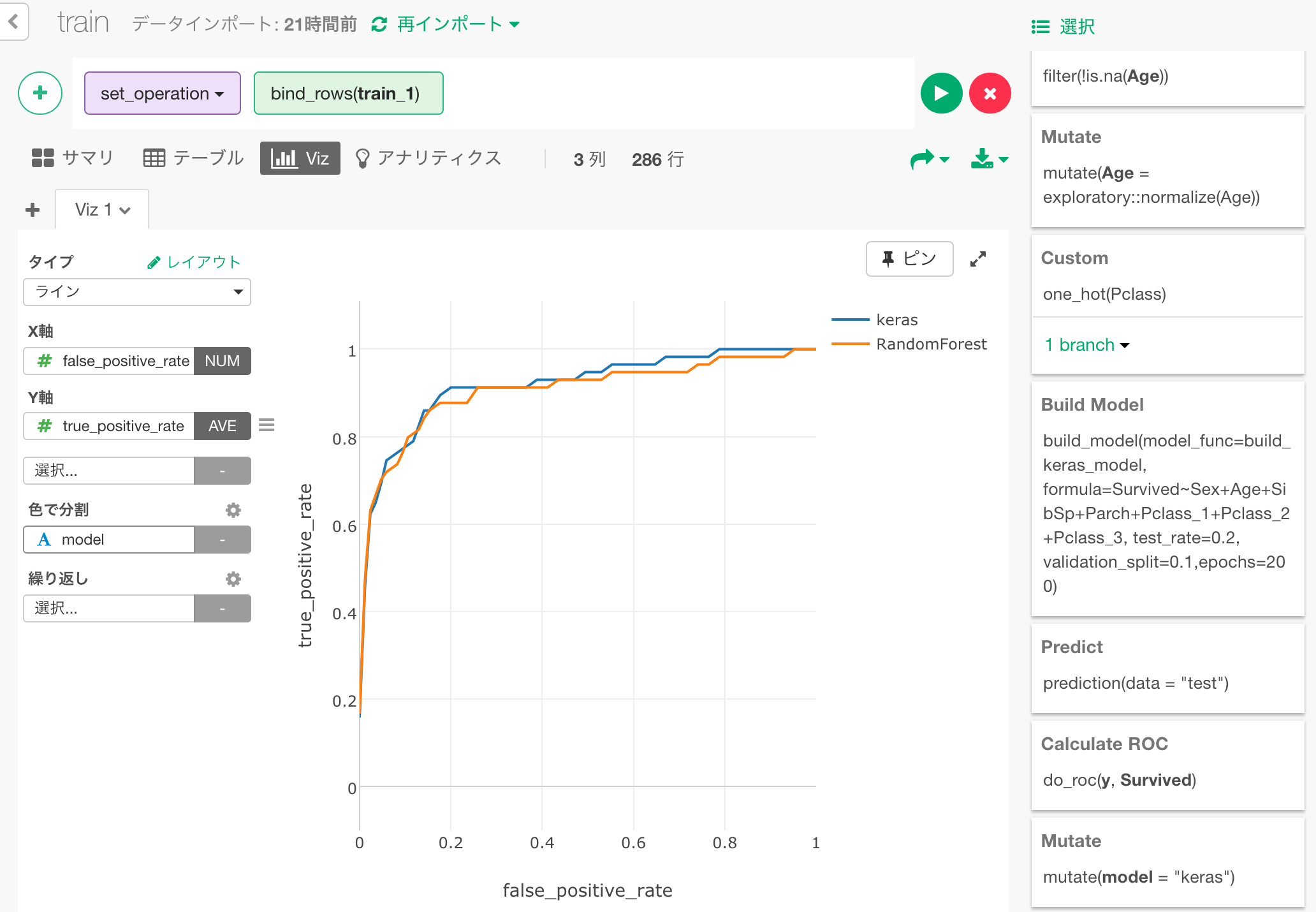
結果をエリアチャートで可視化するとこのようになります。この青い部分の面積が先ほど0.92という結果が出ていた、AUCとなります。
ちなみに、同じ予測をRandom ForestでしたときのROCカーブを一緒にプロットすると、このようになりました。
このモデルではRandom Forestの場合よりわずかに面積が多く、生存結果をクリアに分離できているということになります。(ただしこれは偶然に左右されるモデルの出来によるところもあり、私が何度か試してみたところでは、Random Forestに負けている場合も多々ありました。)
ニューラルネットワークの調整
ここで作ったニューラルネットワークをさらに調整して試してみたい方は、スクリプトの以下の部分を変更してみてください。変更後にSaveボタンを押すと、変更したfunctionがRに登録されます。
以下のように、3層のニューラルネットワークを構成するようになっていますが、kerasのおかげで、層やニューロンを増やしたり減らしたりする変更は以下のように直感的なコードになっています。
# 3層のニューラルネットワークモデルを作成
model <- keras_model_sequential()
model %>%
layer_dense(units = 7, activation = 'relu', input_shape = c(7)) %>% # 1層目に7個のニューロンを設置
layer_dropout(rate = 0.2) %>% # 2割のデータをdropoutする。
layer_dense(units = 5, activation = 'relu') %>% # 2層目に5個のニューロンを設置
layer_dropout(rate = 0.1) %>% # 1割のデータをdropoutする。
layer_dense(units = 1, activation = 'sigmoid') # 3層目で1つの生存確率にまとめ上げる。
まとめ
ExploratoryのUIをつかって、ディープラーニング用ライブラリkerasを使ったニューラルネットワークモデルでタイタニック号の乗客の生存を予測しました。
タイタニック号の乗客データは、900件程度であり、これは大量の学習データがあるときに真の力を発揮するディープラーニングに相応しい問題とは言えません。実際、kaggleでのdiscussionでも、このタイタニックの問題について主に取り上げられているのは、ディープラーニングではなくRandom Forestだったりするのですが、今回は理解のために手頃な問題としてとりあげました。
今後、もっと大量のデータを扱う問題にディープラーニングを適用する場合や、Random ForestやXGBoostといった、ディープラーニング以外のkaggleで人気のモデルについても書いていこうと思います。
データ分析をさらに学んでみたいという方へ
今年10月に、Exploratory社がシリコンバレーで行っている研修プログラムを日本向けにした、データサイエンス・ブートキャンプの第3回目が東京で行われます。本格的に上記のようなデータサイエンスの手法を、プログラミングなしで学んでみたい方、そういった手法を日々のビジネスに活かしてみたい方はぜひこの機会に、参加を検討してみてはいかがでしょうか。こちらに詳しい情報がありますのでぜひご覧ください。