DICOM画像とは、簡単にいうと、医療用の画像を装置間でやりとりするための通信プロトコル(DICOM)上で扱われる画像のことで、画像にいろんな付加情報(患者さんの情報であったり、装置の種類であったり、その画像そのものの情報であったり、いろいろなメタデータ)をつけたもののことである。
お医者さんで、CTやMRIなどの検査をした時に、病院によってはお願いすると画像をデータとしてくれることがある。この時のフォーマットが拡張子.dcmだったらDICOM画像だ。
PythonでDICOMを扱うための準備
PythonでDICOM画像を扱いたい場合は、pydicomを使うことになるようだ。
% anaconda show conda-forge/pydicom
Using Anaconda API: https://api.anaconda.org
Name: pydicom
Summary: Pure python package for DICOM medical file reading and writing
Access: public
Package Types: conda
Versions:
+ 0.9.9
To install this package with conda run:
conda install --channel https://conda.anaconda.org/conda-forge pydicom
というワケなので、conda install -c https://conda.anaconda.org/conda-forge pydicom する。
DICOM画像のサンプルを用意しておく
自分のMRI画像を残念ながら持っていない場合は、sample画像をダウンロードしておく。
DICOM Viewerとして有名なOsirixのサイトに、DICOM Image Libraryがあるので、ここから適当なヤツをもらっておく。
とりあえず、BRAINIXという脳腫瘍のMRI像をダウンロードしてみた。
この.zipファイルを展開すると、MRIで撮像後に、読影医が見るためにいろいろ画処理した画像(T1強調とか、そういうヤツ)があるのだけれど、この中で、「T1-3D-FFE-C\ -\ 801/」というフォルダに入っているファイル100枚を使うことにする。
DICOM画像を読む
以下に、pydicomの機能を使って画像を読み込んでみて、あれこれする例を示す。
DICOM画像を1枚読んでメタデータを参照する
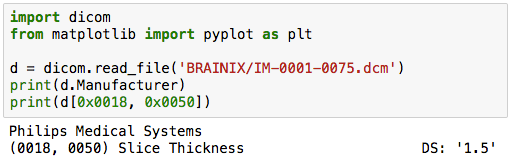
画像を読み込んで、DICOMヘッダを表示させる。
import dicom
d = dicom.read_file('BRAINIX/IM-0001-0075.dcm')
print(d)
ちなみに、特定のメタデータを表示させたい時は、次のようにする。
print(d.Manufacturer)
どうやら、装置メーカーは「Philips Medical Systems」らしい。
print(d[0x0018, 0x0050])
どうやら、Slice厚は1.5mmになっているようだ。
このように、DICOMのメタデータにはタグと呼ばれるキー名が決まっていて、キーには番号が振られている。そして、pydicomでは、キー名でも番号でも表示させることができる。
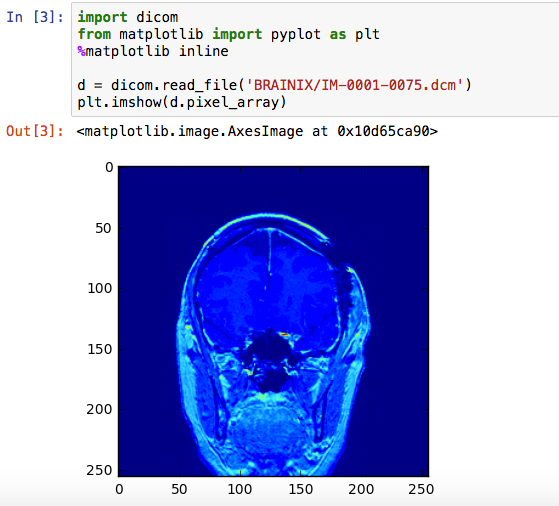
DICOM画像を1枚読んで画像を表示する
DICOM画像の画像部分の実体はnumpyのarray形式で取り出すことができるので、opencvでもmatplotlibでも使えば表示することができる。
複数のDICOM画像を読みこんで1枚の連続した断層画像に変換する
ところで、今回利用したBRAINIXの画像は1.5mm厚の連続した100枚の画像になっている。
この100枚の画像を読み込んで一つの3次元配列に取り込むことができる。
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
要するに、BRAINIXフォルダ以下の拡張子が.dcmになっているファイルを全て読み込んで、順にd_arrayという三次元配列に内容をコピーしている。
任意のスライスを表示したい場合は、例えば50枚目のスライスならば、 pyplt.imshow(d_array[:, :, 49])するだけだ。
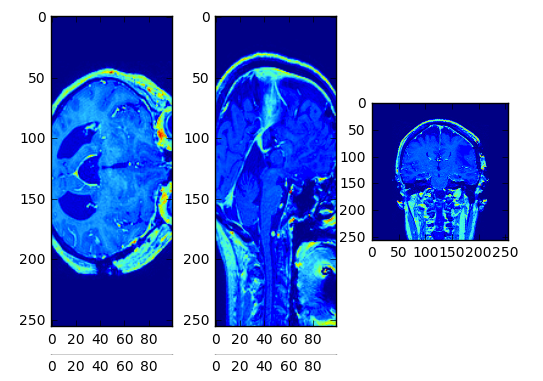
これで、3次元配列に読み込んだので、3断面表示も可能になる。
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
# BRAINIXフォルダ以下のdcmファイルを読み込む
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
d_array[:, :, dcms.index(dcm)] = d.pixel_array
# 3断面表示する
print(d_array.shape)
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])
DICOM画像を加工して保存する
読み込んだDICOM画像を加工してもとのDICOM画像に上書き保存する場合は、pixel_array の配列を編集することになる。
ただし、pixel_arrayの配列を編集するだけではもとの画像は変わらない。これは、DICOM画像のイメージデータの実体はPixelDataという配列に保存されており、その読み出し用のインターフェイスがpixel_arrayになっているという事情による。そこで、編集したpixel_arrayの内容が例えばimgというアレイに保存されているのであれば、ソレをPixelDataに書き戻す処理が必要になる。
d = dicom.read_file(dcm)
img = d.pixel_array
# imgの加工
d.PixelData = img.tostring()
d.save_as(dcm)
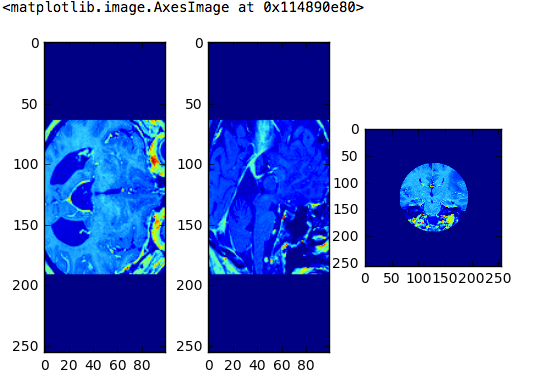
DICOM画像を円形に切り抜いてみる
「DICOM画像を中央部分のみ、画像の縦横の長さの半分の直径の円で切り抜く」という作業を、何のためか分からないけれど、やってみることにする。
以下のコードで、画像の縦横の半分の直径の円の内側が1で、外側が0の画像ができる。
半径rとすると、画像の縦横サイズはref_dicom.pixel_array.shapeから取得できるので、そのサイズの1/4を設定する。
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
このmaskを掛け算すると、円形の内側が1で、外側が0なので、円形に切り抜くことができる。
### 複数のDICOM画像ファイルを読み込んで、中央部を円形に切り抜く
import os
import dicom
from matplotlib import pyplot as plt
%matplotlib inline
# BRAINIXフォルダ以下のdcmファイルを読み込む
root_dir = './BRAINIX'
dcms = []
for d, s, fl in os.walk(root_dir):
for fn in fl:
if ".dcm" in fn.lower():
dcms.append(os.path.join(d, fn))
ref_dicom = dicom.read_file(dcms[0])
# 円に切り抜くためのマスクを作成する
r = ref_dicom.pixel_array.shape[0] / 4
x, y = np.indices((ref_dicom.Rows, ref_dicom.Columns))
circle = (x - (ref_dicom.Columns / 2))**2 + (y - (ref_dicom.Rows / 2))**2 < r**2
mask = circle.astype(int)
# 全ての画像にmask処理を実施する
d_array = np.zeros((ref_dicom.Rows, ref_dicom.Columns, len(dcms)), dtype=ref_dicom.pixel_array.dtype)
for dcm in dcms:
d = dicom.read_file(dcm)
img = d.pixel_array * mask
d_array[:, :, dcms.index(dcm)] = img
# 3断面表示する
plt.figure(figsize=(8, 6))
plt.subplot(1, 3, 1)
plt.imshow(d_array[127, :, :])
plt.subplot(1, 3, 2)
plt.imshow(d_array[:, 127, :])
plt.subplot(1, 3, 3)
plt.imshow(d_array[:, :, 49])
PythonでDICOM画像を扱う時は、だいたいこんな感じ。