2016.09.14 処理時間のバラつきについて追記しました
scikit-learnのSVC(rbfカーネルとlinearカーネル)とLinearSVCの処理速度を比較してみました.
利用したデータはRのkernlabパッケージに含まれているspamデータです.
説明変数は4601サンプル,57次元,
ラベルはspam:1813サンプル,nonspam:2788サンプルです.
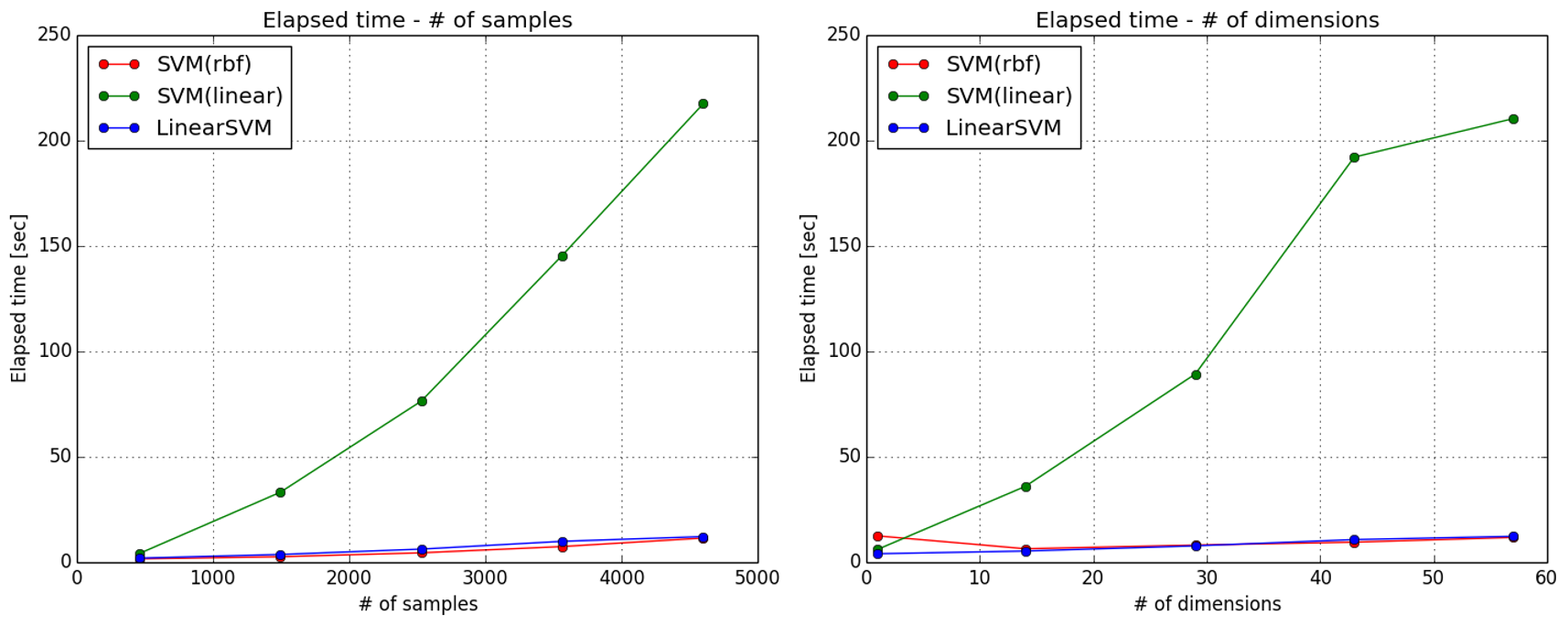
サンプル数,次元数を変えた時の結果は以下の通りです.
SVCのlinearカーネルが遅すぎますね.
ついついカーネル種別まで含めてグリッドサーチしてしまいたくなりますが,
きちんとLinearSVCを使ったほうが良さそうです.
検証用コードは以下.
処理時間計測の都合でパラメータCを振っています.
また特徴量選択(次元削減)はRandomForestのfeature importanceを利用しました.
これは適当に選択したところ,逆に処理時間が長くなったためです.
# -*- coding: utf-8 -*-
import time
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
from sklearn.svm import SVC
from sklearn.svm import LinearSVC
from sklearn import cross_validation
from sklearn.grid_search import GridSearchCV
from sklearn.preprocessing import StandardScaler
from sklearn.utils import shuffle
from sklearn.ensemble import RandomForestClassifier
from scipy.stats.mstats import mquantiles
def grid_search(X, y, estimator, params, cv, n_jobs=3):
mdl = GridSearchCV(estimator, params, cv=cv, n_jobs=n_jobs)
t1 = time.clock()
mdl.fit(X, y)
t2 = time.clock()
return t2 - t1
if __name__=="__main__":
data = pd.read_csv('spam.txt', header=0)
y = data['type']
del data['type']
data, y = shuffle(data, y, random_state=0)
data = StandardScaler().fit_transform(data)
clf = RandomForestClassifier(n_estimators=100)
clf.fit(data, y)
ndim, elp_rbf, elp_lnr, elp_lsvm = [], [], [], []
for thr in mquantiles(clf.feature_importances_, prob=np.linspace(1., 0., 5)):
print thr,
X = data[:,clf.feature_importances_ >= thr]
ndim.append(X.shape[1])
cv = cross_validation.StratifiedShuffleSplit(y, test_size=0.2, random_state=0)
print 'rbf',
elp_rbf.append(grid_search(X, y, SVC(random_state=0),
[{'kernel': ['rbf'], 'C': [1, 10, 100]}], cv))
print 'linear',
elp_lnr.append(grid_search(X, y, SVC(random_state=0),
[{'kernel': ['linear'], 'C': [1, 10, 100]}], cv))
print 'lsvm'
elp_lsvm.append(grid_search(X, y, LinearSVC(random_state=0),
[{'C': [1, 10, 100]}], cv))
plt.figure()
plt.title('Elapsed time - # of dimensions')
plt.ylabel('Elapsed time [sec]')
plt.xlabel('# of dimensions')
plt.grid()
plt.plot(ndim, elp_rbf, 'o-', color='r',
label='SVM(rbf)')
plt.plot(ndim, elp_lnr, 'o-', color='g',
label='SVM(linear)')
plt.plot(ndim, elp_lsvm, 'o-', color='b',
label='LinearSVM')
plt.legend(loc='best')
plt.savefig('dimensions.png', bbox_inches='tight')
plt.close()
nrow, elp_rbf, elp_lnr, elp_lsvm = [], [], [], []
for r in np.linspace(0.1, 1., 5):
print r,
X = data[:(r*data.shape[0]),:]
yy = y[:(r*data.shape[0])]
nrow.append(X.shape[0])
cv = cross_validation.StratifiedShuffleSplit(yy, test_size=0.2, random_state=0)
print 'rbf',
elp_rbf.append(grid_search(X, yy, SVC(random_state=0),
[{'kernel': ['rbf'], 'C': [1, 10, 100]}], cv))
print 'linear',
elp_lnr.append(grid_search(X, yy, SVC(random_state=0),
[{'kernel': ['linear'], 'C': [1, 10, 100]}], cv))
print 'lsvm'
elp_lsvm.append(grid_search(X, yy, LinearSVC(random_state=0),
[{'C': [1, 10, 100]}], cv))
plt.figure()
plt.title('Elapsed time - # of samples')
plt.ylabel('Elapsed time [sec]')
plt.xlabel('# of samples')
plt.grid()
plt.plot(nrow, elp_rbf, 'o-', color='r',
label='SVM(rbf)')
plt.plot(nrow, elp_lnr, 'o-', color='g',
label='SVM(linear)')
plt.plot(nrow, elp_lsvm, 'o-', color='b',
label='LinearSVM')
plt.legend(loc='best')
plt.savefig('samples.png', bbox_inches='tight')
plt.close()
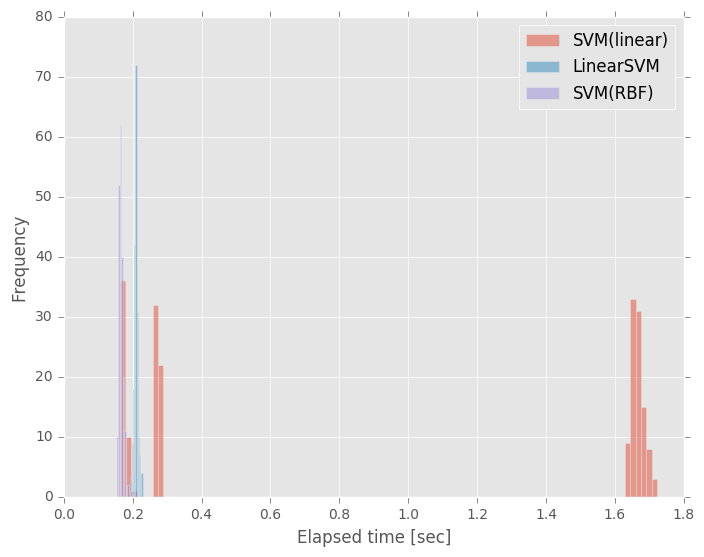
追記
SVM(linear)の処理時間についてコメントをいただいたので調べてみました.
Python2.7.12,scikit-learn0.17.1で,
データ数1000,特徴量数29,200回試行したときの処理時間のバラつきは下図のようになりました.
SVM(linear),怪しいですね…